修回日期: 2010-05-03
接受日期: 2010-05-18
在线出版日期: 2010-06-18
目的: 探讨新疆主要民族之间食管癌患者血清蛋白质指纹图谱的差异.
方法: 用表面增强激光解吸电离飞行时间质谱技术(SELDI-TOF-MS)检测43例汉族、43例维吾尔族及41例哈萨克族食管癌血清蛋白质指纹图谱, 所得结果用ZUCI-PDAS软件分析处理, 筛选血清差异性蛋白质.
结果: 同民族间的血清蛋白质指纹图谱存在明显差异. 汉族组与维吾尔族组中质荷比为4 310.0109, 8 713.0142, 7 993.0223的3个蛋白质峰差异具有统计学意义(P<0.05); 汉族组与哈萨克族食管癌组中质荷比为4 310.0184, 8 167.9277, 8 158.1117, 13 789.4864, 8 067.7056, 4 611.9098, 7 993.4422, 16 146.8706的8个蛋白质峰差异具有统计学意义(P<0.05); 维吾尔族组与哈萨克族组中质荷比分别为9 161.7944, 4 611.6342, 6 649.6163, 4 979.3807的4个蛋白质峰差异具有统计学意义(P<0.05); 质荷比4 310.0109的蛋白峰在汉族组中呈低表达, 而在维吾尔族组和哈萨克族组中均呈高表达. 在维吾尔族组和哈萨克族组之间该蛋白峰未见到有差异.
结论: 汉族、维吾尔族及哈萨克族食管癌患者之间血清蛋白质指纹图谱的差异具有统计学意义, 在新疆地区建立不同民族食管癌患者血清蛋白质指纹图谱疾病诊断模型有一定的临床意义.
引文著录: 张昌明, 张建龙, 张琼, 张铸, 张海平, 孙清超, 丁旭, 刘钰莉, 伊力亚尔·夏合丁. 新疆汉族、维吾尔族及哈萨克族食管癌血清蛋白质指纹图谱. 世界华人消化杂志 2010; 18(17): 1773-1779
Revised: May 3, 2010
Accepted: May 18, 2010
Published online: June 18, 2010
AIM: To conduct a serum protein profile analysis in Han, Uygur and Kazakh patients with esophageal carcinoma (EC) in Xinjiang, China.
METHODS: Serum samples from patients with EC (43 Han, 43 Uygur and 41 Kazakh subjects) were detected by weak cation exchange (CM10) protein chip assay using surface-enhanced laser desorption/ionization time-of-flight mass spectrometry (SELDI-TOF-MS) technology to screen differentially expressed serum markers for EC.
RESULTS: The peaks at the mass to charge ratios (M/Z) 4 310.0109, 8 713.0142 and 7 993.0223 were significantly different between Han and Uygur EC patients (P < 0.05). The peaks at M/Z 4 310.0184, 8 167.9277, 8 158.1117, 13 789.4864, 8 067.7056, 4 611.9098, 7 993.4422 and 16 146.8706 were significantly different between Han and Kazakh EC patients (P < 0.05). The peaks at M/Z 9 161.7944, 4 611.6342, 6 649.6163 and 4 979.3807 were significantly different between Uygur and Kazakh EC patients (P < 0.05). The peat at M/Z 4 310.0109 was highly expressed in Uygur and Kazakh patients but lowly expressed in Han patients.
CONCLUSION: The protein fingerprints are significantly different among Han, Uygur and Kazakh EC patients in Xinjiang, China, which can be used to build a diagnostic model of EC.
- Citation: Zhang CM, Zhang JL, Zhang Q, Zhang Z, Zhang HP, Sun QC, Ding X, Liu YL, Sheyhidin I. Identification of esophageal carcinoma-associated proteins by proteomics in Han, Uygur and Kazakh patients with esophageal carcinoma in Xinjiang, China. Shijie Huaren Xiaohua Zazhi 2010; 18(17): 1773-1779
- URL: https://www.wjgnet.com/1009-3079/full/v18/i17/1773.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i17.1773
食管癌是世界上最常见的恶性肿瘤之一[1]. 居世界恶性肿瘤死亡的第6位[2], 每年约30万人死于食管癌疾病. 其流行病特征除了显著的地域分布差异之外, 民族分布差异亦是其主要的流行病学特征之一[3]. 食管癌发病率地区和种族差异很大. 新疆是多民族的聚集区, 主要是哈萨克、维吾尔族、汉族和回族四个民族, 哈萨克族食管癌粗死亡率为89.82/10万, 其调整死亡率达68.88/10万[4], 明显高于其他三个民族(汉族11.36/10万、维吾尔族13.01/10万、回族25.6/10万).
随着蛋白质组学相关技术的迅速发展, 临床血清蛋白质组学成为当今生命科学领域中极其活跃的学科[5]. 临床血清蛋白质组学从新的角度研究肿瘤, 寻找肿瘤标志物, 为肿瘤的早期诊断、预后和动态监测提供了重要依据. 目前, 蛋白质组学研究包括双相凝胶电泳为主的蛋白质分离技术和以质谱技术、生物信息学为主的蛋白质鉴定技术[6]. 主要核心技术是蛋白质芯片[7]. 双向凝胶电泳技术(two-dimensional gel elec-trophoresis, 2-DE)是蛋白质组学研究中普遍应用的技术平台[8-10]. 其优点是能够同时分离大量蛋白质, 并通过蛋白质点染色的强度对其进行定量分析, 也可提供蛋白质翻译后修饰信息. 但是2-DE技术也存在着一些不足之处, 如检测样本量少、重复性差、染色转移等环节操作困难等缺点[11]. 这些使其在蛋白质组学研究中的应用受到一定的制约. 表面增强激光解吸电离飞行时间质谱技术(surface enhanced laser desorption/ ionization time of flight mass spectrometry, SELDI-TOF-MS)是一种新的蛋白质组学研究手段, 由Yip等[12]将其应用于临床检测, 建立了多种疾病蛋白质指纹图谱诊断模型, SELDI-TOF-MS是发现肿瘤标志物最有希望的工具之一[13,14], 被广泛应用于体液和组织蛋白的分析. 食管癌是世界上最常见的恶性肿瘤之一, 其发病率地区和种族差异很大. 新疆是食管癌的高发区, 同时又是多民族聚集区. 不同民族之间食管癌的血清蛋白质指纹图谱有无差异尚未见文献报道. 我们采用该项技术对新疆地区主要民族(汉族、维吾尔族、哈萨克族)食管癌患者的血清蛋白质指纹图谱进行对比研究, 筛选血清特异性蛋白质.
选取2008-01/2008-08我院胸外科住院经手术病理诊断确诊为食管癌患者127例, 其中汉族43例, 男30例, 女13例, 年龄45-78(63.32±7.31)岁; 维吾尔族43例, 男30例, 女13例, 年龄40-69(55.07±7.80)岁; 哈萨克族41例, 男30例, 女11例, 年龄31-82(57.08±8.90)岁. 所有食管癌患者于术前抽取清晨空腹前臂静脉全血5 mL. 均已获患者知情同意. Protein Chip Biology System(PBS II-C)质谱仪、弱阳离子交换蛋白芯片(CM10蛋白芯片, Cip hergen Biosystems公司, US). 尿素(Urea)、乙腈(CAN)、3-[3-(胆酰胺基丙酸)二甲氨基]丙磺酸盐(CHAPS)、50%乙腈和0.5%三氟乙酸饱和溶液(SPA). 三羟甲基氨基甲烷盐酸盐(Tris-Hcl)、二硫苏糖醇(DDT)等均购自美国Sigma公司. 96孔细胞培养板购自美国BD公司.
1.2.1 样品收集: 外周血采集后立即放入4℃冰箱静置20 min, 3 000 r/min, 于4 ℃离心5 min. 取上清加eppendof管, 3 000 r/min, 4 ℃再次离心5 min, 0.5 mL eppendof管分装(在干冰上分装), 100 μL每管, 放入-80 ℃冰箱保存. 每个样本至少三管以上. 溶血样本弃用.
1.2.2 样品的准备: 使用前自-80 ℃冰箱内取出样本, 冰上溶解30 min左右, 10 000 r/min, 在4 ℃离心2 min. 除去血清中的不溶物. 取5 μL血清加10 μL U9缓冲液(9 mol/L Urea, 2% CHAPS, 1% DTT)稀释混匀, 振荡器600 r/min 4 ℃振荡30 min. 然后向15 μL稀释样品中加185 μL醋酸钠缓冲液(50 mmol/L NaAC, pH4.0), 振荡器上混匀.
1.2.3 芯片预处理: (1)将CM10芯片安装于加样器(bioprocessor)内; (2)平衡芯片: 取醋酸钠缓冲液(50 mmol/L NaAC, pH4.0)200 μL/孔加入芯片的加样孔中, 平衡芯片2次, 每次5 min; (3)芯片上样: 取稀释后的血清样品100 μL/孔加入平衡好的芯片中的加样孔, 置芯片于振荡器振荡(4 ℃ 1 h); (4)样品洗脱: 取醋酸钠缓冲液(50 mmol/L NaAC, pH4.0)200 μL/孔加入芯片加样孔, 洗脱3次, 每次5 min. 取200 μL/孔的HPLC水冲洗芯片2次; (5)蛋白质获能: 卸下加样器, 待芯片表面自然晾干后, 取能量吸收剂SPA(含饱和芥子酸溶于50%乙腈和0.5%三氟乙酸)1 μL/孔加入芯片加样孔, 待自然挥发后再重复加SPA 1 μL/孔.
1.2.4 芯片检测: 将芯片置入蛋白芯片阅读机中, 用加有All-in-one标准蛋白质的NP20芯片校正仪器至0.1%范围内. 芯片阅读仪参数设置如下: 激光强度190, 检测灵敏度8, 收集数据的最高相对分子质量50 000 Da, 优化相对分子质量范围2 000-20 000 Da, 信号收集位置从20-80, 每个样本取144个点. Ciphergen Protein Chip 3.0软件收集数据并输出. 其中纵坐标为蛋白质相对含量, 横坐标为蛋白质质荷比.
统计学处理 采用浙江大学肿瘤研究所蛋白质质谱数据分析软件包(ZUCI- Protein Chip Data Analyze System软件包)分析所得到的数据. 即先将原始数据用离散小波分析去除噪声, 并减掉基线. 将各个样本种质荷比的差异小于0.30%的峰聚为一类. 用支持向量机方法建立蛋白质指纹图诊断模型, 用留一法交叉验证作为评估模型判别效果的方法. 特征向量的选取采用统计过滤结合模型依赖性筛选的方法. 对每个荷质比峰作Wilcoxon秩和检验, 检验水准α = 0.05, 选出P值最小的几个峰进一步分析. 将几个峰的任意组合用于支持向量机模型的输入, 用留一法评估模型的预测效果, 选出建立支持向量机模型预测的约登指数最高的组合作为最终的候选标志物. 用Ciphergen Protein Chip 3.2.1软件初步筛选蛋白质峰. 获得血清蛋白质指纹图谱后, 用ZUCI-PDAS进行数据分析及统计学处理. 两组之间的蛋白质峰的比较采用Wilcoxon秩和检验, P<0.05有统计学意义.
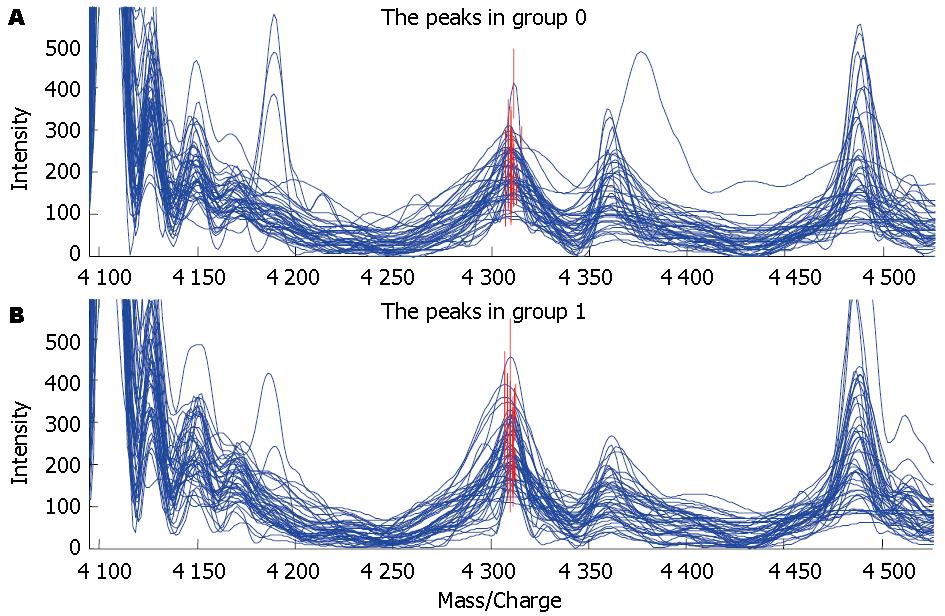
比较43例汉族组与43例维吾尔族组食管癌血清蛋白质指纹图谱数据, 判别分析筛选出质荷比(M/Z)为4 310.0109, 8 713.0142, 7 993.0223的3个蛋白质峰差异具有统计学意义(P<0.05, 表1). 图1显示了在汉族组呈低表达而在维吾尔族组呈高表达的一个蛋白峰(4 310.0109)的质谱图. 用4 310.0109, 8 713.0142, 7 993.0223, 15 963.5803, 6 897.7447, 4 148.6997, 9 162.9468, 8 164.5504, 6 649.2984, 6 909.0273这10个蛋白质峰建立了汉族、维吾尔族食管癌血清蛋白指纹图谱验证模型. 用该分类树模型作为生物标志物来检测, 在学习模式下分组正确率均是100%(43/43), 在测试模式下分组正确率65.1%(28/43)和67.4%(29/43).
| 蛋白质峰(M/Z) | 平均峰强度 | P值 | |
| 汉族组 | 维吾尔族组 | ||
| 4 310.0109 | 201.8093 | 239.4164 | 0.023115 |
| 8 713.0142 | 1 605.8303 | 1 264.4156 | 0.032196 |
| 7 993.0223 | 383.6436 | 446.9580 | 0.044176 |
| 15 963.5803 | 261.4016 | 316.8008 | 0.053029 |
| 6 897.7447 | 277.0574 | 223.8065 | 0.067094 |
| 4 148.6997 | 218.9902 | 248.1421 | 0.071054 |
| 9 162.9468 | 125.3912 | 164.8305 | 0.078078 |
| 8 164.5504 | 131.2737 | 293.6366 | 0.081042 |
| 6 649.2984 | 2 334.8658 | 1 845.1582 | 0.088851 |
| 6 909.0273 | 218.5518 | 185.4641 | 0.090484 |
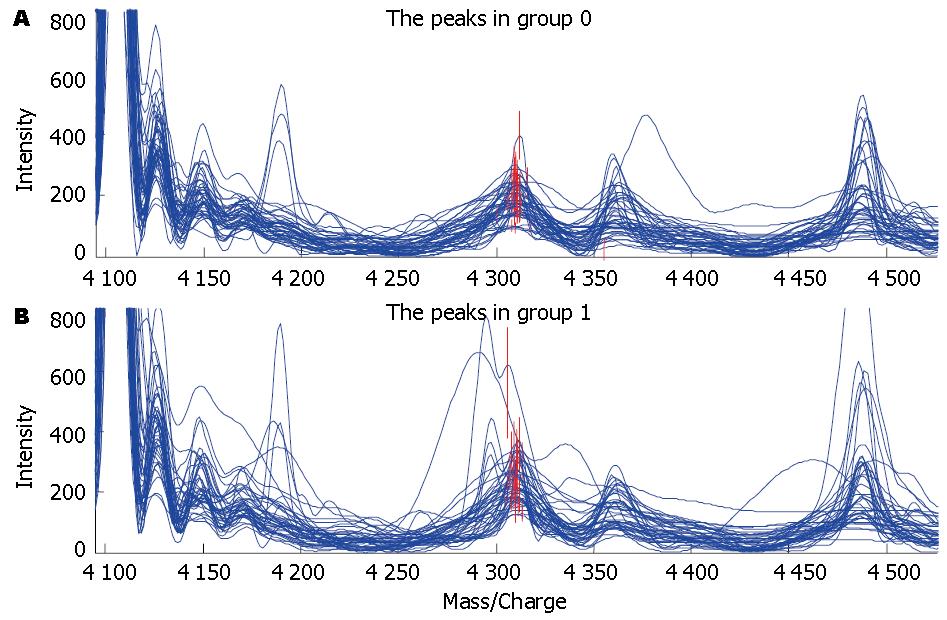
比较43例汉族组与41例哈萨克族食管癌组血清蛋白质指纹图谱数据, 经判别分析筛选出质荷比分别为4 310.0184, 8 167.9277, 8 158.1117, 13 789.4864, 8 067.7056, 4 611.9098, 7 993.4422, 16 146.8706的8个蛋白质峰差异具有统计学意义(P<0.05, 表2). 图2显示了在汉族组呈低表达而在哈萨克族组呈高表达的一个蛋白峰(4 310.0184)的质谱图.
| 蛋白质峰(M/Z) | 平均峰强度 | P值 | |
| 汉族组 | 维吾尔族组 | ||
| 4 310.0184 | 195.9261 | 251.8594 | 0.0040724 |
| 8 167.9277 | 125.4354 | 197.7775 | 0.0078668 |
| 8 158.1117 | 124.1499 | 205.9943 | 0.0131840 |
| 13 789.4864 | 254.0904 | 169.7487 | 0.0149320 |
| 8 067.7056 | 118.2393 | 187.2620 | 0.0204660 |
| 4 611.9098 | 329.1491 | 183.4712 | 0.0219730 |
| 7 993.4422 | 366.0990 | 620.9567 | 0.0362620 |
| 16 146.8706 | 88.2441 | 164.4688 | 0.0469680 |
| 6 898.7666 | 268.4546 | 215.9549 | 0.0706660 |
| 9 213.4132 | 1 849.3786 | 1 023.8855 | 0.0764240 |
用4 310.0184, 13 789.4864, 8 067.7056这3个蛋白质峰建立了汉族、哈萨克族食管癌血清蛋白质指纹图谱验证模型. 用该分类树模型作为生物标志物来检测, 在学习模式下分组正确率分别为86.0%(37/43)和87.8%(36/41), 在测试模式下分组正确率79.1%(34/43), 73.2%(30/41).
比较43例维吾尔族组与41例哈萨克族组蛋白质指纹图谱数据, 经判别分析筛选出质荷比分别为9 161.7944, 4 611.6342, 6 649.6163, 4 979.3807的4个蛋白质峰差异具有统计学意义(P<0.05), 结果见表3. 用4 611.6342, 4 979.3807, 5 672.3799这3个蛋白质峰建立了维吾尔族、哈萨克族食管癌血清蛋白指纹图谱验证模型. 用该分类树模型作为生物标志物来检测, 在学习模式下分组正确率分别为81.4%(35/43)和65.9%(27/41), 在测试模式下分组正确率67.4%(29/43), 48.8%(20/41).
| 蛋白质峰(M/Z) | 平均峰强度 | P值 | |
| 汉族组 | 维吾尔族组 | ||
| 9 161.7944 | 162.8568 | 112.8158 | 0.0066995 |
| 4 611.6342 | 415.6786 | 190.2466 | 0.0168820 |
| 6 649.6163 | 1 843.3545 | 2 307.8194 | 0.0459840 |
| 4 979.3807 | 102.1327 | 275.7468 | 0.0469680 |
| 9 213.5520 | 2 056.7564 | 1 059.4678 | 0.0532490 |
| 9 443.0115 | 385.6755 | 300.8297 | 0.0566440 |
| 6 451.5978 | 682.3664 | 859.0737 | 0.0942510 |
| 9 425.7070 | 401.0780 | 262.9021 | 0.1111900 |
| 9 384.7967 | 261.7463 | 197.4603 | 0.1237700 |
| 5 672.3799 | 351.4679 | 399.3350 | 0.1522100 |
食管癌是一种常见的消化系肿瘤, 目前食管癌患者的预后极差, 临床就诊的患者大多数属中晚期, 其主要原因是: 食管癌病因不清, 发病机制不明确, 缺乏敏感有效的早期生物学诊断指标和方法[15]. 全世界每年约有30万人死于食管癌. 我国是世界上食管癌高发地区之一, 每年平均病死约15万人, 占各部位癌死亡的第2位, 仅次于胃癌. 流行病学研究发现, 食管癌的发病地区差异很大, 高发区人群食管癌的发病率和死亡率与低发区相差50倍. 食管癌在种族方面也有很大的差异. 南非和北美的黑人中, 尤其是年龄在55岁以前, 食管鳞状细胞癌发病的危险明显高于同地区的白种人. 这种情况在该地区其他肿瘤中并没有发现. 新疆哈萨克民族是食管癌的高发民族, 其中新疆托里县哈萨克民族EC的发病率是155.9/10万, 而同地区的其他民族EC癌的发病率仅是22.3/10万. 中国部分少数民族食管癌死亡情况统计显示, 新疆哈萨克族男女合计死亡率是33.90/10万人口, 是其他民族的2-31倍; 维吾尔族是男女合计死亡率是10.60/10万人口.
SELDI-TOF-MS是近几年新兴的差异蛋白质组学技术, 整合了芯片和质谱的优点, 方便, 快速, 灵敏度高, 蛋白信息量多[16], 可直接对原始生物样品(血清[17]、尿液[18]、脑脊液[19,20]、胸腔积液[21]、乳头分泌物[22]、细胞裂解液[23,24]等)进行分析. 不仅可发现一种蛋白质或生物标志分子, 而且还可以发现不同的多种方式的组合蛋白质谱, 目前被广泛用于肿瘤标志物的筛选[25-27].
在以血清蛋白质指纹图谱技术建立疾病诊断模型时必须考虑到人种之间的差异[28]. He等以中国汉族人群为研究对象, 采用霰弹策略(shortgun strategy)建立了蛋白质指纹图谱, 并鉴定了944种蛋白质[29]. 王庆荣等[30]应用SELDI-TOF-MS对新疆302例血清样本进行族别、性别之间的血清蛋白质指纹图谱的比较. 结果显示, 不同民族间的血清蛋白质指纹图谱存在明显差异, 汉族血清中4个差异蛋白质的丰度均低于维吾尔族和哈萨克族.
本研究应用SELDI-TOF-MS对新疆43例汉族、43例维吾尔族及41例哈萨克族食管癌血清标本的蛋白质指纹图谱进行比较. 结果显示, 不同民族间的血清蛋白质指纹图谱之间的差异具有统计学意义. 汉族组与维吾尔族组中质荷比为4 310.0109, 8 713.0142, 7 993.0223的3个蛋白质峰差异具有统计学意义, 用该分类树模型作为生物标志物来检测, 在学习模式下分组正确率均是100%(43/43), 在测试模式下分组正确率65.1%(28/43)和67.4%(29/43); 汉族组与哈萨克族食管癌组中质荷比为4 310.0184, 8 167.9277, 8 158.1117, 13 789.4864, 8 067.7056, 4 611.9098, 7 993.4422, 16 146.8706的8个蛋白质峰差异具有统计学意义(P<0.05), 用4 310.01843, 13 789.4864, 8 067.70556这3个蛋白质峰建立了汉族、哈萨克族食管癌血清蛋白质指纹图谱验证模型. 用该分类树模型作为生物标志物来检测, 在学习模式下分组正确率分别为86.0%(37/43)和87.8%(36/41), 在测试模式下分组正确率79.1%(34/43), 73.2%(30/41). 维吾尔族组与哈萨克族组中质荷比分别为9 161.7944, 4 611.6342, 6 649.6163, 4 979.3807的4个蛋白质峰差异具有统计学意义(P<0.05), 用4 611.6342, 4 979.3807, 5 672.3799这3个蛋白质峰建立了维吾尔族、哈萨克族食管癌血清蛋白指纹图谱验证模型. 用该分类树模型作为生物标志物来检测, 在学习模式下分组正确率分别为81.4%(35/43)和65.9%(27/41), 在测试模式下分组正确率67.4%(29/43), 48.8%(20/41). 本研究发现质荷比4 310.0109的蛋白峰在汉族组中呈低表达, 而在维吾尔族组和哈萨克族组中均呈高表达. 在维吾尔族组和哈萨克族组之间该蛋白峰未见到有差异. 提示该蛋白可能是食管癌种族差异的特异蛋白. 本实验所建立的验证模型的分组正确率相对较低, 与软件在选取建立模型的蛋白峰有关, 有些选取的蛋白峰两组比较中并无差异.
SELDI-TOF-MS对不同民族食管癌差异蛋白的检测具有良好的应用前景. 由于肿瘤细胞的复杂性, 联合其他肿瘤标志物共同检测可以提高肿瘤诊断准确性. 为了进一步了解肿瘤的基因和蛋白质的特点, 我们还需要开发功能更加强大的生物信息工具[31]. 当然, SEILD-TOF-MS技术也有其局限性, 还需不断研究, SEILD-TOF-MS技术可以得出蛋白或肽段的近似分子质量, 但却无法鉴定是何种蛋白[32], 对同一病例组织和血清的蛋白谱进行分析比较, 分离纯化和鉴定这些蛋白将是以后实验的重点. 这将有助于进一步更深层次的理解食管癌蛋白组学改变和食管癌种族差异发生机制的认识.
食管癌是世界上最常见的恶性肿瘤之一, 居世界恶性肿瘤死亡的第6位, 难于早期诊断, 仅有少数患者能够及时接受手术, 因此在食管癌临床治疗没有取得突破性进展之前, 研究重点是对食管癌患者的血清蛋白质指纹图谱进行对比研究, 筛选血清特异性蛋白质, 以此来早期发现食管癌患者.
李淑德, 主任医师, 中国人民解放军第二军医大学长海医院消化内科.
采用新的蛋白质组学方法, 可以直接对食管癌患者血清进行分析, 具有灵敏度高、检测样本量大的优点, 其中, SELDI蛋白指纹技术作为一种较新的质谱技术, 擅长于检测小分子低丰度蛋白分子. 目前其在美国已应用于卵巢癌和前列腺癌的诊断, 其特异性和敏感性远远高于传统方法.
王庆荣等应用SELDI-TOF-MS对新疆302例血清样本进行族别、性别之间的血清蛋白质指纹图谱的比较. 结果显示, 不同民族间的血清蛋白质指纹图谱存在明显差异, 汉族血清中4个差异蛋白质的丰度均低于维吾尔族和哈萨克族.
实验再次验证SELDI-TOF-MS技术是筛选食管癌患者的血清潜在肿瘤标志物的方法之一. 本研究首次发现质荷比4 310.0109的蛋白峰在汉族组中呈低表达, 而在维吾尔族组和哈萨克族组中均呈高表达. 在维吾尔族组和哈萨克族组之间该蛋白峰未见到有差异. 提示该蛋白可能是食管癌种族差异的特异蛋白.
本文采用SELDI-TOF-MS技术建立食管癌组织蛋白谱, 并筛选出一些标志蛋白, 为下一步进行分离纯化鉴定这些蛋白提供了依据, 有助于更深层次的理解食管癌的蛋白组学改变.
表面增强激光解吸电离飞行时间质谱技术(SELDI-TOF-MS): 该技术主要是在飞行质谱的检测系统中, 芯片特异性地与被测标本中蛋白结合, 再通过选择性清洗, 获得高分辨率的保留蛋白质谱, 当加入能量吸收分子后, 芯片上保留的蛋白形成晶体, 在激光照射下, 晶体发生解离作用, 带电分子在通过电场时加速, 检测仪记录飞行时间的长短, 质荷比(M/Z)越小, 飞行时间越短, 就会被最先检测到. 被测定蛋白质以一系列峰值的形式呈现.
本研究临床指导意义大, 研究手段先进, 结果可靠.
编辑: 李军亮 电编: 吴鹏朕
| 1. | Lam AK. Molecular biology of esophageal squamous cell carcinoma. Crit Rev Oncol Hematol. 2000;33:71-90. [PubMed] [DOI] |
| 2. | Parkin DM, Bray F, Ferlay J, Pisani P. Estimating the world cancer burden: Globocan 2000. Int J Cancer. 2001;94:153-156. [PubMed] [DOI] |
| 3. | Pearson FG, Cooper JD, Deslauriers J, Ginsberg RJ, Hiebert CA, Patterson GA, Urschel HC Jr. Churchill Livingstone, Elsevicr Science. Kidlington, UK: Churchill Livingstone, Elsevicr Science 2002; 714. |
| 5. | Smith RD. Probing proteomes--seeing the whole picture? Nat Biotechnol. 2000;18:1041-1042. [PubMed] [DOI] |
| 6. | Issaq HJ, Conrads TP, Prieto DA, Tirumalai R, Veenstra TD. SELDI-TOF MS for diagnostic proteomics. Anal Chem. 2003;75:148A-155A. [PubMed] |
| 7. | Weinberger SR, Dalmasso EA, Fung ET. Current achievements using ProteinChip Array technology. Curr Opin Chem Biol. 2002;6:86-91. [PubMed] [DOI] |
| 8. | Anderson NL, Anderson NG. The human plasma proteome: history, character, and diagnostic prospects. Mol Cell Proteomics. 2002;1:845-867. [PubMed] [DOI] |
| 9. | Bergman AC, Benjamin T, Alaiya A, Waltham M, Sakaguchi K, Franzén B, Linder S, Bergman T, Auer G, Appella E. Identification of gel-separated tumor marker proteins by mass spectrometry. Electrophoresis. 2000;21:679-686. [PubMed] [DOI] |
| 10. | He QY, Chen J, Kung HF, Yuen AP, Chiu JF. Identification of tumor-associated proteins in oral tongue squamous cell carcinoma by proteomics. Proteomics. 2004;4:271-278. [PubMed] [DOI] |
| 11. | Garbis S, Lubec G, Fountoulakis M. Limitations of current proteomics technologies. J Chromatogr A. 2005;1077:1-18. [PubMed] [DOI] |
| 12. | Yip TT, Hutchens TW. Mapping and sequence-specific identification of phosphopeptides in unfractionated protein digest mixtures by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. FEBS Lett. 1992;308:149-153. [PubMed] [DOI] |
| 13. | Rosty C, Christa L, Kuzdzal S, Baldwin WM, Zahurak ML, Carnot F, Chan DW, Canto M, Lillemoe KD, Cameron JL. Identification of hepatocarcinoma-intestine-pancreas/pancreatitis-associated protein I as a biomarker for pancreatic ductal adenocarcinoma by protein biochip technology. Cancer Res. 2002;62:1868-1875. [PubMed] |
| 14. | Petricoin EF, Ardekani AM, Hitt BA, Levine PJ, Fusaro VA, Steinberg SM, Mills GB, Simone C, Fishman DA, Kohn EC. Use of proteomic patterns in serum to identify ovarian cancer. Lancet. 2002;359:572-577. [PubMed] [DOI] |
| 15. | Wang LD, Zhou Q, Feng CW, Liu B, Qi YJ, Zhang YR, Gao SS, Fan ZM, Zhou Y, Yang CS. Intervention and follow-up on human esophageal precancerous lesions in Henan, northern China, a high-incidence area for esophageal cancer. Gan To Kagaku Ryoho. 2002;29 Suppl 1:159-172. [PubMed] |
| 16. | Zhang Z, Bast RC, Yu Y, Li J, Sokoll LJ, Rai AJ, Rosenzweig JM, Cameron B, Wang YY, Meng XY. Three biomarkers identified from serum proteomic analysis for the detection of early stage ovarian cancer. Cancer Res. 2004;64:5882-5890. [PubMed] [DOI] |
| 17. | 胡 朝军, 李 永哲, 赵 冠飞, 李 宁, 许 洋, 佟 大伟, 张 蜀澜. 应用蛋白指纹图谱技术筛选原发性胆汁性肝硬化患者血清特异性标志物. 世界华人消化杂志. 2008;16:277-283. [DOI] |
| 18. | Vlahou A, Schellhammer PF, Mendrinos S, Patel K, Kondylis FI, Gong L, Nasim S, Wright Jr GL. Development of a novel proteomic approach for the detection of transitional cell carcinoma of the bladder in urine. Am J Pathol. 2001;158:1491-1502. [PubMed] [DOI] |
| 19. | Sauter ER, Zhu W, Fan XJ, Wassell RP, Chervoneva I, Du Bois GC. Proteomic analysis of nipple aspirate fluid to detect biologic markers of breast cancer. Br J Cancer. 2002;86:1440-1443. [PubMed] [DOI] |
| 20. | Carrette O, Demalte I, Scherl A, Yalkinoglu O, Corthals G, Burkhard P, Hochstrasser DF, Sanchez JC. A panel of cerebrospinal fluid potential biomarkers for the diagnosis of Alzheimer's disease. Proteomics. 2003;3:1486-1494. [PubMed] [DOI] |
| 21. | Lewczuk P, Esselmann H, Groemer TW, Bibl M, Maler JM, Steinacker P, Otto M, Kornhuber J, Wiltfang J. Amyloid beta peptides in cerebrospinal fluid as profiled with surface enhanced laser desorption/ionization time-of-flight mass spectrometry: evidence of novel biomarkers in Alzheimer's disease. Biol Psychiatry. 2004;55:524-530. [PubMed] [DOI] |
| 22. | Uchida T, Fukawa A, Uchida M, Fujita K, Saito K. Application of a novel protein biochip technology for detection and identification of rheumatoid arthritis biomarkers in synovial fluid. J Proteome Res. 2002;1:495-499. [PubMed] [DOI] |
| 23. | Prietoa D, Conradsa TP, Scudierob DA; Veenstraa TD, Issaq HJ. Profiling of Secreted Proteins from Human Ovarian Cancer Cell Lines by Surface-Enhanced Laser Desorption Ionization Time-of-Flight Mass Spectrometry. J Liquid Chromatogr Relat Technol. 2003;26:2315-2328. [DOI] |
| 24. | Shiwa M, Nishimura Y, Wakatabe R, Fukawa A, Arikuni H, Ota H, Kato Y, Yamori T. Rapid discovery and identification of a tissue-specific tumor biomarker from 39 human cancer cell lines using the SELDI ProteinChip platform. Biochem Biophys Res Commun. 2003;309:18-25. [PubMed] [DOI] |
| 25. | Jr GW, Cazares LH, Leung SM, Nasim S, Adam BL, Yip TT, Schellhammer PF, Gong L, Vlahou A. Proteinchip(R) surface enhanced laser desorption/ionization (SELDI) mass spectrometry: a novel protein biochip technology for detection of prostate cancer biomarkers in complex protein mixtures. Prostate Cancer Prostatic Dis. 1999;2:264-276. [PubMed] [DOI] |
| 26. | Verma M, Wright GL, Hanash SM, Gopal-Srivastava R, Srivastava S. Proteomic approaches within the NCI early detection research network for the discovery and identification of cancer biomarkers. Ann N Y Acad Sci. 2001;945:103-115. [PubMed] [DOI] |
| 27. | Paweletz CP, Trock B, Pennanen M, Tsangaris T, Magnant C, Liotta LA, Petricoin EF. Proteomic patterns of nipple aspirate fluids obtained by SELDI-TOF: potential for new biomarkers to aid in the diagnosis of breast cancer. Dis Markers. 2001;17:301-307. [PubMed] [DOI] |
| 28. | Gelfi C, De Palma S, Ripamonti M, Eberini I, Wait R, Bajracharya A, Marconi C, Schneider A, Hoppeler H, Cerretelli P. New aspects of altitude adaptation in Tibetans: a proteomic approach. FASEB J. 2004;18:612-614. [PubMed] [DOI] |
| 29. | He P, He HZ, Dai J, Wang Y, Sheng QH, Zhou LP, Zhang ZS, Sun YL, Liu F, Wang K. The human plasma proteome: analysis of Chinese serum using shotgun strategy. Proteomics. 2005;5:3442-3453. [PubMed] [DOI] |
| 31. | Zhukov TA, Johanson RA, Cantor AB, Clark RA, Tockman MS. Discovery of distinct protein profiles specific for lung tumors and pre-malignant lung lesions by SELDI mass spectrometry. Lung Cancer. 2003;40:267-279. [PubMed] [DOI] |