修回日期: 2010-02-15
接受日期: 2010-02-23
在线出版日期: 2010-04-28
目的: 研究MAL基因在胃癌和癌旁组织中的甲基化状态及其mRNA表达.
方法: 应用甲基化特异性PCR(MSP)和实时荧光定量RT-PCR法检测37例人胃癌组织及对应癌旁5 cm组织中MAL基因的甲基化状态和mRNA表达, 并分别将其与各临床病理指标进行统计学分析.
结果: 37例胃癌组织中MAL基因甲基化率为78.4%. 对应37例癌旁组织中MAL基因甲基化率为5.4%. 癌组织与癌旁组织的甲基化率差异有统计学意义(P<0.01). 胃癌组织中MAL基因甲基化状态与患者年龄、性别、肿瘤大小、有无淋巴结转移、胃癌分化程度、临床分期无统计学意义(P>0.05). 在胃癌组织与对应癌旁组织中MAL基因mRNA定量表达差异有统计学意义(P<0.05), 胃癌组织中MAL基因mRNA定量表达与患者年龄、性别、肿瘤大小、胃癌分化程度、有无淋巴结转移、有无神经束浸润及肿瘤TNM分期无相关(P>0.05).
结论: 胃癌组织中MAL基因表达缺失或低下, 并且存在高甲基化率, MAL基因发生甲基化可能与胃癌发生有关.
引文著录: 林若阳, 沈萍萍, 黄智铭, 郑建建, 王良兴. MAL基因在胃癌组织中的甲基化及其mRNA的表达. 世界华人消化杂志 2010; 18(12): 1232-1236
Revised: February 15, 2010
Accepted: February 23, 2010
Published online: April 28, 2010
AIM: To investigate the mRNA expression and DNA methylation of the MAL gene in gastric carcinoma.
METHODS: Methylation-specific PCR (MSP) and real-time reverse transcription-polymerase chain reaction (RT-PCR) were used to detect DNA methylation and mRNA expression of the MAL gene in 37 gastric carcinoma samples and matched tumor-adjacent tissue samples, respectively. The correlations of DNA methylation and mRNA expression of the MAL gene with clinical and pathological parameters in gastric carcinoma were then analyzed.
RESULTS: The methylation rate of the MAL gene is 78.4% (29/37) in gastric carcinoma tissue and 5.4% (2/37) in tumor-adjacent tissue, with a significant difference between the two groups (P < 0.01). MAL gene methylation is not associated with age, sex, tumor size, lymph node metastasis, tumor differentiation and clinical stage (all P > 0.05). The expression level of the MAL mRNA differed significantly between gastric carcinoma tissue and tumor-adjacent tissue (P < 0.05). MAL mRNA expression is not associated with age, sex, tumor size, tumor differentiation, lymph node metastasis and clinical stage (all P > 0.05).
CONCLUSION: Loss of MAL gene expression plays an important role in the development and progression of gastric carcinoma. DNA methylation may negatively regulate the expression of the MAL gene.
- Citation: Lin RY, Shen PP, Huang ZM, Zheng JJ, Wang LX. MAL mRNA expression and DNA methylation in gastric cancer. Shijie Huaren Xiaohua Zazhi 2010; 18(12): 1232-1236
- URL: https://www.wjgnet.com/1009-3079/full/v18/i12/1232.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i12.1232
胃癌的发生、发展涉及多种癌基因与抑癌基因的表达异常. 启动子甲基化是肿瘤中普遍存在的现象, 常发生在肿瘤抑癌基因, 抑制启动子功能, 使基因不能正常表达, 导致肿瘤的发生. MAL基因作为一种较新型的抑癌基因, 目前逐渐受到关注, 但国内未见其有关胃癌的报道. 本研究通过甲基化特异性PCR及实时荧光定量PCR检测MAL基因在胃癌组织中mRNA水平表达和甲基化状态, 探讨该基因甲基化对胃癌组织中MAL mRNA表达的影响.
收集2009-01/02浙江省温州医学院附属第一医院胃癌患者手术切除新鲜标本33例, 所有患者均经病理确诊, 包括不同临床分期和不同组织学类型, 其中女13例, 男24例, 年龄47-80岁, 所有患者术前均未接受放、化疗. 每份标本均取癌组织、对应癌旁组织(距癌灶边缘5 cm以外)各2块, 液氮休克后-80 ℃保存. Fermentas DNA提取试剂盒(MBI公司); EZ DNA甲基化试剂盒-Gold(北京天漠公司); Hot start Taq DNA聚合酶(大连宝生物公司); SssⅠ酶(New England Biolabs公司); TRIzol试剂(invitrogen公司); 逆转录酶(MBI公司); SYBR Green(Applied Biosystems公司); 普通PCR仪(Mycycler, BIO-RAD公司); Real Time PCR仪(ABI7500, Applied Biosystems公司); 用于methylation-specific PCR(MSP)的甲基化引物(M)和非甲基化引物(U)由上海生工合成, 用于Real-time RT-PCR的引物由上海英骏公司合成.
1.2.1 甲基化特异性PCR: 组织DNA按照DNA提取试剂盒说明书提取后, 紫外分光光度计检测DNA浓度及纯度(A260/A280>1.8). 取小于2 µg DNA按照EZ DNA甲基化试剂盒-Gold说明书进行亚硫酸氢钠的修饰与DNA的纯化后, 用于PCR扩增. PCR反应体系: 10×Buffer 2 µL, dNTP Mixture(2.5 mmol/L)3.2 µL, 上下游引物(10 µmol/L)各1 µL, 修饰后的DNA 4 µL, Hot start Taq DNA聚合酶(5 U/µL) 0.2 µL, 去离子水补足至20 µL. 95 ℃预变性15 min之后进行35个循环反应: 95 ℃变性1 min, 52 ℃复性1 min, 72 ℃延伸30 s. 最后, 72 ℃延伸7 min. 甲基化引物(M)和非甲基化引物(U)序列根据外文文献[1], 见表1. MSP产物由2%琼脂糖凝胶(含0.1 g/L溴化乙锭)电泳检测. 以CpG甲基化转移酶(Sss.Ⅰ酶)处理后的健康人基因组DNA作甲基化阳性对照, 未经该酶处理的健康人基因组DNA为非甲基化阳性对照, 以双蒸水作阴性对照.
| 引物 | 上游5'-3' | 下游5'-3' | 扩增产物(bp) | 退火温度(℃) |
| MAL-M | TTCGGGTTTTTTTGTTTTTAATTC | GAAAACCATAACGACGTACTAACGT | 139 | 56 |
| MAL-U | TTTTGGGTTTTTTTGTTTTTAATTT | ACAAAAACCATAACAACATACTAACATC | 142 | 56 |
| MAL-P | GCAAGACGGCTTCACCTACAG | GCAGAGTGGCTATGTAGGAGAACA | 74 | 59 |
| 18SrRNA | CGAGCCGCCTGGATACC | CCTCAGTTCCGAAAACCAACAA | 76 | 61 |
1.2.2 实时荧光定量RT-PCR: 组织RNA按照TRIzol试剂说明书提取, 并紫外分光光度计检测RNA浓度及纯度(A260/A280>1.8). 取4 µg总RNA进行逆转录合成cDNA, 逆转录条件按逆转录酶说明书进行. 合成后的cDNA用于实时荧光定量PCR, 其体系为: 1 µL cDNA,上下游引物的混合物(10 µmol/L)1 µL, 7.5 µL SYBR Green, 去离子水补足至15 µL. 反应条件: 95 ℃预变性10 min, 95 ℃变性15 s, 60 ℃复性15 s, 72 ℃延伸45 s, 共40个循环反应. 采用18SrRNA为内参. Real-time RT-PCR引物序列根据外文文献[2], 见表1. 电泳检测扩增产物条件同前.
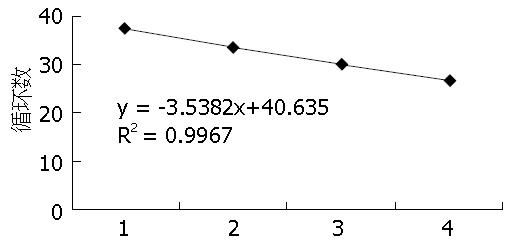
1.2.3 标准曲线的绘制: 将逆转录后的cDNA按照10倍, 100倍, 1 000倍浓度梯度稀释进行实时荧光定量PCR测得四个不同浓度下荧光信号到达设定的阈值时所经历的循环数(threshold cycle, Ct)值绘制标准曲线.
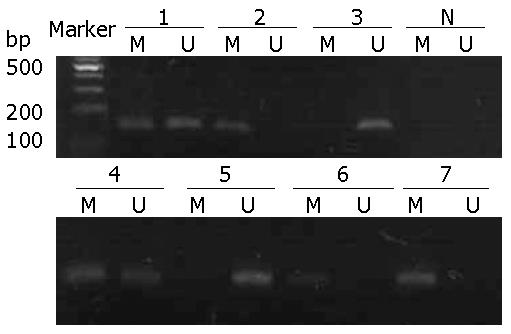
1.2.4 MSP结果判定: 甲基化和非甲基化引物阳性的目的条带分别为139 bp和142 bp, 如果甲基化引物扩增后出现大小为139 bp特异性条带, 则认为启动子区域存在甲基化, 若非甲基化引物扩增后出现大小为142 bp特异性条带, 则认为启动子区域存在非甲基化, 甲基化和非甲基化引物扩增后同时出现139 bp和142 bp特异性条带为部分甲基化, 也认为启动子区域存在甲基化.
统计学处理 用Pfaffi法计算胃癌和癌旁组织中MAL基因mRNA表达差异倍数, 以mean±SD表示, 采用样本t检验或近似t检验进行统计学分析. 计数资料采用Fisher精确概率法和Ridit分析. 所有数据运用SPSS14.0软件进行分析. 以P<0.05差异有统计学意义.
37例胃癌组织中有29例检测到甲基化, 甲基化率为78.4%. 对应37例癌旁组织2例出现甲基化, 甲基化率为5.4%. 癌组织与癌旁组织之间统计学分析有非常显著性差异(P<0.01, 图1). 胃癌组织中MAL基因甲基化状态与患者年龄、性别、肿瘤大小、有无淋巴结转移、胃癌分化程度、临床分期无统计学意义(P>0.05, 表2).
| 临床病理指标 | n | 甲基化阳性(n) | P值 | Pfaffi法结果 | P值 |
| 性别 | |||||
| 男 | 24 | 17 | 0.216 | 0.8870±0.3978 | 0.907 |
| 女 | 13 | 12 | 0.9016±0.3105 | ||
| 年龄(岁) | |||||
| ≥60 | 23 | 16 | 0.123 | 0.8427±0.3762 | 0.361 |
| <60 | 14 | 13 | 0.9608±0.3403 | ||
| 分化程度 | |||||
| 低分化 | 24 | 20 | 0.413 | 0.8832±0.4007 | 0.646 |
| 中分化与 | |||||
| 高分化 | 13 | 9 | 0.9553±0.2926 | ||
| 淋巴转移 | |||||
| 有 | 24 | 18 | 0.685 | 0.9672±0.4001 | 0.144 |
| 无 | 13 | 11 | 0.7783±0.2636 | ||
| 肿瘤大小(cm) | |||||
| ≥5 | 18 | 16 | 0.232 | 0.9449±0.4347 | 0.474 |
| <5 | 19 | 13 | 0.8493±0.2915 | ||
| 神经束浸润 | |||||
| 有 | 18 | 15 | 0.693 | 0.9113±0.4457 | 0.780 |
| 无 | 19 | 14 | 0.8754±0.2710 | ||
| TNM分期 | |||||
| 0+Ⅰ+Ⅱ | 10 | 7 | 0.655 | 0.7791±0.2183 | 0.070 |
| Ⅲ+Ⅳ | 27 | 22 | 1.0135±0.4350 | ||
| 癌组织 | 37 | 29 | 0.000 | 0.044 | |
| 癌旁组织 | 37 | 2 |
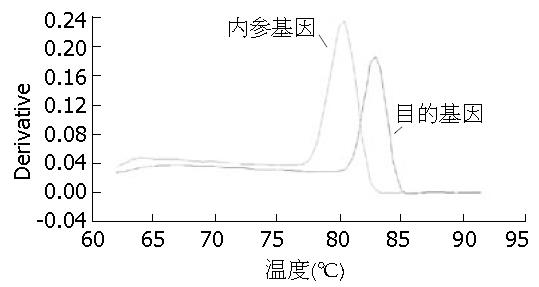
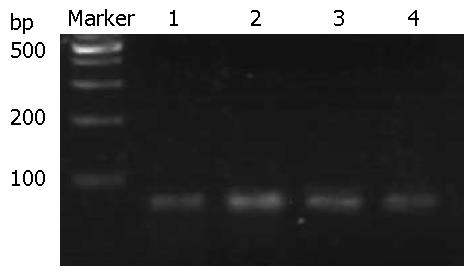
通过溶解曲线分析扩增产物显示为单峰(图2), 提示产物特异性高, 定量准确, 扩增片段经2%琼脂糖电泳, 扩增片段长度与理论大小一致(图3). 以cDNA稀释倍数取对数值为横坐标, 以对应Ct值为纵坐标作图, 做出的标准曲线(图4), 回归系数为0.9967, 接近1, 说明直线性好并计算MAL基因扩增效率为92%. 同样方法计算内参基因的扩增效率为106%, 两者均接近100%, 但相差大于5%, 用Pfaffi法计算MAL基因的mRNA相对表达水平, 公式如下: 表达水平 = 目的基因扩增效率(癌组织目的基因Ct值-癌旁组织目的基因Ct值)/内参基因扩增效率(癌旁组织内参基因Ct值-癌组织内参基因Ct值), 结果显示MAL基因mRNA在胃癌组织与对应癌旁组织中表达有统计学差异(P<0.05), 且发现其在胃癌组织中的表达下调与患者年龄、性别、肿瘤大小、胃癌分化程度、有无淋巴结转移、有无神经束浸润及肿瘤TNM分期无统计学意义(P>0.05, 表1).
MAL基因是Alonson等[3]在1987年发现的一个在T细胞分化中晚期时表达的基因. MAL基因组定位于人染色体2cen-q13, 全长21 kb, 相对分子质量16.7103 Da, 有4个外显子和3个内含子. MAL基因编码T细胞分化蛋白, 与BENE、Plasmolipin、EST克隆H09290同属于一个大的基因家族. 结构分析显示, 他们所编码的蛋白均为四折跨膜蛋白质, 是不溶于去垢剂的富含糖脂和胆固醇的膜微结构域成分. 其成员在多种组织如食管、胃肠道、肺、肾等广泛表达, 而且不同种属之间有同源蛋白质, 提示他们在细胞中有重要的功能. 1997年Millán等[4]提出MAL参与顶端传送体的形成, 可能是高尔基体和远端质膜之间囊泡转运和蛋白质分选的一个功能成分. 顶端运输是一些特定的细胞极性运输脂肪和蛋白成分至顶膜, 这种运输方式是上皮细胞所固有的, 功能极性运输功能的丧失往往与恶性转化过程有关[5]. 目前已经证实MAL在顶端运输中发挥作用. MAL基因涉及细胞内运输、基因表达、免疫调节等重要方面. 目前多项研究显示, MAL具有抑瘤功能, 能抑制肿瘤细胞的活性、侵袭性和致肿瘤性. MAL基因表达缺失和人类多种恶性肿瘤发生、发展有关.
Mimori等[6]在研究食管癌组织基因表达时发现食管癌发生、发展与MAL基因表达关系密切, 通过微点阵技术发现MAL基因在食管癌中比正常食管上皮表达显著下调, 其在正常食管上皮细胞中强烈表达, 在食管癌细胞中不表达. Wang等[7]采用非放射性RNA原位杂交方法对33例食管癌及其癌旁黏膜组织进行了MAL基因的检测和分析, 发现所有正常食管上皮组织均表达MAL基因, 只有8例肿瘤细胞表达MAL, 两者MAL基因的表达有显著差异, 当将肿瘤组织的表达情况与临床各参数分析时, MAL基因与病理分级、原发病灶、淋巴结转移、远处转移、临床分期等无关, 提示该基因异常可能是食管癌发生中的较早期事件. 本研究结果显示MAL mRNA在胃癌组织与对应癌旁组织中表达有统计学差异(P<0.05), 可见MAL mRNA的表达下调与胃癌的发生有一定关系. 其在胃癌组织中的表达下调与患者年龄、性别、肿瘤大小、胃癌分化程度、有无淋巴结转移、有无神经束浸润及肿瘤TNM分期无统计学意义(P>0.05).
DNA甲基化是最早发现的基因表观修饰方式之一, 他是指在DNA甲基转移酶(DNA methyltransferase, DNMT)催化下, 以S-腺苷甲硫氨酸为甲基供体, 将甲基转移到特定碱基上的过程. DNA甲基化能关闭某些基因的活性, 去甲基化则诱导了基因的重新活化和表达. Lind等[1]通过MSP及测序的方法显示肠腺瘤与肠癌组织中较正常肠黏膜组织MAL基因发生甲基化的频率高, 分别是71%与80%, 而正常组织甲基化率为4%; 在肠癌细胞株中, MAL基因甲基化水平与其mRNA表达缺失相关; 在肠癌标本中, MAL mRNA表达阳性的组织中其甲基化水平低于表达阴性组织, 但在统计学上无意义. 本研究中结果显示37例胃癌组织的MAL基因甲基化率为78.4%, 37例癌旁组织的MAL基因甲基化率为5.4%, 癌组织与癌旁组织的甲基化率的差异有统计学意义(P<0.01), 表明在胃癌组织中MAL基因存在着高甲基化. MAL mRNA表达不同的胃癌组织中MAL基因的甲基化水平有差异, 由此表明MAL基因甲基化可能是其表达缺失或低下的重要原因. 我们还发现, 胃癌组织中MAL基因甲基化状态与患者肿瘤大小、有无淋巴结转移、胃癌分化程度、临床分期无统计学意义(P>0.05), 可能存在以下原因: (1)MAL基因甲基化发生于胃癌早期; (2)基因启动子甲基化区域、水平存在差异造成了统计学上无意义. Buffart等[8]对MAL基因启动子上两个发生了甲基化的区域进行了研究, 分别是M1区[(-680)-(-573) bp]和M2区[(-92)-(-7) bp], 不同胃癌组织在这两个区域发生甲基化率不同, 其单变量分析显示MAL基因甲基化率在胃癌组织病理类型、胃癌患者临床分期上无统计学意义; (3)还存在其他基因表达调节机制和癌细胞侵袭转移机制.
总之, MAL基因表达在胃癌组织中异常减少或缺失, 胃癌组织中MAL基因存在高甲基化, 其甲基化可能是该基因失活的重要方面. MAL基因若要作为胃癌诊断指标、指导预后和治疗靶点应用于临床仍有待进一步研究.
胃癌在我国是最常见的恶性肿瘤之一. 胃癌发生的分子机制中, 遗传和表观遗传机制均起着重要的作用. DNA甲基化是目前研究最多也最深入的一种表观遗传学表达机制. 胃癌相关基因尤其是抑癌基因的启动子区异常甲基化导致其表达失活是胃癌发生、发展重要因素之一, 现已成为胃癌发病机制研究中的热点.
于颖彦, 教授, 上海交通大学医学院附属瑞金医院器官移植中心病理室; 王晓艳, 副教授, 湖南长沙中南大学湘雅三医院消化内科
MAL基因若要作为胃癌诊断指标、指导预后和治疗靶点应用于临床仍有待进一步研究.
Mimori等在研究食管癌组织基因表达时发现食管癌发生、发展与MAL基因表达关系密切, 通过微点阵技术发现MAL基因在食管癌中比正常食管上皮表达显著下调, 其在正常食管上皮细胞中强烈表达, 在食管癌细胞中不表达.
本研究设计基本合理, 内容新颖, 有一定的临床意义.
编辑: 李军亮 电编: 吴鹏朕
| 1. | Lind GE, Ahlquist T, Kolberg M, Berg M, Eknaes M, Alonso MA, Kallioniemi A, Meling GI, Skotheim RI, Rognum TO. Hypermethylated MAL gene - a silent marker of early colon tumorigenesis. J Transl Med. 2008;6:13. [PubMed] [DOI] |
| 2. | Wilting SM, de Wilde J, Meijer CJ, Berkhof J, Yi Y, van Wieringen WN, Braakhuis BJ, Meijer GA, Ylstra B, Snijders PJ. Integrated genomic and transcriptional profiling identifies chromosomal loci with altered gene expression in cervical cancer. Genes Chromosomes Cancer. 2008;47:890-905. [PubMed] [DOI] |
| 3. | Alonso MA, Weissman SM. cDNA cloning and sequence of MAL, a hydrophobic protein associated with human T-cell differentiation. Proc Natl Acad Sci U S A. 1987;84:1997-2001. [PubMed] [DOI] |
| 4. | Millán J, Puertollano R, Fan L, Rancaño C, Alonso MA. The MAL proteolipid is a component of the detergent-insoluble membrane subdomains of human T-lymphocytes. Biochem J. 1997;321:247-252. [PubMed] [DOI] |
| 5. | Marazuela M, Alonso MA. Expression of MAL and MAL2, two elements of the protein machinery for raft-mediated transport, in normal and neoplastic human tissue. Histol Histopathol. 2004;19:925-933. [PubMed] |
| 6. | Mimori K, Nishida K, Nakamura Y, Ieta K, Yoshikawa Y, Sasaki A, Ishii H, Alonso MA, Mori M. Loss of MAL expression in precancerous lesions of the esophagus. Ann Surg Oncol. 2007;14:1670-1677. [PubMed] [DOI] |
| 7. | Wang Z, Wang M, Xu X, Xu Z, Han Y, Cai Y, Sun Y, Wu M. Studies of MAL gene in human esophageal cancer by RNA in situ hybridization. Zhonghua Yixue Yichuanxue Zazhi. 2000;17:329-331. [PubMed] |
| 8. | Buffart TE, Overmeer RM, Steenbergen RD, Tijssen M, van Grieken NC, Snijders PJ, Grabsch HI, van de Velde CJ, Carvalho B, Meijer GA. MAL promoter hypermethylation as a novel prognostic marker in gastric cancer. Br J Cancer. 2008;99:1802-1807. [PubMed] [DOI] |