修回日期: 2009-05-08
接受日期: 2009-05-11
在线出版日期: 2009-06-08
目的: 探讨5-氮-2-脱氧胞苷(5-Aza-CdR)和曲古抑菌素A(TSA)对人胃癌细胞系MGC-803生物学行为的影响.
方法: 人胃癌细胞系MGC-803按常规培养于含100 g/L小牛血清的RPMI 1640培养液中. 实验分为5组: (1)空白对照组: 不含细胞的培养液; (2)阴性对照组: 细胞只换液, 不加药物; (3) 5-Aza-CdR组: 细胞接种24 h后, 加入10.0 μmol/L的5-Aza-CdR; (4)TSA组: 细胞接种24 h后, 加入300 μg/L TSA; (5) 5-Aza-CdR+TSA组: 细胞接种24 h后, 加入10.0 μmol/L 5-Aza-CdR, 24 h后再加入300 μg/L TSA. 应用RT-PCR检测各组细胞培养72 h后细胞Runx3 mRNA的表达, MTT比色法测定各组细胞分别培养24、48、72 h后细胞增殖.
结果: 单独应用5-Aza-CdR和TSA作用于MGC-803细胞72 h后, Runx3 mRNA相对表达量增加, 联合用药组分别与5-Aza-CdR组及TSA组相比, 差异有统计学意义(0.883±0.025 vs 0.760±0.286, 0.735±0.018, 均P<0.05). 细胞生长抑制率在同一组别随着药物作用时间的延长而增加, 并且呈正相关(r = 0.738, P<0.05); 而在同一时间不同组别比较联合用药组细胞生长抑制率较单用药物组明显增加(24 h: 57.3% vs 40.4%, 39.0%; 48 h: 70.0% vs 56.0%, 51.3%; 72 h: 86.3% vs 68.0%, 65.8%, 均P<0.05). 细胞Runx3 mRNA的相对表达量及生长抑制率在药物组与对照组间比较, 差异均有统计学意义(P<0.05).
结论: 与单用5-Aza-CdR或TSA相比, 联合应用2种药物更显著的增强Runx3 mRNA的表达, 抑制细胞的增殖.
引文著录: 郑向欣, 王建. 5-氮-2-脱氧胞苷联合曲古抑菌素A对人胃癌细胞系MGC-803生物学行为的影响. 世界华人消化杂志 2009; 17(16): 1655-1659
Revised: May 8, 2009
Accepted: May 11, 2009
Published online: June 8, 2009
AIM: To explore the effects of 5-aza-2-deoxycytidine (5-Aza-CdR) and trichostatin A (TSA) on biological behavior in MGC-803 Cells.
METHODS: MGC-803 cells were cultured in RPMI 1640 culture medium containing 100 g/L fetal calf serum. The cells in experiment were at logarithmic phase, and the number of viable cells was 95%-98%. The trial consisted of five groups. (1) Blank control group: there was only culture medium and no cells or drugs; (2) Negative control group: there were cells, culture medium and no drugs; (3) 5-Aza-CdR group: 24 h after inoculation, MGC-803 cells were treated with 10.0 μmol/L 5-Aza-CdR; (4) TSA group: 24 h after inoculation, MGC-803 cells were treated with 300 μg/L TSA;. (5) 5-Aza-CdR+TSA group: 24 h after inoculation, MGC-803 cells were treated with 10.0 μmol/L 5-Aza-CdR, and then treated with 300 μg/L TSA after 24 h. After MGC-803 cells were treated with different concentrations of 5-Aza-CdR and TSA for different cultural time, the expression of Runx3 mRNA in different groups for 72 h was detected by RT-PCR, and the cell proliferation in different groups for 24, 48, 72 h was determined using MTT colorimetry.
RESULTS: When MGC-803 cells were treated with 5-Aza-CdR and TSA alone for 72 h, the relative expression of Runx3 mRNA was increased. It was significantly increased in combination group, and compared with 5-Aza-CdR group and TSA group (0.883 ± 0.025 vs 0.760 ± 0.286, 0.735 ± 0.018, both P < 0.05). The cell growth inhibition rate increased with the time prolonging in the same group and there was a positive relationship (r = 0.738, P < 0.05). The cell growth inhibition rate increased more significantly in combination group compared with 5-Aza-CdR group and TSA group in the same time (24 h: 57.3% vs 40.4%, 39.0%; 48 h: 70.0% vs 56.0%, 51.3%; 72 h: 86.3% vs 68.0%, 65.8%, all P < 0.05). There were significant differences between drug group and control group in relative expression of Runx3 mRNA and cell growth inhibition rate (P < 0.05).
CONCLUSION: Compared with 5-Aza-CdR or TSA alone, combination of the two drugs can significantly induce Runx3 mRNA expression and inhibit cell proliferation.
- Citation: Zheng XX, Wang J. Effects of 5-aza-2-deoxycytidine and trichostatin A on biological behavior in MGC-803 cells. Shijie Huaren Xiaohua Zazhi 2009; 17(16): 1655-1659
- URL: https://www.wjgnet.com/1009-3079/full/v17/i16/1655.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v17.i16.1655
Runx3(PEBP2aC/CBFA3/AML2)是新发现的一种抑癌基因, 在胃癌中主要以基因纯合或杂合缺失以及甲基化方式失活, 其中甲基化是表达下调的重要机制[1]. 为研究抑癌基因Runx3去甲基化后对胃癌细胞生物学行为的影响, 我们应用5-氮-2-脱氧胞苷(5-aza-2-deoxycytidine, 5-Aza-CdR)和曲古抑菌素A(trichostatin A, TSA)对胃癌细胞系MGC-803进行处理, 观察抑癌基因Runx3的转录表达及细胞增殖的改变.
人胃癌细胞系MGC-803为上海亚培生物科技有限公司产品. 5-Aza-CdR和TSA购于Sigma公司. 含100 g/L小牛血清的RPMI 1640培养液为美国Gibco公司产品. 总RNA提取试剂盒(TRIzol法)及AMV第一链cDNA合成试剂盒均购于上海捷瑞生物工程有限公司. MTT试剂盒购于碧云天生物技术研究所.
1.2.1 试剂配置: 5-Aza-CdR和TSA以RPMI 1640培养液(含100 g/L小牛血清)和DMSO(0.5%)溶解, 分别配成浓度为10-3 mol/L和10-4 mol/L工作母液, 用微孔滤膜除菌, -20 ℃保存, 使用时用RPMI 1640稀释成工作浓度.
1.2.2 细胞培养、分组及处理: 人胃癌细胞系MGC-803按常规培养于含100 g/L小牛血清的 RPMI 1640培养液中. 实验细胞处于对数生长期, 活细胞数达95%-98%. 实验分为5组: (1)空白对照组: 不含细胞的培养液; (2)阴性对照组: 细胞只换液, 不加药物; (3)5-Aza-CdR组: 细胞接种24 h后, 加入10.0 μmol/L的5-Aza-CdR; (4)TSA组: 细胞接种24 h后, 加入300 µg/L TSA; (5)5-Aza-CdR+TSA组: 细胞接种24 h后, 加入10.0 μmol/L 5-Aza-CdR, 24 h后再加入300 μg/L TSA.
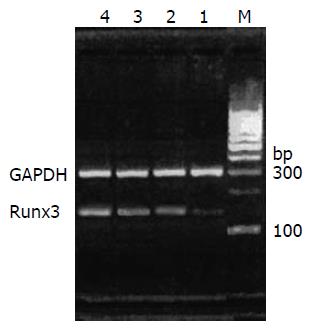
1.2.3 RT-PCR检测药物处理后细胞的Runx3 mRNA的表达: 接种细胞于培养瓶, 实验分组如前述. 培养72 h后收集细胞, 应用TRIzol法提取总RNA, 紫外分光光度仪测纯度和浓度, 以DNaseⅠ处理, 去除基因组DNA, 采用AMV法进行反转录cDNA第一链合成. 取cDNA进行PCR扩增, 引物序列及PCR产物长度序列如下. Runx3引物序列, 上游: 5'-AGGCATTGCGCAGCTCAGCGGAGT-3', 下游: 5'-TCTGCTCCGTGCTGCCCTCGCACT-3', 扩增长度156 bp; 内参照GAPDH引物序列, 上游: 5'-CCATGTTCGTCATGGGTGTGAACC-3', 下游: 5'-GCCAGTAGAGGCAGGGATGATGTT-3', 扩增长度307 bp. 目的基因Runx3及内参照GAPDH的引物均由Primer5.0软件设计. 在50 μL反应体系中, 加入3.0 μL cDNA, 上游及下游引物各1.0 μL, 2 mmol/L dNTPs 1.5 μL, 25 mmol/L MgCl2 5.0 μL, 10×Buffer 5 μL, Taq酶0.5 μL, 94 ℃预变性4 min后, 进行PCR反应: 94 ℃, 59 ℃, 72 ℃各45 s, 共35个循环, 72 ℃延伸7 min后终止反应. 以 GAPDH作为内参照, 将产物进行琼脂糖凝胶电泳, 凝胶成像分析系统测得各组条带的平均灰度值, 重复3次. 计算Runx3与GAPDH的灰度比值, 以其比值代表Runx3 mRNA的相对量.
1.2.4 MTT法检测药物作用后细胞增殖: 取70%-80%汇合的细胞, 用2.5 g/L胰蛋白酶消化单层培养细胞MGC-803, 制备成单细胞悬液, 镜下见细胞团小于5%. 以每孔5×103个细胞接种于96孔培养板(每孔200 μL), 37 ℃, 50 mL/L CO2及饱和湿度的培养箱培养24 h后分为用药组及对照组(分组同前), 每孔设3个复孔, 单用药组中分别加入10.0 μmol/L 5-Aza-CdR和300 μg/L TSA, 联合用药组加入10.0 μmol/L 5-Aza-CdR和300 ng/mL TSA, 阴性对照组只加等量培养液, 试剂及培养液每24 h更换1次. 每隔24 h取样1次, 连续3 d. 每次取样时移去培养液, 每孔加入MTT液(5 g/L)20 μL, 37 ℃培养4 h后弃去上清液, 每孔加入DMSO 150 μL, 轻轻振荡10 min, 使结晶物充分溶解, 在570 nm波长酶联免疫检测仪上测定各孔吸光度值(A). 取平均值计算抑制率: 细胞生长抑制率(%) = (对照组A-实验组A)/(对照组A-空白组A)×100%.
统计学处理 采用 SPSS13.0统计软件进行分析, 结果用mean±SD表示, 各组间变量比较采用单因素方差分析, 以P<0.05作为差异有统计学意义.
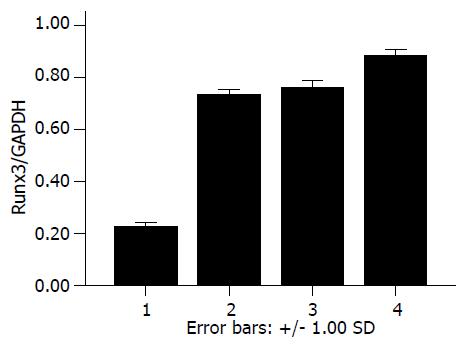
阴性对照组细胞Runx3 mRNA表达很低, 药物处理组细胞Runx3 mRNA重新表达, 10.0 μmol/L 5-Aza-CdR, 300 μg/L TSA及联合应用2种药物处理细胞72 h后, 细胞Runx3 mRNA的相对表达量分别为0.760±0.286、0.735±0.018、0.883±0.025(图1-2). 经单因素方差分析细胞Runx3 mRNA表达在实验各组之间差异有统计学意义(P<0.05), 联合用药组分别与5-Aza-CdR组及TSA组相比, 差异有统计学意义(P<0.05), 而5-Aza-CdR组与TSA组比较, 差异无统计学意义.
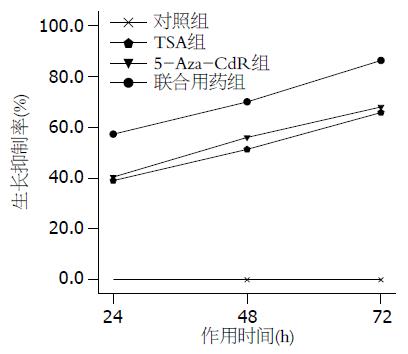
MTT比色法显示单独应用5-Aza-CdR作用MGC-803细胞24、48、72 h后A值分别为0.261±0.011、0.204±0.009、0.163±0.012, 细胞生长抑制率分别为40.4%、56.0%、68.0%; 单独应用TSA作用MGC-803细胞24、48、72 h后A值分别为0.267±0.004、0.226±0.017、0.175±0.009, 细胞生长抑制率分别为39.0%、51.3%、65.8%; 而联合应用药物作用MGC-803细胞24、48、72 h后A值分别为0.187±0.011、0.139±0.006、0.070±0.027, 细胞生长抑制率分别为57.3%、70.0%、86.3%. 经方差分析在同一时间点不同药物组之间的细胞生长抑制率差异有统计学意义(P<0.05), 联合用药组分别与单用药组间比较差异也有统计学意义(P<0.05), 而单用药组间比较差异无统计学意义; 而细胞生长抑制率在同一组别不同时间之间差异也有统计学意义(P<0.05), 在一定范围内随时间的延长而增加, 呈正相关(r = 0.738, P<0.05). 不同实验组细胞生长抑制率与药物作用时间的关系(图3).
表型遗传修饰是基因表达调控的重要形式, 包含甲基化和组蛋白去乙酰化[2]. 这种异常修饰抑制基因转录, 使抑癌基因丧失功能. 甲基转移酶抑制剂和去乙酰化酶抑制剂能够恢复组蛋白乙酰化, 去除异常的甲基化修饰, 激活抑癌基因, 发挥抑癌作用[3]. Runx3为新近克隆的一种候选抑癌基因, 位于染色体1p36.1, 是Runx家族中最小也是迄今对其研究最少的基因, 全长67 kb, 含有6个外显子和p1、p2 2个启动子, 2个启动子富含GC, p2启动子GC含量达到64%, 易发生甲基化[4]. 该基因在人类多种肿瘤中存在CpG区异常甲基化[5-8], 在胃癌发生过程中较为明显[1]. 当启动过程点甲基化后, 转录停止, Runx3失活, 可影响到TGF-β的凋亡通路, 导致凋亡抑制, 胃黏膜细胞的正常生长和凋亡失去平衡, 最终发生胃癌[9].
DNA甲基化是一种发生在CpG二核苷酸上对胞嘧啶的共价修饰物, 由S-腺苷蛋氨酸提供甲基, 在细胞内DNA甲基转移酶(DNA methyltransferase, DNMT)催化作用下, 于DNA复制后形成5-甲基胞嘧啶的反应[10]. 其中CpG岛甲基化抑制基因表达的机制有二[11]: 一是甲基化后直接抑制转录因子与模板的结合, 但这一抑制作用是有限的, 因为有一些转录因子对甲基化并不十分敏感, 甚至有许多转录因子的识别位点根本就不在CpG岛内; 二是发现一种甲基结合蛋白家族能够特异性地与甲基化位点结合, 然后与组蛋白去乙酰化酶和具有染色体变形活性的sin3a, mi-2或ski蛋白或蛋白家族形成大分子复合物, 使组蛋白脱酰基, 染色体结构变得更加紧密, 基因不能转录, 因此组蛋白去乙酰化亦可导致基因沉默. 这为组蛋白去乙酰化酶抑制剂通过抑制组蛋白脱酰基, 逆转转录抑制引起的基因沉默, 提供了广阔的舞台. 5-Aza-CdR为一种嘧啶核苷类似物, 其可以与DNMT结合形成一种共价复合物, 特异性抑制其活性, 逆转基因CpG岛甲基化, 重新激活由于高甲基化而失活的抑癌基因, 恢复抑癌基因的功能, 抑制肿瘤细胞的生长, 而且甲基化异常所致灭活的基因对DNA甲基化抑制剂非常敏感, 易重新活化[12]. TSA源自链霉菌代谢产物, 最先作为抗真菌药物使用. 近来研究表明TSA有组蛋白去乙酰化酶抑制剂(histone deacetylases inhibitor, HDACI)的特征, 可逆抑制哺乳动物组蛋白去乙酰化酶活性, 诱导肿瘤细胞周期阻滞、分化及凋亡, 抑制肿瘤细胞的生长[13]. Ou et al[14]研究表明, TSA可以广泛的诱导特定基因去甲基化并重新表达, 并证实组蛋白乙酰化和DNA去甲基化之间通过甲基结合蛋白(methyl-CpG binding proteins, MBDs)实现相互作用. 基于DNA去甲基化和组蛋白乙酰化之间存在相互作用, 我们可以通过联合应用5-Aza-CdR与TSA, 在降低各自药物浓度, 减低其不良反应的基础上, 使其充分发挥协同作用, 从而改善基因启动子区域的甲基化状态, 使沉寂的肿瘤抑癌基因重新表达, 达到治疗肿瘤的目的.
本研究基于甲基化和组蛋白去乙酰化会导致基因沉默, 抑制转录和表达, 选择甲基转移酶和去乙酰化酶作为治疗靶点. 实验中我们分别应用10.0 μmol/L 5-Aza-CdR和300 μg/L TSA作用于MGC-803细胞72 h后, 细胞Runx3 mRNA重新表达, 其相对量分别为0.760±0.286, 0.735±0.018; 联合应用10.0 μmol/L 5-Aza-CdR和300 μg/L TSA作用于MGC-803细胞72 h后, 细胞Runx3 mRNA表达明显增加, 其相对量为0.883±0.025, 与单用药组比较差异均有统计学意义(P<0.05). MTT法显示细胞生长抑制率在同一组别随着药物作用时间的延长而增加, 而在同一时间不同组别内联合用药组细胞生长抑制率较单用药物组明显增加, 以联合药物作用MGC-803细胞72 h后最高, 为86.3%. 以上结果说明药物作用于MGC-803细胞后, 抑癌基因Runx3重新表达, 发挥抑癌作用, 从而抑制肿瘤细胞的生长, 而且联合用药组效果明显好于单用药物组. 依据药物作用机制及本实验结果, 我们可以认为联合用药组Runx3 mRNA表达及细胞生长抑制率较单用药组明显升高是去甲基化和组蛋白乙酰化协同作用的结果; 也就是说药物作用后, 原来Runx3 CpG区的高甲基化状态被可逆性消除, 而且联合用药能发挥协同作用, 使得原来被封闭的肿瘤抑癌基因重新表达, 发挥抑癌作用, 从而抑制肿瘤细胞的生长, 达到治疗肿瘤的目的, 这与国外学者的研究结果一致[14].
DNA甲基转移酶抑制剂5-Aza-CdR和组蛋白去乙酰化转移酶抑制剂TSA均能使胃癌细胞抑癌基因Runx3重新表达, 抑制细胞增殖, 并且具有良好的协同作用, 二者联合应用可使得胃癌细胞Runx3 mRNA及生长抑制率较单用药组明显增加. 因此我们可以在不影响治疗效果的前提下, 联合应用这两种药物, 减少各自的药物剂量, 从而减少药物不良反应的发生, 这为进一步研究胃癌的治疗提供了新的思路. 目前我们的实验研究只是药物作用于体外细胞, 而对于这2种药物在动物实验甚至临床实验的研究, 还需要我们进一步努力.
胃癌在我国恶性肿瘤的发病率和死亡率中都居于前列, 患者由于早期临床症状不典型, 就诊时多已到中晚期, 增加了手术难度, 术后5年生存率较低, 甚至失去了手术意义. 随着人们对胃癌发生发展过程的精确解析, 越来越多的证据表明基因遗传信息的紊乱以及基因转录表达调控的紊乱是其发生的关键因素. 因此寻找有效的药物来恢复基因正常表达对胃癌治疗有着重要的意义.
邹小明, 教授, 哈尔滨医科大学附属第二医院普外二科
胃癌基因表达调控中表基因水平的甲基化和去乙酰化改变起着非常重要的作用, 而且两者在导致基因转录抑制方面具有协同作用. 针对这2种机制, 联合应用药物对其治疗已成为近年来研究的热点.
目前已有较多文献报道Runx3甲基化与胃癌发生的关系以及5-Aza-CdR与TSA恢复肿瘤抑癌基因表达, 抑制肿瘤细胞生长的作用.
本文主要以甲基化和去乙酰化作为治疗靶点, 联合应用5-Aza-CdR与TSA逆转抑癌基因Runx3甲基化, 恢复其活性, 发挥抑癌作用.
5-Aza-CdR与TSA联合应用能够发挥协同作用, 明显逆转基因甲基化, 使得沉寂的抑癌基因重新发挥功能, 并且具有较低的药物毒性作用, 在胃癌治疗上有着很好的前景.
本文有一定的科学性和创新性, 能够反映当前胃肠病学的研究水平.
编辑: 李军亮 电编: 何基才
| 1. | Waki T, Tamura G, Sato M, Terashima M, Nishizuka S, Motoyama T. Promoter methylation status of DAP-kinase and RUNX3 genes in neoplastic and non-neoplastic gastric epithelia. Cancer Sci. 2003;94:360-364. [PubMed] [DOI] |
| 2. | Jee CD, Lee HS, Bae SI, Yang HK, Lee YM, Rho MS, Kim WH. Loss of caspase-1 gene expression in human gastric carcinomas and cell lines. Int J Oncol. 2005;26:1265-1271. [PubMed] |
| 3. | Nishigaki M, Aoyagi K, Danjoh I, Fukaya M, Yanagihara K, Sakamoto H, Yoshida T, Sasaki H. Discovery of aberrant expression of R-RAS by cancer-linked DNA hypomethylation in gastric cancer using microarrays. Cancer Res. 2005;65:2115-2124. [PubMed] [DOI] |
| 4. | Bangsow C, Rubins N, Glusman G, Bernstein Y, Negreanu V, Goldenberg D, Lotem J, Ben-Asher E, Lancet D, Levanon D. The RUNX3 gene--sequence, structure and regulated expression. Gene. 2001;279:221-232. [PubMed] [DOI] |
| 5. | Guo C, Ding J, Yao L, Sun L, Lin T, Song Y, Sun L, Fan D. Tumor suppressor gene Runx3 sensitizes gastric cancer cells to chemotherapeutic drugs by downregulating Bcl-2, MDR-1 and MRP-1. Int J Cancer. 2005;116:155-160. [PubMed] [DOI] |
| 6. | Xiao WH, Liu WW. Hemizygous deletion and hypermethylation of RUNX3 gene in hepatocellular carcinoma. World J Gastroenterol. 2004;10:376-380. [PubMed] |
| 7. | Sato K, Tomizawa Y, Iijima H, Saito R, Ishizuka T, Nakajima T, Mori M. Epigenetic inactivation of the RUNX3 gene in lung cancer. Oncol Rep. 2006;15:129-135. [PubMed] |
| 8. | Ku JL, Kang SB, Shin YK, Kang HC, Hong SH, Kim IJ, Shin JH, Han IO, Park JG. Promoter hypermethylation downregulates RUNX3 gene expression in colorectal cancer cell lines. Oncogene. 2004;23:6736-6742. [PubMed] [DOI] |
| 9. | Fukamachi H. Runx3 controls growth and differentiation of gastric epithelial cells in mammals. Dev Growth Differ. 2006;48:1-13. [PubMed] [DOI] |
| 10. | Slack A, Cervoni N, Pinard M, Szyf M. Feedback regulation of DNA methyltransferase gene expression by methylation. Eur J Biochem. 1999;264:191-199. [PubMed] [DOI] |
| 11. | Jones PL, Veenstra GJ, Wade PA, Vermaak D, Kass SU, Landsberger N, Strouboulis J, Wolffe AP. Methylated DNA and MeCP2 recruit histone deacetylase to repress transcription. Nat Genet. 1998;19:187-191. [PubMed] [DOI] |
| 12. | Jones PA. DNA methylation errors and cancer. Cancer Res. 1996;56:2463-2467. [PubMed] |
| 13. | Donadelli M, Costanzo C, Faggioli L, Scupoli MT, Moore PS, Bassi C, Scarpa A, Palmieri M. Trichostatin A, an inhibitor of histone deacetylases, strongly suppresses growth of pancreatic adenocarcinoma cells. Mol Carcinog. 2003;38:59-69. [PubMed] [DOI] |
| 14. | Ou JN, Torrisani J, Unterberger A, Provençal N, Shikimi K, Karimi M, Ekström TJ, Szyf M. Histone deacetylase inhibitor Trichostatin A induces global and gene-specific DNA demethylation in human cancer cell lines. Biochem Pharmacol. 2007;73:1297-1307. [PubMed] [DOI] |