修回日期: 2008-01-08
接受日期: 2008-01-15
在线出版日期: 2008-02-08
目的: 探讨不同结肠癌细胞系中表观遗传学酶谱的表达水平.
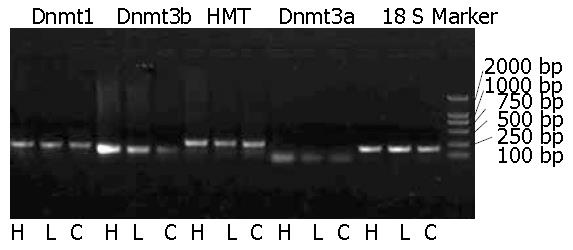
方法: RT-PCR分别检测结肠癌细胞系HT-29、Lovo和Caco-2中Dnmt1、Dnmt3a、Dnmt3b和HMT mRNA表达水平.
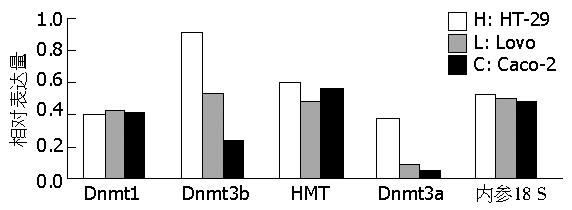
结果: Dnmt3b mRNA在结肠癌细胞系HT-29、Lovo中高度表达, Dnmt1与HMT次之, Dnmt3a表达水平最低; Dnmt3b、Dnmt3a在不同结肠癌细胞系之间的表达水平存在显著的差异.
结论: 不同分化程度的结肠癌细胞系可能存在不同的表观遗传学发生机制; Dnmt3b在建立和维持结肠癌细胞系DNA甲基化模式中可能起更为重要的角色.
引文著录: 翟荣林, 蔡开琳, 王国斌, 许飞, 田元, 张景辉. Dnmt1, Dnmt3a, Dnmt3b和HMT在不同结肠癌细胞系的表达及意义. 世界华人消化杂志 2008; 16(4): 426-430
Revised: January 8, 2008
Accepted: January 15, 2008
Published online: February 8, 2008
AIM: To determine the expression of DNA methyltransferase and histone methyltransferase (HMT) mRNA in different colonic cancer cell lines.
METHODS: Expression of Dnmt1, Dnmt3a, Dnmt3b and HMT mRNA was detected in HT-29, Lovo and Caco-2 cells by RT-PCR.
RESULTS: Dnmt3b mRNA was over-expressed in HT-29 and Lovo cells, while Dnmt1 and HMT were moderately expressed and Dnmt3a was poorly expressed in colon cancer cell lines. A significant difference was found in the expression of Dnmt3b and Dnmt3a in HT-29, Lovo and Caco-2 cells.
CONCLUSION: Differentiated colon cancer cell lines may have a different epigenetic mechanism. Dnmt3b may play a vital role in the process of establishing and maintaining the DNA methylation pattern.
- Citation: Zhai RL, Cai KL, Wang GB, Xu F, Tian Y, Zhang JH. Expression of Dnmt1, Dnmt3a, Dnmt3b and HMT in different colonic cancer cell lines and its significance. Shijie Huaren Xiaohua Zazhi 2008; 16(4): 426-430
- URL: https://www.wjgnet.com/1009-3079/full/v16/i4/426.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i4.426
基因的表遗传学修饰是一种不依赖DNA序列改变的可逆的基因表达调控过程. 主要包括DNA甲基化与去甲基化、组蛋白乙酰化与脱乙酰化和组蛋白甲基化去甲基化等改变. 这些可逆的表遗传学修饰可以方便地关闭和开放某些特定基因, 有选择性表达基因组的信息, 在维持染色体结构的稳定、基因组的完整、组织特异性基因的表达调节、胚胎发育、基因组印记、女性X染色体失活、细胞分化和个体成长等过程中发挥着至关重要的作用[1-3]. 回顾性的研究资料也表明, 肿瘤细胞中存在异常的DNA甲基化模式: 基因组整体甲基化水平降低, 导致遗传不稳定性增加; 组织特异性基因的启动子区域出现从头甲基化; 原癌基因多为低甲基化或不充分甲基化, 低甲基化使原癌基因活化, 导致重新开放或异常表达, 形成突变热点, 增加染色体的不稳定性; 某些调控细胞生长分化的基因和抑癌基因启动子因异常甲基化而失活关闭[4-6]. 这些都提示我们, 甲基化等表遗传学修饰与癌症的发生密切相关[7]. 甲基化必然离不开甲基化酶的催化作用[8], Dnmt1, Dnmt3a和Dnmt3b的过度表达在很多恶性肿瘤中都有报道[9-10], 本文分析比较了不同结肠癌细胞系中表遗传学酶谱的表达水平, 为进一步研究结肠癌表观遗传学发生机制打下基础.
DMEM(Hyclone), 胎牛血清(Gibco), TRIzol(武汉晶美), 逆转录酶SuperscriptⅡ (Invitrogen), RevertAidTM First Strand cDNA Synthesis Kit(MBI Fermentas), PCR仪(Rotor Gene 3000), PCR引物由上海生工合成.
1.2.1 结肠癌细胞株体外培养: 结肠癌细胞株HT-29、Lovo、Caco-2培养于高糖型DMEM+100 mL/L胎牛血清. 在pH7.4、37 ℃、50 mL/L CO2培养箱中孵育. 每天观察细胞生长状况, 每2 d换液, 每3-4 d传代.
1.2.2 RT-PCR检测Dnmts、HMT基因mRNA表达情况: 收集HT-29、Lovo、Caco-2细胞, 采用TRIzol分别抽提各种细胞的总RNA, 以人18S rRNA为内参照, 行逆转录PCR扩增. 15 g/L琼脂糖凝胶电泳分别检测各自扩增产物, 凝胶成像分析系统进行半定量分析, 以目的基因与内参的灰度比代表mRNA表达丰度. 反应条件: 94 ℃预变性5 min, 94 ℃变性30 s, Tm退火45 s, 72 ℃延伸30 s, 72 ℃终末延伸7 min, 共进行45个循环. 引物序列及Tm值见表1.
| Gene | Accession No. | Primer sequence | Position | Tm(℃) | Products |
| Dnmt1 | NM_001379 | 5'-TCCACAGTGTTCACAGAGG-3' | 3787-4013 | 54.0 | 227 bp |
| 5'-CGGTAGTAGTCGCAGTAGC-3' | |||||
| Dnmt3a | NM_175629 | 5'-GAAGCGGGCAAAGAACAG-3' | 780-1024 | 52.7 | 245 bp |
| 5'-TCTTCCACAGCATTCAATTCC-3' | |||||
| Dnmt3b | NM_006892 | 5'-GCTCTTACCTTACCATCG-3' | 809-937 | 48.2 | 165 bp |
| 5'-GATACTCTGAACTGTCTCC-3' | |||||
| HMT | NM_024757 | 5'-TCATCAGTCGCTACCTCAGAAC-3' | 749-1023 | 57.3 | 275 bp |
| 5'-AGTCCATCTCCAGGCTCTCC-3' | |||||
| 18S | M10098 | 5'-CGGCTACCACATCCAAGGAA-3' | 551-750 | 60.0 | 200 bp |
| 5'-GCTGGAATTACCGCGGCT-3' |
统计学处理 各组实验数据之间的比较采用方差分析t检验. P<0.05有统计学意义.
不同酶之间, 从头甲基化酶Dnmt3b mRNA在结肠癌细胞系HT-29中呈现高度表达, 维持甲基化酶Dnmt1与组蛋白甲基化酶HMT次之, 从头甲基化酶Dnmt3a mRNA在结肠癌细胞系中的表达水平最低(P<0.05, 图1, 图2). 这表明不同的DNA甲基化酶在建立和维持DNA甲基化过程中的作用和份量不同, Dnmt3b在结肠癌形成中可能扮演更为重要的角色.
不同细胞之间, 维持甲基化酶Dnmt1与组蛋白甲基化酶HMT的mRNA表达水平没有显著差异, 从头甲基化酶Dnmt3b、Dnmt3a mRNA表达水平存在明显的差异(P<0.05). HT-29细胞中Dnmt3b呈现为高度表达, Lovo和Caco-2细胞中Dnmt3b则为中低度表达; Dnmt3a也是如此, 在HT-29中表现为中度表达水平, 在Lovo和Caco-2中则几乎不表达(P<0.05, 图1, 图2). HT-29为高分化的结肠癌细胞株, 呈上皮细胞样, Caco-2为中分化结肠癌细胞, Lovo则为未分化, Dnmt3b和Dnmt3a在HT-29、Lovo和Caco-2细胞中不同表达水平提示, 不同类型与分化程度的结肠癌细胞系可能存在着迥然不同的表观遗传学发生机制, Dnmt3b、Dnmt3a尤其在分化程度良好的结肠癌细胞株中表现为高表达状态(与在正常结肠上皮中的表达水平类似), 而在分化不良或未分化的癌细胞中表现为中低表达水平.
Dnmt1的正确表达和阶段特异性的翻译后调节, 是维持胚胎正常DNA甲基化模式和顺利发育所不可或缺的[1]. Dnmt1、Dnmt3a、3b这三个基因在几乎所有的正常组织中均呈现广泛的共同表达, 在肿瘤组织中则为过度表达[9]. Dnmt1高表达是甲基化的重要因素之一, 与CpG岛甲基化紊乱有关, Dnmt1高表达总是伴有总基因组DNA低甲基化和癌基因低甲基化或抑癌基因高甲基化[11-12]; RNA干扰抑制Dnmt1表达后RASSF1A和p16等肿瘤相关基因启动子发生去甲基化而重新表达[13], Dnmt1基因敲除则与染色质不稳定性密切相关[14]. 与结肠黏膜和良性病变相比, 结肠癌组织Dnmt1和Dnmt3a表达显著增高, 结直肠癌的表遗传学调节是通过甲基化活性增强而不是去甲基化活性减弱来实现的[9]. 有研究表明, Dnmt1蛋白在胃癌和结直肠癌患者中的表达与年龄、性别不相关, 与肿瘤分化程度显著相关[15-16]. Girault et al[17]检测130例乳腺癌中Dnmt1, Dnmt3a, Dnmt3b的mRNA表达情况, 结果显示Dnmt3b的表达量最高, 而Dnmt1、Dnmt3a表达丰度较低. 我们的研究也发现, 结肠癌细胞株HT-29中Dnmt3b mRNA表现为高表达, Dnmt1和Dnmt3a则分别为中低度表达. 不过也有报道认为, 结肠癌组织中DNA甲基化酶与CpG岛的甲基化并不相关[18-19], 说明基因启动子的甲基化过程有多种蛋白和机制参与.
尽管在哺乳动物中, Dnmt1的主要功能被认为是维持甲基化, 而Dnmt3a和Dnmt3b的功能主要是从头甲基化, 有研究却表明这三个酶很可能共同参与、协同作用于维持和建立DNA甲基化模式的过程[20-22]. Dnmt1和Dnmt3a同时起作用时, 表现出近5倍的相比于各自单独作用的甲基化酶活性, Dnmt3a始发启动从头甲基化后, 半甲基化DNA底物激活Dnmt1完成后续的从头甲基化修饰, 两者在从头甲基化过程中发挥协同作用[20]. Hsieh[21]分析比较了Dnmt1和Dnmt3a的作用发现, Dnmt1可以催化基因组上瞬时出现的单链DNA(如复制起始区)的局部位点发生从头甲基化并向周围扩布, 也可以被甲基化的CpG岛底物所激活. 而Dnmt3a则不能催化单链DNA底物发生甲基化, 也不被甲基化的CpG岛所激活. 这表明尽管两者都具有从头甲基化活性, 但是作用机制却是完全不同的. 在从头甲基化过程中Dnmt3a扮演启动始发的作用, 他启动双链DNA中的其中一条链发生从头甲基化, 形成的半甲基化位点激活Dnmt1完成后续的从头甲基化和维持甲基化修饰. 深入研究发现, Dnmt1可以催化未修饰的DNA发生甲基化, 他的从头甲基化活性是通过甲基化的DNA结合到Dnmt1酶位于N端的变构位点上而被激活的[23-24].
Rhee et al[25]通过同源重组将Dnmt1基因敲除后发现, 结肠癌细胞DNA甲基转移酶活性明显下降, 然而基因组甲基化程度却只下降了20%左右, p16等很多基因仍处于甲基化致转录失活的状态. Dnmt1与Dnmt3b的共同灭活则几乎消除了甲基化转移酶的活性, 基因组DNA甲基化程度减少了95%以上, p16基因重新表达, 肿瘤细胞生长受到抑制[26]. Leu et al[27]应用RNAi下调Dnmt1和Dnmt3b在卵巢癌细胞CP70中的表达水平后发现, 单独抑制Dnmt1能部分消除目的基因(TWIST, RASSF1A, HIN-1)的高甲基化状态并诱导表达, 单独下调Dnmt3b的去甲基化和转录激活效应非常小, 但同时下调这两个酶却能明显增加去甲基化效应, 目的基因的表达增加7-15倍. Ting et al[28]的研究也表明, 即使采用RNA干扰技术下调人结肠癌细胞中Dnmt1基因的表达, 细胞基因组CpG岛甲基化模式仍然能够得以维持. 正反两方面的资料告诉我们, Dnmt不同成员在细胞中并不单独发挥作用, 他们在维持DNA甲基化和基因甲基化致沉默过程中协同作用, 共同发挥生物学效应. 此外, DNA甲基化酶(Dnmt)与组蛋白甲基化酶(HMT)之间也可能存在着协同作用[29-30], DNA CpG岛甲基化与组蛋白H3-K9甲基化在异染色质形成与基因沉默过程中共同发挥作用, 新发现的组蛋白甲基化酶(HMT)SETDB1/ESET与DNA甲基化酶尤其是从头甲基化酶Dnmt3a和Dnmt3b之间在功能上有密切的关联, 而与维持甲基化酶Dnmt1之间没有关系[31]. 然而Espada et al[32]的研究却发现, Dnmt1在维持组蛋白H3位点正常甲基化和乙酰化模式方面发挥重要的作用. 对此有待进一步的研究.
总之, 我们的研究发现, Dnmt1、Dnmt3a、Dnmt3b和HMT在结肠癌细胞系中呈现不等的表达水平, Dnmt3a、Dnmt3b在分化良好的结肠癌细胞系中呈现过度表达状况, 而在分化不良或未分化的癌细胞则被抑制表达. 这提示表遗传学酶谱之间在建立和维持DNA甲基化过程中的作用和份量可能并不相同, 不同分期与分化程度的结肠癌细胞系可能存在着不同的表观遗传学发生机制. 此外, 既然癌基因低甲基化与抑癌基因高甲基化在恶性肿瘤组织中是个普遍事件, 那么对在甲基化过程中发挥枢纽作用的甲基化酶的研究必然为结肠癌等肿瘤的防治提供了一个重要的靶点和方向. 下一步我们将在此实验资料基础上通过对特定基因的调控来进一步研究结肠癌表观遗传学发生机制.
表遗传学研究是目前国内外研究的热点之一, 回顾性的研究资料表明, 肿瘤细胞中存在异常的DNA甲基化模式, 甲基化等表遗传学修饰与癌症的发生密切相关.
曹杰, 主任医师, 广州市第一人民医院普通外科
分析不同结肠癌细胞系中表观遗传学酶谱的表达水平对研究结肠癌表观遗传学发生机制有较好的指导作用, 国内目前尚未见相关报道.
表遗传学酶谱作为甲基化等表遗传学修饰的决定因素, 对其的研究和调控在结直肠肿瘤的靶向化疗方面具有潜在的临床应用价值.
本文选题新颖, 资料合适, 数据可靠, 结论客观, 文章的科学性, 创新性和可读性较好的反映了我国结肠癌表现遗传学基础研究的先进水平.
编辑: 师忠芳 电编: 何基才
| 1. | Chung YG, Ratnam S, Chaillet JR, Latham KE. Abnormal regulation of DNA methyltransferase expression in cloned mouse embryos. Biol Reprod. 2003;69:146-153. [PubMed] [DOI] |
| 2. | Li E, Bestor TH, Jaenisch R. Targeted mutation of the DNA methyltransferase gene results in embryonic lethality. Cell. 1992;69:915-926. [PubMed] [DOI] |
| 3. | Turek-Plewa J, Jagodziński PP. The role of mammalian DNA methyltransferases in the regulation of gene expression. Cell Mol Biol Lett. 2005;10:631-647. [PubMed] |
| 4. | Herman JG, Baylin SB. Gene silencing in cancer in association with promoter hypermethylation. N Engl J Med. 2003;349:2042-2054. [PubMed] [DOI] |
| 5. | Cui H, Cruz-Correa M, Giardiello FM, Hutcheon DF, Kafonek DR, Brandenburg S, Wu Y, He X, Powe NR, Feinberg AP. Loss of IGF2 imprinting: a potential marker of colorectal cancer risk. Science. 2003;299:1753-1755. [PubMed] [DOI] |
| 6. | Jones PA, Takai D. The role of DNA methylation in mammalian epigenetics. Science. 2001;293:1068-1070. [PubMed] [DOI] |
| 7. | Luczak MW, Jagodziński PP. The role of DNA methylation in cancer development. Folia Histochem Cytobiol. 2006;44:143-154. [PubMed] |
| 8. | Bestor TH. The DNA methyltransferases of mammals. Hum Mol Genet. 2000;9:2395-2402. [PubMed] [DOI] |
| 9. | Schmidt WM, Sedivy R, Forstner B, Steger GG, Zöchbauer-Müller S, Mader RM. Progressive up-regulation of genes encoding DNA methyltransferases in the colorectal adenoma-carcinoma sequence. Mol Carcinog. 2007;46:766-772. [PubMed] [DOI] |
| 10. | Robertson KD, Uzvolgyi E, Liang G, Talmadge C, Sumegi J, Gonzales FA, Jones PA. The human DNA methyltransferases (DNMTs) 1, 3a and 3b: coordinate mRNA expression in normal tissues and overexpression in tumors. Nucleic Acids Res. 1999;27:2291-2298. [PubMed] [DOI] |
| 11. | Hermann A, Gowher H, Jeltsch A. Biochemistry and biology of mammalian DNA methyltransferases. Cell Mol Life Sci. 2004;61:2571-2587. [PubMed] [DOI] |
| 12. | Robert MF, Morin S, Beaulieu N, Gauthier F, Chute IC, Barsalou A, MacLeod AR. DNMT1 is required to maintain CpG methylation and aberrant gene silencing in human cancer cells. Nat Genet. 2003;33:61-65. [PubMed] [DOI] |
| 13. | Suzuki M, Sunaga N, Shames DS, Toyooka S, Gazdar AF, Minna JD. RNA interference-mediated knockdown of DNA methyltransferase 1 leads to promoter demethylation and gene re-expression in human lung and breast cancer cells. Cancer Res. 2004;64:3137-3143. [PubMed] [DOI] |
| 14. | Karpf AR, Matsui S. Genetic disruption of cytosine DNA methyltransferase enzymes induces chromosomal instability in human cancer cells. Cancer Res. 2005;65:8635-8639. [PubMed] [DOI] |
| 15. | Zhu YM, Huang Q, Lin J, Hu Y, Chen J, Lai MD. Expression of human DNA methyltransferase 1 in colorectal cancer tissues and their corresponding distant normal tissues. Int J Colorectal Dis. 2007;22:661-666. [PubMed] [DOI] |
| 16. | Etoh T, Kanai Y, Ushijima S, Nakagawa T, Nakanishi Y, Sasako M, Kitano S, Hirohashi S. Increased DNA methyltransferase 1 (DNMT1) protein expression correlates significantly with poorer tumor differentiation and frequent DNA hypermethylation of multiple CpG islands in gastric cancers. Am J Pathol. 2004;164:689-699. [PubMed] [DOI] |
| 17. | Girault I, Tozlu S, Lidereau R, Bièche I. Expression analysis of DNA methyltransferases 1, 3A, and 3B in sporadic breast carcinomas. Clin Cancer Res. 2003;9:4415-4422. [PubMed] |
| 18. | Eads CA, Danenberg KD, Kawakami K, Saltz LB, Danenberg PV, Laird PW. CpG island hypermethylation in human colorectal tumors is not associated with DNA methyltransferase overexpression. Cancer Res. 1999;59:2302-2306. [PubMed] |
| 19. | Oue N, Kuraoka K, Kuniyasu H, Yokozaki H, Wakikawa A, Matsusaki K, Yasui W. DNA methylation status of hMLH1, p16(INK4a), and CDH1 is not associated with mRNA expression levels of DNA methyltransferase and DNA demethylase in gastric carcinomas. Oncol Rep. 2001;8:1085-1089. [PubMed] |
| 20. | Fatemi M, Hermann A, Gowher H, Jeltsch A. Dnmt3a and Dnmt1 functionally cooperate during de novo methylation of DNA. Eur J Biochem. 2002;269:4981-4984. [PubMed] [DOI] |
| 21. | Hsieh CL. The de novo methylation activity of Dnmt3a is distinctly different than that of Dnmt1. BMC Biochem. 2005;6:6. [PubMed] [DOI] |
| 22. | Gowher H, Stockdale CJ, Goyal R, Ferreira H, Owen-Hughes T, Jeltsch A. De novo methylation of nucleosomal DNA by the mammalian Dnmt1 and Dnmt3A DNA methyltransferases. Biochemistry. 2005;44:9899-9904. [PubMed] [DOI] |
| 23. | Araujo FD, Croteau S, Slack AD, Milutinovic S, Bigey P, Price GB, Zannis-Hadjopoulos M, Szyf M. The DNMT1 target recognition domain resides in the N terminus. J Biol Chem. 2001;276:6930-6936. [PubMed] [DOI] |
| 24. | Fatemi M, Hermann A, Pradhan S, Jeltsch A. The activity of the murine DNA methyltransferase Dnmt1 is controlled by interaction of the catalytic domain with the N-terminal part of the enzyme leading to an allosteric activation of the enzyme after binding to methylated DNA. J Mol Biol. 2001;309:1189-1199. [PubMed] [DOI] |
| 25. | Rhee I, Jair KW, Yen RW, Lengauer C, Herman JG, Kinzler KW, Vogelstein B, Baylin SB, Schuebel KE. CpG methylation is maintained in human cancer cells lacking DNMT1. Nature. 2000;404:1003-1007. [PubMed] [DOI] |
| 26. | Rhee I, Bachman KE, Park BH, Jair KW, Yen RW, Schuebel KE, Cui H, Feinberg AP, Lengauer C, Kinzler KW. DNMT1 and DNMT3b cooperate to silence genes in human cancer cells. Nature. 2002;416:552-556. [PubMed] [DOI] |
| 27. | Leu YW, Rahmatpanah F, Shi H, Wei SH, Liu JC, Yan PS, Huang TH. Double RNA interference of DNMT3b and DNMT1 enhances DNA demethylation and gene reactivation. Cancer Res. 2003;63:6110-6115. [PubMed] |
| 28. | Ting AH, Jair KW, Suzuki H, Yen RW, Baylin SB, Schuebel KE. CpG island hypermethylation is maintained in human colorectal cancer cells after RNAi-mediated depletion of DNMT1. Nat Genet. 2004;36:582-584. [PubMed] [DOI] |
| 29. | Geiman TM, Sankpal UT, Robertson AK, Chen Y, Mazumdar M, Heale JT, Schmiesing JA, Kim W, Yokomori K, Zhao Y. Isolation and characterization of a novel DNA methyltransferase complex linking DNMT3B with components of the mitotic chromosome condensation machinery. Nucleic Acids Res. 2004;32:2716-2729. [PubMed] [DOI] |
| 30. | Kang MY, Lee BB, Kim YH, Chang DK, Kyu Park S, Chun HK, Song SY, Park J, Kim DH. Association of the SUV39H1 histone methyltransferase with the DNA methyltransferase 1 at mRNA expression level in primary colorectal cancer. Int J Cancer. 2007;121:2192-2197. [PubMed] [DOI] |
| 31. | Li H, Rauch T, Chen ZX, Szabó PE, Riggs AD, Pfeifer GP. The histone methyltransferase SETDB1 and the DNA methyltransferase DNMT3A interact directly and localize to promoters silenced in cancer cells. J Biol Chem. 2006;281:19489-19500. [PubMed] [DOI] |
| 32. | Espada J, Ballestar E, Fraga MF, Villar-Garea A, Juarranz A, Stockert JC, Robertson KD, Fuks F, Esteller M. Human DNA methyltransferase 1 is required for maintenance of the histone H3 modification pattern. J Biol Chem. 2004;279:37175-37184. [PubMed] [DOI] |