修回日期: 2008-10-17
接受日期: 2008-11-03
在线出版日期: 2008-11-28
目的: 检测HBV pre-X在真核表达载体pc DNA3.1-myc-his -HBV pre-X转染的HepG2细胞中的表达,并筛选其中的代谢相关差异表达基因.
方法: 将构建的真核表达载体pcDNA3.1-myc-his-HBV pre-X转染HepG2细胞后蛋白免疫印迹检测; 将pcDNA3.1-myc-his-HBV pre-X和pcDNA3.1-myc-his载体分别转染HepG2细胞后, 提取mRNA后逆转录为cDNA, 运用基因表达谱芯片技术分析差异表达基因.
结果: 构建的真核表达载体经ApaⅠ、BstⅪ双酶切鉴定, 转染HepG2细胞后HBV pre-X表达经蛋白免疫印迹证实; 经基因表达谱芯片分析发现, 其中基因表达水平显著上调和下调的分别是200个和62个.
结论: 筛选HBV pre-X转染HepG2细胞后的代谢相关差异表达基因, 从而为乙型肝炎病毒合并糖尿病、脂肪肝等代谢性疾病的分子生物学机制的研究提供了重要依据.
引文著录: 柴艳云, 张锦前, 赵龙凤, 王琪, 成军. HBV pre-X转染HepG2细胞后差异表达基因的筛选. 世界华人消化杂志 2008; 16(33): 3724-3728
Revised: October 17, 2008
Accepted: November 3, 2008
Published online: November 28, 2008
AIM: To detect the expression of HBV pre-X and screen the different gene expression in HepG2 cells transfected with HBV pre-X using gene chip technology.
METHODS: The expressive vector of pcDNA3.1-myc-his-HBV pre-X was transfected into HepG2 cell line. The expression of HBV pre-X protein was observed by western-blotting method. cDNA microarray technique was applied to detect the differentially expressed genes in HepG2 transfected either with pcDNA3.1-myc-his-HBV pre-X or with pcDNA3.1-myc-his.
RESULTS: The expressive vector was confirmed by restriction enzyme digestion. The expression of HBV pre-X protein was confirmed by western-blotting method. High quality mRNA and cDNA was prepared and successful microarray screening was conducted. Based on the scanning results, 200 genes were found to be up-regulated and 62 genes down-regulated in HepG2 cell line transfected with HBV pre-X.
CONCLUSION: cDNA microarray technology is successfully used to screen the genes differentially expressed in HepG2 cell line transfected with HBV pre-X, which may bring some new clues for studying the molecular biology mechanism of hepatitis B virus with metabolic diseases.
- Citation: Chai YY, Zhang JQ, Zhao LF, Wang Q, Cheng J. Screening of genes differentially expressed in HepG2 cells transfected with HBV pre-X. Shijie Huaren Xiaohua Zazhi 2008; 16(33): 3724-3728
- URL: https://www.wjgnet.com/1009-3079/full/v16/i33/3724.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i33.3724
HBV基因组划分出4个开放读码框架(ORF), 分别命名为S、C、P、X区[1-2]. 4个ORF中表达的氨基酸长度不同, 在X基因之前存在一个ORF, 长度168 bp, 编码56个氨基酸残基(aa), 命名其为前-X(pre-X)区[3]. 前-X与X基因之间没有终止密码子, 前-X区编码氨基酸与X蛋白融合表达, 序列为MGLGYWPSPPAWNLCGSSADPYCGTPSSLFCSQPVWSETYRNRQLCCPLSQIHLLS[4]. 将前-X+X基因称为全X, 全长约630 bp, 编码210个氨基酸. 前-X区是一种HBV血清型特异性编码区, 该编码序列仅在adr血清型中有编码表现[4]. 前-X基因的编码具有一定的HBV C基因型倾向性, 但并不具有基因型特异性[5]. 在HBV基因组中发现前-前-S和前-X基因序列的存在改写了HBV DNA编码基因序列研究的历史[6]. 本研究应用基因芯片技术, 筛选HBV pre-X对HepG2细胞差异表达的代谢相关基因, 为进一步深入研究HBV合并糖尿病、脂肪肝等代谢性疾病的生物学机制提供重要依据.
真核表达质粒pcDNA3.1-myc-his-HBV pre-X、HepG2细胞系本实验室保存; 细胞培养相关试剂及总RNA提取试剂TRIzol均购自Gibco公司; 小鼠抗人抗His标签单抗, 购自Simga公司. 真核表达载体pcDNA3.1-myc-his-HBV pre-X系本实验室保存.
1.2.1 HepG2细胞的转染和筛选: 细胞株用DMEM培养基(含有100 kU/L氨苄青霉素的100 mL/L小牛血清)常规培养, 待生长到50%-70%融合时, 采用Lipofectin脂质体法(转染程序参照Manitsans方法)将pcDNA3.1-myc-his及pcDNA3.1-myc-his-HBV pre-X质粒转染细胞, 含G418的培养基继续培养48 h, 裂解细胞后留取上清, 用于表达产物Western blot检测.
1.2.2 HBV pre-X蛋白Western blot检测: 将上清于SDS-PAGE胶电泳, 于50 g/L的脱脂奶粉室温封闭2 h, 加入TBST稀释过的抗体(小鼠抗人抗His标签抗体, 1:500稀释), 4 ℃孵育1 h, 用TBST液漂洗4-6次, 每次5 min, 再用TBST稀释的酶标抗体(山羊抗鼠IgG-HRP, 1:5000稀释)于30 ℃温育60 min, TBST漂洗4-5次后滤纸吸干残余液体, 加入ECL化学发光底物, 在X光片曝光, 显影、定影后可见结果.
1.2.3 总RNA提取: 在35 mm培养皿中常规培养HepG2细胞, 细胞生长至对数期时分别将pcDNA3.1-myc-his及pcDNA3.1-myc-his-HBV pre-X转染HepG2细胞48 h后收获细胞. 使用TRIzol试剂一步法提取细胞总RNA(对照组和实验组), 样品经分光光度计检测吸光度A值, 并行琼脂糖凝胶电泳检测.
1.2.4 mRNA纯化, 探针标记和芯片制备: 以Qiagen公司Oligotex mRNA Midi Kit纯化得mRNA, 并行电泳检测常规方法逆转录标记cDNA探针并纯化. Cy3-dUTP标记对照组细胞mRNA(5 μg), Cy5-dUTP标记实验组细胞mRNA(5 μg). 乙醇沉淀后溶解于杂交液中. 芯片包含的20000个cDNA由生物芯片上海国家工程研究中心上海生物芯片有限公司提供, 包括原癌基因和抑癌基因、免疫调节相关基因、细胞凋亡和应激反应蛋白相关基因、信号转导相关基因等. 以通用引物进行PCR扩增, 靶基因溶解于3×SSC溶液中, 用Cartesian公司的Cartesian 7500点样仪及TeleChem公司的硅烷化玻片进行点样. 玻片经水合(2 h)、室温干燥(0.5 h)、UV交联, 再分别用2 g/L SDS、水及2 g/L的硼氢化钠溶液处理10 min, 晾干备用.
1.2.5 杂交及洗涤: 将基因芯片和杂交探针在95 ℃水浴变性5 min, 将混合探针加在基因芯片上, 置于60 ℃杂交15-17 h. 以SSC洗涤10 min, 室温晾干.
1.2.6 检测与分析: 用General Scanning公司的ScanArray 3000扫描芯片. 用预先选定的内参照基因对Cy3和Cy5的原始提取信号进行均衡和修正. 用ImaGene3.0软件分析Cy3、Cy5两种荧光信号的强度, 计算Cy5/Cy3比值. 阳性结果判断: Cy5/Cy3>1.8, 红色荧光, 显示表达增强; Cy5/Cy3<0.6, 为绿色荧光, 显示表达减弱.
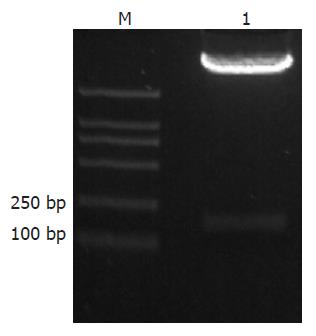
真核表达质粒pcDNA3.1-myc-his-HBV pre-X, 转染前ApaⅠ、BstⅪ双酶切鉴定(图1).
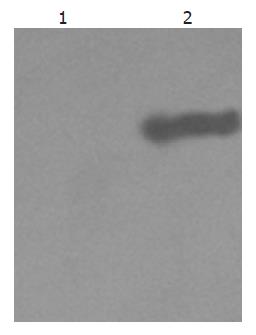
pre-X分别转染HepG2细胞后, 经蛋白免疫印迹检测结果 HepG2细胞株用DMEM培养基培养, 待生长到50%-70%融合时, Lipofectin脂质体法将pcDNA3.1-myc-his及pcDNA3.1-myc-his-HBV pre-X质粒分别转染到HepG2细胞, 培养48 h后裂解细胞后留取上清于SDS-PAGE胶电泳, Western blot检测(图2).
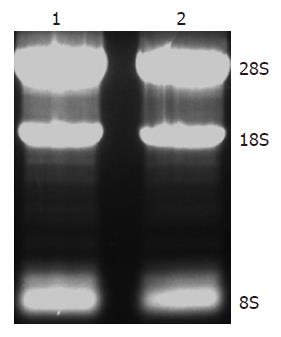
在35 mm培养皿中常规培养HepG2细胞, 细胞生长至对数期时分别将pcDNA3.1-myc-his及pcDNA3.1-myc-his-HBV pre-X转染细胞48 h后收获细胞, 使用TRIzol试剂一步法提取细胞总RNA, 琼脂糖凝胶电泳可见28S、18S及8S条带(图3).
基因表达水平显著上调和下调的分别是200个和62个, 摘取代谢相关重要基因见表1-2.
| 序号 | GenBank | 编码蛋白 | Cy5/Cy3 |
| 1 | NM_000209.1 | Homo sapiens insulin promoter factor 1(IPF1) | 2.045 |
| 2 | BQ549626 | Human insulinoma Homo sapiens cDNA clone | 2.019 |
| 3 | NM_018690 | Homo sapiens apolipoprotein B48 receptor(apoB48R) | 2.693 |
| 4 | NM_005108.2 | Homo sapiens xylulokinase homolog(H.influenzae)(xylB) | 2.043 |
| 序号 | GenBank | 编码蛋白 | Cy5/Cy3 |
| 1 | NM_001506.1 | Homo sapiens G protein-coupled receptor 32(GPR32) | 0.318 |
| 2 | NM_007193.3 | Homo sapiens annexin A10(ANXA10) | 0.408 |
| 3 | NM_000860.3 | Homo sapiens hydroxyprostaglandin dehydrogenase 15-(NAD)(HPGD) | 0.489 |
大量的统计学资料表明: HBV感染人体后可导致患者出现胰岛素抵抗、糖耐量降低、糖尿病、脂肪肝等代谢性疾病, 这提示HBV与糖尿病、脂肪肝等代谢性疾病的发生、发展密切相关[7-9]. Custro et al[10]报道, 成人慢性乙型肝炎2型糖尿病的发病率为25%, 是普通人群发病率的4倍. 大量临床观察的病例资料提示2型糖尿病患者HBV感染率超过普通人群, 而慢性乙型肝炎患者2型糖尿病发病率也显著高于普通人群, 表明HBV与糖尿病的发生有更强的相关性, 但HBV导致糖尿病等代谢性疾病发生、发展的分子生物学机制尚不明确. 研究表明: 乙肝病毒前-X基因的存在与HBV病毒株的突变有关, 这种突变在严重的肝脏疾病患者中可以检测到. 同时, 前-X基因也存在不能编码的情况, 在HBV C型基因型中不能编码前-X区的原因主要是A2608→C/T替换突变或C/A2733→T替换突变单一突变; 而B型、B/C型不能编码前-X区蛋白, 主要因为双突变的存在[11-12]. 在已知基因型的HBV DNA阳性的HCC患者体内普遍表达前-X基因, 同时, 经过比较, 前-X基因在HCC患者中表达率明显高于慢性乙型肝炎患者, 具有显著性差异, 说明在疾病的不同阶段, 其前-X基因表达不相同, 提示前-X基因可能与HCC的发生密切相关[13-15]. 进一步研究证实: 前-X基因的编码在HCC患者中具有普遍性, 其表达与患者的性别、年龄、乙肝病毒复制指标(HBV DNA、HBeAg阳性率)、ALT水平、AFP阳性率等临床相关指标无明显关系[16]. 酵母双杂交系统研究表明: 乙肝病毒全X基因编码的蛋白质与前-X基因编码的蛋白质存在相互作用, 这为全X蛋白生物学功能的研究提供了新的思路[17]. 因此, 进一步研究HBV pre-X转染HepG2细胞后的差异表达基因, 对深入研究相关的作用机制具有十分重要的意义. 本研究应用表达谱芯片技术筛选HBV pre-X转染HepG2细胞中的差异表达基因, 并从20 000个基因中筛选出262个差异表达的基因, 其中基因表达水平显著上调和下调的分别是200个和62个, 这些基因涉及细胞信号转导、物质代谢、肿瘤发生、细胞凋亡等多个领域, 这与HBV pre-X的生物学功能密切相关.
膜联蛋白是一类重要的钙结合蛋白, 参与各种重要的生物学过程. 膜联蛋白A10(ANX10)是此类蛋白中表达量很少的功能未知的新成员. 在成人肝脏和原发性肝癌组织中可以检测到ANX10的表达. ANX10的表达下调与原发性肝癌(HCC)的发生、发展及预后不良等关系密切, 这可能为原发性肝癌的基因治疗提供了一条潜在的思路[18]. 我们研究发现: HBV pre-X在HepG2细胞中可以下调ANX10的表达, 从而促进HCC的发生、发展, 加重肝细胞损害. 有研究表明: apoB48R与动脉粥样硬化、高胆固醇血症等心血管疾病的发生密切相关, 其机制可能与apoB48R基因核苷酸序列改变有关[19-21]. 我们研究发现HBV pre-X在HepG2细胞中上调ApoB48受体, 提示HBV pre-X可能参与载脂蛋白的表达调控, 这可能与脂类代谢和脂肪肝密切相关. 我们研究也发现HBV pre-X在HepG2细胞中上调胰岛素促进因子1(IPF1)、木酮糖激酶(xylB)及胰岛素瘤基因的表达, 说明HBV pre-X可能参与了肝细胞对糖类代谢的调节. 同时, 本研究也发现HBV pre-X在HepG2细胞中可以下调G蛋白耦联受体32(GPR32)(与胰岛素等受体传导通路有关)及羟基前列腺素脱氢酶15(HPGD)的表达, 其表达下调可能与胰岛素抵抗、糖尿病等代谢性疾病的形成有关, 这也说明HBV pre-X可能参与了肝细胞对糖类代谢的调节. 以上结果为相关研究提示了大量的研究方向和思路, 我们拟进一步深入研究确定.
由此, 可以推论: HBVpre-X转染肝细胞后通过相应的信号转导通路可以导致某些重要功能基因表达水平的改变(上调或下调), 导致基因编码的蛋白功能的改变, 促进一系列代谢性疾病的发生, 如: 胰岛素瘤, 糖尿病, 脂肪肝等. 但HBV pre-X相关生物学过程及其信号转导机制目前仍尚未明确, 有待深入研究.
总之, 我们筛选了HBV pre-X转染HepG2细胞的差异表达基因后发现, 参与多种生物学过程的多种基因都存在明显的表达差异, 其主要与细胞信号转导、细胞增殖与分化、能量代谢、细胞凋亡和肿瘤发生等密切相关. 本实验结果为进一步研究HBV pre-X在肝细胞中相关生物学过程及其信号转导机制提供了坚实的基础, 尤其在HBV pre-X与糖、脂类代谢相关性方面则更具重要意义.
HBV慢性感染可引起胰岛素抵抗、糖耐量降低, 甚至并发糖尿病、脂肪肝等代谢性疾病, 提示HBV和糖类、脂类物质代谢密切相关, 但其分子生物学机制尚不明确. 近年来, HBV新的ORF即pre-X的发现引人注目, 但相关的深入研究甚少.
刘杞, 教授, 重庆医科大学病毒性肝炎研究所
关于HBV pre-X基因生物学功能的相关机制研究热点逐渐深入并涉及到肿瘤发生、细胞凋亡、细胞信号转导、物质代谢等领域, 而后两方面的研究则更为引人注目, 但这些作用机制仍不明确.
有关HBV pre-X的研究发现, 在乙型肝炎后肝细胞癌患者中其表达量明显高于慢性乙型肝炎患者, 提示pre-X基因可能与肝细胞癌密切相关; 通过酵母双杂交技术可知乙肝病毒全X基因编码的蛋白质与前-X基因编码的蛋白质存在相互作用.
本研究应用基因芯片技术筛选HBV pre-X对HepG2细胞差异表达的基因, 发现了与多种生物学功能有关的多种基因都存在明显的表达差异, 其中部分在国内外相关的研究工作中未见报道, 尤其与物质代谢和信号转导相关的基因更加具有研究意义.
本文的相关工作为HBV pre-X的生物学功能研究及其参与物质代谢、肿瘤发生等的相关机制研究提供了实验依据和方向, 极具研究价值和前景, 并为相关疾病的预防、治疗提供理论依据.
本研究选题明确, 设计思路清晰合理, 实验方法简单可靠, 可重复性强, 具有一定理论和科学价值.
编辑: 李军亮 电编: 何基才
| 1. | Galibert F, Mandart E, Fitoussi F, Tiollais P, Charnay P. Nucleotide sequence of the hepatitis B virus genome (subtype ayw) cloned in E. coli. Nature. 1979;281:646-650. [PubMed] [DOI] |
| 2. | Ganem D, Varmus HE. The molecular biology of the hepatitis B viruses. Annu Rev Biochem. 1987;56:651-693. [PubMed] [DOI] |
| 3. | 杨 倩, 董 菁, 成 军, 刘 妍, 洪 源, 王 建军, 王 琳, 张 树林. 乙型肝炎病毒基因组中前-X-编码基因启动子序列的确定及转录活性的鉴定. 解放军医学杂志. 2003;28:763-765. |
| 7. | Demir M, Serin E, Göktürk S, Ozturk NA, Kulaksizoglu S, Ylmaz U. The prevalence of occult hepatitis B virus infection in type 2 diabetes mellitus patients. Eur J Gastroenterol Hepatol. 2008;20:668-673. [PubMed] |
| 8. | Shi JP, Fan JG, Wu R, Gao XQ, Zhang L, Wang H. [Prevalence and risk factors of hepatic steatosis in patients with chronic hepatitis B]. Zhonghua Ganzangbing Zazhi. 2008;16:519-523. [PubMed] |
| 9. | Wang CC, Hsu CS, Liu CJ, Kao JH, Chen DS. Association of chronic hepatitis B virus infection with insulin resistance and hepatic steatosis. J Gastroenterol Hepatol. 2008;23:779-782. [PubMed] [DOI] |
| 10. | Custro N, Carroccio A, Ganci A, Scafidi V, Campagna P, Di Prima L, Montalto G. Glycemic homeostasis in chronic viral hepatitis and liver cirrhosis. Diabetes Metab. 2001;27:476-481. [PubMed] |
| 11. | Faure E. Alternative peptide-fusion proteins generated by out-of-frame mutations, just upstream ORFs or elongations in mutants of human hepatitis B viruses. Virus Res. 2006;117:185-201. [PubMed] [DOI] |
| 14. | Takahashi K, Akahane Y, Hino K, Ohta Y, Mishiro S. Hepatitis B virus genomic sequence in the circulation of hepatocellular carcinoma patients: comparative analysis of 40 full-length isolates. Arch Virol. 1998;143:2313-2326. [PubMed] [DOI] |
| 15. | Tang H, Oishi N, Kaneko S, Murakami S. Molecular functions and biological roles of hepatitis B virus x protein. Cancer Sci. 2006;97:977-983. [PubMed] [DOI] |
| 17. | Yang Q, Cheng J, Dong J, Zhang J, Zhang SL. Molecular epidemiological study on pre-X region of hepatitis B virus and identification of hepatocyte proteins interacting with whole-X protein by yeast two-hybrid. World J Gastroenterol. 2005;11:3473-3478. [PubMed] |
| 18. | Liu SH, Lin CY, Peng SY, Jeng YM, Pan HW, Lai PL, Liu CL, Hsu HC. Down-regulation of annexin A10 in hepatocellular carcinoma is associated with vascular invasion, early recurrence, and poor prognosis in synergy with p53 mutation. Am J Pathol. 2002;160:1831-1837. [PubMed] |
| 19. | Fujita Y, Ezura Y, Bujo H, Nakajima T, Takahashi K, Kamimura K, Iino Y, Katayama Y, Saito Y, Emi M. Association of nucleotide variations in the apolipoprotein B48 receptor gene (APOB48R) with hypercholesterolemia. J Hum Genet. 2005;50:203-209. [PubMed] [DOI] |
| 20. | Kawakami A, Tani M, Chiba T, Yui K, Shinozaki S, Nakajima K, Tanaka A, Shimokado K, Yoshida M. Pitavastatin inhibits remnant lipoprotein-induced macrophage foam cell formation through ApoB48 receptor-dependent mechanism. Arterioscler Thromb Vasc Biol. 2005;25:424-429. [PubMed] [DOI] |
| 21. | Brown ML, Ramprasad MP, Umeda PK, Tanaka A, Kobayashi Y, Watanabe T, Shimoyamada H, Kuo WL, Li R, Song R. A macrophage receptor for apolipoprotein B48: cloning, expression, and atherosclerosis. Proc Natl Acad Sci USA. 2000;97:7488-7493. [PubMed] [DOI] |