修回日期: 2008-01-08
接受日期: 2008-01-15
在线出版日期: 2008-01-28
目的: 克隆健脾益气法明显下调基因中EST片段cDNA的完整表达序列.
方法: 从689个健脾益气法明显下调的基因中, 采用电子克隆与PCR相结合的方法, 克隆出有关EST片段的cDNA完整表达序列.
结果: 成功筛选出11个EST片段的cDNA序列(G2、G4、G5、G6、G11、G14、G15、G16、G17、G18、G20), 经进一步BLAST对比分析, 证明G14、G15、G20 3个基因为新基因, 并进行GenBank登录, 登录号分别为DQ480745、DQ480746、DQ480747.
结论: 本研究成功克隆出3个中医健脾益气法能明显调控的新基因cDNA序列. 这为今后进一步深入观察这些基因的功能以及探讨不同中医治法的作用机制提供了良好的研究基础.
引文著录: 管冬元, 方肇勤, 朱希, 吴中华, 张辉. 克隆与健脾益气法有关的大鼠肝癌新基因. 世界华人消化杂志 2008; 16(3): 265-271
Revised: January 8, 2008
Accepted: January 15, 2008
Published online: January 28, 2008
AIM: To clone the integrated cDNA expression sequence of EST segments in down-regulated genes by invigorating the spleen to supplement Qi.
METHODS: Integrated cDNA expression sequences of EST gene segments were cloned for the 689 down-regulated genes by invigorating the spleen to supplement Qi, with electron cloning in combination with PCR.
RESULTS: cDNA expression sequences were detected in 11 EST segments (G2, G4, G5, G6, G11, G14, G15, G16, G17, G18, G20). BLAST analysis showed that G14, G15 and G20 were novel genes which were submitted to GenBank (their accession number is DQ480745, DQ480746 and DQ480747, respectively).
CONCLUSION: Invigorating the spleen to supplement Qi can clone the cDNA expression sequences of EST segments in the down-regulated genes. Further study is needed to observe the functions of these novel genes and the mechanism of action of different TCM therapies.
- Citation: Guan DY, Fang ZQ, Zhu X, Wu ZH, Zhang H. Cloning of novel hepatoma gene in rats by invigorating the spleen to supplement Qi. Shijie Huaren Xiaohua Zazhi 2008; 16(3): 265-271
- URL: https://www.wjgnet.com/1009-3079/full/v16/i3/265.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i3.265
原发性肝癌(primary liver cancer, PLC)是我国高发的恶性肿瘤之一, 据统计, 每年约有13万人死于肝癌, 占全世界肝癌年死亡人数的43.7%. 中医药辨证论治在防治肝癌方面疗效肯定, 并积累了丰富的临床经验, 其治则治法主要集中在健脾益气、清热解毒、活血化瘀等治法. 大量的研究表明, 脾虚是肝癌的重要病理基础, 脾虚证是肝癌发生发展中的常见证型, 健脾益气法是防治肝癌的常用有效治法[1-2].
我们观察并报道了健脾益气、清热解毒、活血化瘀等治法方药对肝癌大鼠的治疗作用, 发现与模型组和西药对照组相比, 这些治法均能不同程度地延长二乙基亚硝胺诱发肝癌大鼠生存期. 同时, 不同中医治法在肝癌中的作用、生存期延长等方面作用不尽相同, 其中健脾益气法对肝癌大鼠肝功能的改善最明显[3]. 进一步采用美国Affymetrix公司Rat 230A GeneChip检测, 研究表明: (1)西药组表达明显小于模型组, 以及各中药治疗组的基因计476个; (2)全方组表达明显小于模型组, 以及各中西药治疗组的基因计342个; (3)清热组表达明显小于模型组, 以及各中西药治疗组的基因计140个; (4)活血组表达明显小于模型组, 以及各中西药的基因计186个; (5)健脾组表达明显小于模型组, 以及各中西药治疗组的基因计689个[4].
为了进一步揭示健脾益气法防治原发性肝癌的分子机制, 本课题在基因芯片检测的基础上, 应用表达序列标签(expressed sequence tag, EST)电子克隆与PCR技术相结合的办法, 从689个健脾益气法明显下调的基因中克隆出部分(20个待筛选基因)EST序列的cDNA全长序列, 为进一步研究这些新基因的功能打下坚实的基础.
♂Wistar大鼠1只, 体质量305 g, TRIzol试剂盒(Invitrogen公司), Mu-MLV反转录酶(Gibco BRC公司), Ex Taq酶(宝生物工程有限公司), 待克隆的新基因共20个, 编号为G1-20的EST片段的gi登录号及芯片检测结果中健脾益气法对该EST片段的调整作用(表1).
| 编号 | GenBank的gi号 | EST长度(bp) | 正常组 | 模型组 | 健脾组 |
| G1 | 5000060 | 458 | 3210 | 5045 | 2983 |
| G2 | 14886250 | 472 | 3 | 3069 | 1222 |
| G3 | 14931989 | 746 | 696 | 2000 | 931 |
| G4 | 13303048 | 697 | 969 | 1991 | 1001 |
| G5 | 8513155 | 496 | 987 | 1705 | 972 |
| G6 | 11381923 | 483 | 447 | 1688 | 959 |
| G7 | 14979816 | 553 | 650 | 1577 | 624 |
| G8 | 14962954 | 404 | 638 | 1425 | 606 |
| G9 | 14926442 | 681 | 783 | 1311 | 767 |
| G10 | 18183378 | 669 | 648 | 1198 | 631 |
| G11 | 13305474 | 476 | 789 | 1197 | 699 |
| G12 | 8508677 | 590 | 391 | 1132 | 630 |
| G13 | 8510803 | 561 | 563 | 1127 | 568 |
| G14 | 8499623 | 639 | 601 | 1093 | 643 |
| G15 | 14930616 | 646 | 218 | 1068 | 574 |
| G16 | 14939126 | 641 | 174 | 1058 | 559 |
| G17 | 14976479 | 548 | 158 | 1035 | 616 |
| G18 | 3709796 | 623 | 958 | 8348 | 4687 |
| G19 | 13893620 | 768 | 2980 | 6422 | 3539 |
| G20 | 4132294 | 410 | 515 | 2589 | 1525 |
1.2.1 EST的cDNA全序列电子克隆的方法: (1)将EST序列进行BLAST比对, 检索有无相同已知基因的序列登录. (2)在NCBI网站的UniGene数据库中查找与EST序列有关的序列片段, 并下载到PC机上, 以文本文件保存. (3)在下载的文本文件中各序列单独用一系列的文本文件保存(均为FASTA格式). (4)应用DNAMAN6.0.40生物软件, 拼接上述保存的一系列文本文件中的序列. (5)用拼接后的序列与原EST序列进行相似性比较, 如果拼接后的序列包含原EST序列, 并得到一定程度的延伸, 则说明该EST片段的cDNA全长序列已能初步完成, 并能进行下面的校对工作. (6)再将拼接后的序列与大鼠的基因组进行BLAST比对. (7)下裁比对后的基因组序列, 并按次序连接好, 从而生成EST的电子延伸序列. (8)最后, 通过Primer5生物软件, 将上述生成EST的电子延伸序列反向互补, 得到该EST片段的cDNA全长序列.
1.2.2 PCR扩增的引物设计: 通过EST的电子克隆方法得到各EST片段的cDNA全长序列后, 用Primer3在线生物软件进行PCR引物设计.
1.2.3 大鼠肝组织总RNA的提取: 大鼠处死后取新鲜肝组织50 mg, 再按照Invitrogen公司的TRIzol试剂盒说明进行肝组织总RNA提取.
1.2.4 反转录: RT反应体系中含: DEPC处理水7 mL, dNTP(each 10 mmol/L)2 mL, 5×Buffer 4 mL, DTT(100 mmol/L)1 mL, RNAsin(40000 kU/L)1 mL, Olig(dT15)(100 mg/L)1 mL总RNA(1 g/L)2 mL. 混匀, 65 ℃ 5 min; 冰上加入RNAsin、M-MuLV各1 mL, 混匀, 37 ℃ 60 min后, 再加M-MuLV 1 mL, 混匀, 37 ℃ 60 min; 最后94 ℃ 5 min灭活逆转录酶; -20 ℃保存.
1.2.5 扩增PCR: PCR反应体系中含: 水22 mL, dNTP(each 2.5 mmol/L)2 mL, 10×Buffer 4 mL, 上游引物(5 mmol/L)4 mL, 下游引物(5 mmol/L)4 mL, RT产物4 mL, Ex Taq酶0.4 mL. 混匀后, 进行扩增. PCR反应条件为: 94 ℃ 5 min→94 ℃ 1 min, 55 ℃ 1 min, 72 ℃ 4 min, 35个循环→72 ℃ 5 min→4 ℃保存.
1.2.6 电泳、回收与测序: PCR产物经琼脂糖电泳后, 对相应大小的产物进行回收并送公司测序.
1.2.7 测序后基因全长cDNA序列的蛋白阅读框架分析(ORF): 将测序后基因全长cDNA序列复制到NCBI的ORF系统中进行自动蛋白阅读框架分析, 以+1的氨基酸序列作为该基因的蛋白表达序列.
1.2.8 测序后基因全长cDNA序列的比对与登录: 测序后的基因全长cDNA序列与NCBI的BLAST系统进行核酸的非冗余数据库(nr)比对, 如果为新基因, 则进行基因库登录.
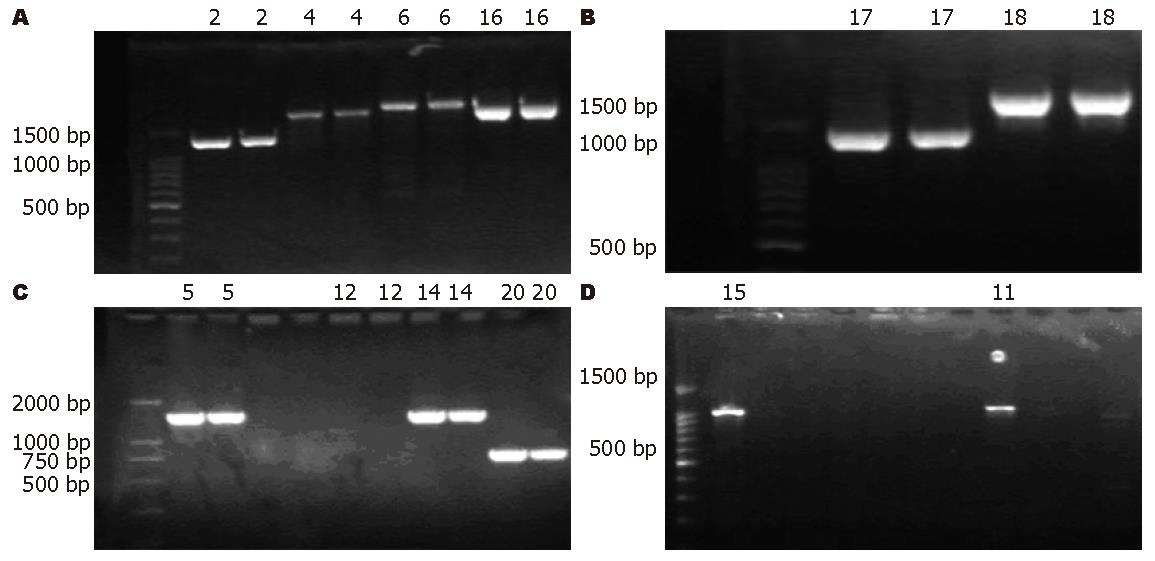
在候选的20个EST中, G1、G3、G9、G13、G19等5个EST未得到有效的电子延伸序列.
在延伸序列的15个未知基因中, G2、G4、G5、G6、G11、G12、G14、G15、G16、G17、G18、G20等12个基因得到了相应的扩增产物(图1), 并测序验证了序列的准确性, 但其中G12反复测序均未测出相应的序列, 测序公司认为可能是引物不适宜测序或PCR回收产物浓度不够或不纯.
经PCR及测序验证的G2、G4、G5、G6、G11、G14、G15、G16、G17、G18、G20等11个基因序列, 与NCBI的BLAST系统进行核酸的非冗余数据库(nr)比对, 结果表明, G2、G4、G5、G11、G18等5个基因已于2005年底至2006年初被其他的研究机构登录, 而G6、G16、G17等3个基因的序列还有相当一部分未得到扩增与测序的验证. 于是, 将G14、G15、G20等3个新基因进行GenBank登录, 录号分别为DQ480745、DQ480746、DQ480747. 3个新基因的核酸序列如下(各基因的序列中, 斜体的部分为相应原EST序列颠倒反相后的序列, 其余为新延伸的部分). G14(1537 bp): GTCTCCCTAAGTGCTGGGGTCACAGACATGTGCCACTGCCCCAGCATCCATGAATCTTCTTGAAGATCCATTTCTCCCTTTATCTTCCCTAACCTTTTCATTTTTCTACTTTGTGTGTGTGTGGTGATGGGGATCAGACGTCGGCCCTCACATGCTGGGCGAGTGTTCTACCACTGAACCACACCCTCCAGCCCCTTCCTTGTCTTTCCTCTTCCCCTTTCTTATTGCATTTCTACTATAGAATGATCTCCCTGATACATCCTGGGCCATAAGGAAATGTAGGACTCTGGGGTTTACTAAACATGGTGGGCAGTCCAAAGCTGTGCCCTATTTCCCCATTGTGAAAATATGGCAGTTCCTTTGATTACCTGGCTTAGGCCAGAGTACCATTGCAGGAGAGGCCTCAGGGCCTGCACTCGGGATGAGGAAGGTGGGGTATATTCAGCATTGGGTTTCTGAGAGGAGAGCTGATCTGAGCAGTTACCATGAACCCTGAGGCAGGGTTCCCAGAGTTGTCCTCTCTGCTCCGGTGAGGCAGCAACTGAGGGTCTCATGTCTCCAGTCTTGTTCACCTCAAGCCATCTACATTCTCTCTTTCTGCCATACTGTCATTGCTGCTAATGGTCAGAGTCAAGCATGTGGCATTACAGAATGAGCAGGTGGTCTTGGGCTGCTATCCGGACATCACTTTTTAAATGTGGAAACATGCAGATGACTTCTCCAAGAGGCTTAGGCTAATGTGTCAGCCAGAAACAGTAGGCTAAGAGAAGCTTGAACTCCAGAAGAAAGCTGTTGACATTCTGTGTGACAGCTGGAAAATGTATAAGCACCCGCCCTTGAGACAACTCAGTGAAGCCGAACTGTGCTATCCGCCATTTGATAGAAATGTTCTCACCTTGGGCTGACTGTGATGTACAATGTGTTATGAAAATTGTCGAGCTGAAGCTTTAGTCAAATACGAGTCACTTGCTTTGATCCCGTGTATTTTATGATTCCTCTGTCAGTGACTTTCCCTCGCCTTACCCTGAGTGTGAATTGTAGCATGATTGTATAAACCTCTATGTAGAAAATGGAGATTTCATGTTCTACAAAATGTTAAGCTCTAACTGATCCATTTCTGTGTTCTCTATAGCCTCATGTGTATGACCTCACTTTCTTATCTATTTAAACTTTCCTTCTGTTCTAAAGCTTTCGTAGCACCCCTGTTCAGGTGGAGAGGAAGCTGTCTCTTTGCAGAACCGTACTGTAACCATTTCTCCTTTATAAATATTATTTTCACAGGGCTGATTGTATACAGGGCTTGTAATAAAATTTTAACACTGTGTTGTGAATGAGCTGCGGTTTCTCCGCTGGAGGTGACTTCAAAAGCACCTGAGGGTGGAGCGTTCTTTCTGTGACCTTACTTCCTAGTACGTGAAGTCTAATCTCACAGTCTTGTTCTGTTTCAGTCTCCTGTATAAGAAGTACCATATTTTCTGCCCATCATACTTTGTAATAAAACTTGAACATGTATAGATTGAAAAAAAAAAAAAAAAAA. G15(2171 bp): GGCCTGGAAAAGGTGTTCCCAGGGATAATACACTGTAAACCCCACAAGCCTTCTGGATGGGTGACAGCTGCAAGCTCGTCTCTGTGGTCCTTAGGGCTCTCCTTGGCTTTTACTCTACAAACCACCCAGGCAAACTCATCTGTTCATGAAATAACTCCAGAGAAAGAAAACCAACAAAGAAAAGGCAGAAAGGATCTATAAACAGCGCTGCCAGCTTCCTGTTTCCACTGGCATCCATTGTAATTTAATCAGGCTAGTTCAGCCCTCATGGACAAAACAACAGTTAGCACACAGATGAAACAAGCTTTAAAAACTTCTCAAATTCTGTCTAGCCAAACCATGCCGCCTCTCCTCTTAGACTGGCTCCAGTTCCTTAGAAATGCCAACTTGCCATTGGTACCTCCTACCACCAGGCTAGCACAGAGCTCTCCCTGCTTGCCTAACATCTTAATCAGCCCTTGCTCCCTGGGGCTGCTGTGGAAGTGGAGACTAAACCTCCACTTCCCTAACTACCTTTTATGTACACACACAACTCTCTTACCTTCTCAAACCAGGAGCATGACTGAGTCAAATCAGTCTGTCCCCCTAATGCCAACAGGATGAAGGTTCCCTACGTCCTGGCCTCTCATCTCTGTGCCTCTGCTCAGCCAAGGTCTTCCTGCCCTGATGTTCATGTCCCTCACATTAGGCTTGGGTAGGGTGGTTCACCAGGATGCCATGCCCACACTATGCCGGGACAGGCCACACTAAAGCCCTGATATGTCCACCTGTACGGAGAGTGATTATGTAAAGCTTCACAGCACTGAGGCACAAAGCGCTGCCTCACAGTGAATGAAGCAAGTACAGGGGGTGCTCGAAGCCTCCCTCCTAGTGGTGTCTGGACCAAGATCTGATTTCTCTGACTGGACTCCCCACCATCCCTCTGAGGGTCATGGAGTAGAAGAGTCATTTGACACTCACCTCAAATGATCCAGGTACTAACTTCTACAGAAGTTGCTGGCGCTGGTGCGTCTTGCTGGCACGTGGAGTGGCAGCCGAACCTCGCGGGGCGGGAAGCCGCGAGATGGACACCCCTCCGCTCTCAGACTCGGACTCCGGGTCGGATGAGTGCCTGGCCTCAGATCAAGAGTTGCAGGATGCGTTTTCCCGCGGACTCCTAAAGCCAGGCCTCAATGTCGTGCTAGAGAAGCCGAAGAAGGCGGTGAATGACGAGAATGGCCTGAAGCAGTGCTTGGCTGAATTCAAACGGGATCTGGAGTGGGTTGAAAGGCTCGATGTGACCCTGGGTCCTGTGCCTGAAGCCAGTGAAACTCAGTCAACACCCCAGAACAAGGACCAGAAGAAAGGTGTTAATCCAGAAGACGACTTCCAGAGGGAAATGAGTTTCTACCGCCAGGCCCAGGCTGCTGTGCTTGCAGTATTACCCCGACTCCACCAGCTCCAAGTCCCTACGAAGAGGCCCACTGATTATTTTGCAGAAATGGCCAAGTCTGATCAACAGATGCAAAAGATTCGACAGAAGCTGCAGACTAAACAGGCTGCCATGGAGAAATCTGAAAAGGCCAAGCAACTTCGAGCGCTTAGGAAATACGGAAAGAAGGTGCAAACTGAGGTCCTTCAGAAGAGGCAGCAGGAGAAAGCGCATATGATGAATGCCATCAAGAAGTACCAGAAAGGCTTCTCTGATAAGCTGGATTTCCTTGAGGGCGATCAGAAGCCTGTGGAACGAGGTGCAAAGGCAGGAGGAGCTAAAGGCCAGCAGATAAGTAAAGGGGCCCAATGCCAAACGAAGATACAAAAATCAGAAGTTTGGTTTTGGCGGAAAGAAGAAAGGCTCCAAATGGAACACACGGGAGAGCTACGACGATGTCTCCAGCTTCCGGGGCAAGGTGGCTCATGGCAAGGGCCCCAGAAGGCCTGGGAAGAAAGGGGCAAACAAGAGACCCGGAAAACGTACCAGACAGAAACTGAAAAGCAAAACCCGCTGAACAGCACTGTCCTGTGTAAAGAACCAGGAGAAGGATGCACAGCACACTCAGTGTTCGTCCTGATTTTTGTAGGAAATTATTTTAATAAATTAAACCTATGGAAGAAAGATACTCTTGGGTCAAAACCAGATACTTTTTAAAAGATGGAGAAGTTTGCACATTAAAAAATATTGCTGCTCCTCA. G20(866 bp): CCCACGCGTCCGCATGTGTGTCCCACCCCCAAATGTACAGTGTCCACCTAGTTACCTGAGAGAACGCCCCAGTCAAGTGTTACTAGTCTTTTAGATTTCAGCAGGGAAGCCTCCACCTAATGTGGACCAACTCTGAACTATTCCTGTCCCTAAGGTCAGAGAGGCCATAAATGAACATTTACCTTATATTTTTATTTCATAATGAAATCTTTTAGGCCAAACAACAATGTCTCGCTTCTTAAAGCCACAAAGAGCCAACCCCCCTCCTAGATCCGCTGTGCCAGGACGGATAGTGGGAGGCTGATTCTCAACCTTTACCAGAACAGCAAAATGTCTAAGACACATGTAACGGGCAAAAACAGAATGCTCAGACCAAATGCGTTGTAAAGTGATATGCACTGTTAATTACAGATCTTTGAAAGTCTCTGTGCACATATGTAAGTTTTATTCAACCATAAAATTTATTATCAGTCTTAGGTAAATGAACAGTATTTTTATACCACATAAACCCTGCTTTCCAAAACTCATTTCCCTCTAACTTACCAGGCACATTATACACACAGCTTAAAAAAGATCATATTTTTTTATTCTTGACCATCCACTTATTTTGAAATTTCCAAAGTTTCTTAGAAGCGAATTAAATAGAAACTCATGGGGAGTGTCTTATGGAGACGTCAGAAAATGTCACACCAGAGATCATCCATGCTGTAGCCAAATGCATCTTCTAAGTCTGTCAGTGGTGGGGTGTACCCACCTTCCCTGTGTCTAAAGTATACTTTGCCCACTGACAGTGGGCTCTGGCACCTTGGGATTGCTGTGAACCCAAATCAAAATAAAATCTGAGAAGCAAAAAAAAAAAAAAAAAA.
3个新基因中, 以电子克隆序列为有意义链(mRNA序列), 进行在线ORF分析. 结果其蛋白阅读框架如下: G14: 65aa; 1137-1334 bp, MCMTSLSYLFKLSFCSKAFVAPLFRWRGSCLFAEPYCNHFSFINIIFTGLIVYRACNKILTLCCE. G15: 354aa; 1064-2128 bp, MDTPPLSDSDSGSDECLASDQELQDAFSRGLLKPGLNVVLEKPKKAVNDENGLKQCLAEFKRDLEWVERLDVTLGPVPEASETQSTPQNKDQKKGVNPEDDFQREMSFYRQAQAAVLAVLPRLHQLQVPTKRPTDYFAEMAKSDQQMQKIRQKLQTKQAAMEKSEKAKQLRALRKYGKKVQTEVLQKRQQEKAHMMNAIKKYQKGFSDKLDFLEGDQKPVERGAKAGGAKGQQISKGAQCQTKIQKSEVWFWRKEERLQMEHTGELRRCLQLPGQGGSWQGPQKAWEERGKQETRKTYQTETEKQNPLNSTVLCKEPGEGCTAHSVFVLIFVGNYFNKLNLWKKDTLGSKPDTF. G20: 34aa; 377-481 bp, MRCKVICTVNYRSLK VSVHICKFYSTIKFIISLR.
EST的概念于1991年被首次提出. 为了从海量基因组数据中得到可靠的蛋白质编码序列信息, 需要构建一个cDNA文库, 即从mRNA反转录生成cDNA链, cDNA的互补链呈现mRNA的序列信息, 因此某一组织或某一状态所有mRNA来源的cDNA文库即可反应该状态下完整的转录信息. 但是由于克隆技术的局限性, 最终得到的cDNA序列往往偏向3'端, 再加上测序反应能力所限, 所以cDNA测序所得到的信息通常只是mRNA的一部分. 这样一段测序结果虽然没有包含完整的mRNA序列信息, 但是足以代表一条mRNA. 因此, 这样一段序列被称为表达序列标签.
目前研究认为, 人基因组中只有2%-3%的序列是蛋白质编码序列, 因此代表蛋白质编码序列的EST数据可以帮助我们方便地进行新基因识别和注释, mRNA成熟过程中的可变剪接分析, 编码序列的多态性位点识别, EST序列中物种特异的重复序列的发现, 以及不同物种之间基于表达序列的比较基因组研究等. 此外, EST序列为Microarray提供了大量的数据源, 以更精细地研究不同组织、不同发育阶段以及不同条件下的转录谱.
生物信息学是一门实践性很强的新兴科学, 早期的生物信息学知识主要来自对核酸序列和蛋白质序列的分析过程, 并将分析结果与实验研究结果进行对比分析, 从中总结出关于序列数据的规律, 后者进一步成为生物信息学的理论基础. 在此基础上, 逐步形成了生物信息学的算法, 体现在核酸和蛋白质序列基本性质的分析、核酸和蛋白质二级结构分析等方面. 作为生物医学研究工作者, 重要的是利用生物信息学的基本原理, 充分利用各种信息资源, 尤其是各种在线的生物医学数据库及相关软件. 近年来, 中医药的研究正逐步深入到生物医学的前沿, 在生物医学信息不断膨胀的21世纪, 如何将中医药的研究与现代最新科技有机的结合, 并充分地利用各种现有的科技资源, 是我们新时代中医药研究工作者的基本要求和迫切需要. 我们课题组在充分利用不同中医治法方药调控大鼠肝癌转录水平的基因芯片信息资源的基础上, 选择其具有明显调控作用的EST为研究目标, 筛选出其全长cDNA序列, 并在可能的条件下进行其功能探讨. 这样的研究, 目前在国内中医药研究领域还是很少报道, 而国外的研究正在蓬勃发展, 各研究机构或研究工作者正在抓紧时间, 以最快的速度抢占各种资源. 本课题筛选到的G2、G4、G5、G11、G18等5个基因已于2005年底至2006年初已被其他的研究机构抢先登录.
在通过mRNA差异显示、代表性差异分析、基因芯片检测等实验技术获得某些基因的部分序列后, 广大研究工作者都迫切地希望得到其全长的cDNA序列, 以便进行其功能分析, 在筛选EST片段全长cDNA序列方面, 目前国内外常用的筛选方法有传统经典的cDNA文库杂交筛选、RACE-PCR扩增及电子克隆等方法. 其中传统经典的cDNA文库杂交筛选方法在基因的cDNA克隆方面作出过重要的贡献, 其方法稳定可靠, 但其缺点也很明显, 即受cDNA文库本身的质量、目的基因在文库中的拷贝数量、操作需要同位素的使用条件等影响与限制. 由于大鼠基因组序列的测序工作已完成[5], 同时, 世界各地的生物医学工作者在研究大鼠的功能基因组方面产生大量的基因片段的数据信息, 以及生物信息学及相关生物软件的深入与开发, 为大多数EST的有效延伸提供可能. 大量的研究证明, EST的电子克隆是克隆其全长cDNA序列的有效途径, 常常起到事半功倍的作用[6-7]. 我们研究的20个EST片段中, 有15个得到了有效的电子延伸, 其中有11个基因的大部分或全部序列通过PCR扩增得到了充分的验证. 也充分说明 生物信息学的有关技术与方法在中医药研究中的应用前景是非常广阔的.
当然, 得到这些EST片段的cDNA序列, 只是我们课题组在深入研究不同治法防治肝癌分子机制方面的一部分. 重要的是后续工作, 即这些基因的功能预测及实验室检测, 因为, 基因的功能检测才是我们的最终目标. 这样的研究工作, 既拓宽了不同中医治法防治肝癌的作用分子机制研究, 也必将丰富生命科学的有关内容.
近年来, 中医药的研究正逐步深入到生物医学的前沿, 在生物医学信息不断膨胀的21世纪, 如何将中医药的研究与现代最新科技有机的结合, 并充分地利用各种现有的科技资源, 是新时代中医药研究工作者的基本要求和迫切需要.
邰升, 副教授, 哈尔滨医科大学附属二院肝胆外科; 陈洪, 副教授, 东南大学附属中大医院消化科.
本文重点克隆部分健脾益气法方药能明显下调肝癌高表达的新基因的cDNA表达序列, 这既是中医治则治法机制研究的重要部分, 也是生命科学研究领域中探讨肿瘤发生发展因素的重要方面.
大量的研究表明, 中医药防治肿瘤是多途径、多基因共同作用的结果. 在已有文献报道中, 大多数报道健脾益气方药对已知基因的作用, 如对信号转导通路的研究.
本研究成功克隆出3个健脾益气方药下调的与肝癌有关的新基因, 并实现登录. 这3个新基因既是中医健脾益气法的作用靶点之一, 也可能与肝癌的发生发展有关.
本文对进一步揭示健脾益气法防治肝癌的分子机制做了探索, 对指导中医临床实践提供了理论依据. 同时, 在未来的新基因功能检测基础上, 将有可能进一步补充肝癌发生的分子机制.
本文立意新颖, 将中医诊治与现代分子生物学结合, 值得推广.
编辑: 程剑侠 电编: 何基才
| 1. | Guan D, Fang Z. Advances in TCM treatment of primary hepatocarcinoma. J Tradit Chin Med. 2000;20:223-226. [PubMed] |
| 4. | 盛 学仕, 方 肇勤, 管 冬元, 赵 晓珍, 吴 中华, 阚 卫兵, 高 必峰. DEN诱发大鼠肝癌过程中肝组织基因表达谱的演变. 中国中医基础医学杂志. 2005;11:737-746. |
| 5. | Gibbs RA, Weinstock GM, Metzker ML, Muzny DM, Sodergren EJ, Scherer S, Scott G, Steffen D, Worley KC, Burch PE. Genome sequence of the Brown Norway rat yields insights into mammalian evolution. Nature. 2004;428:493-521. [PubMed] |