修回日期: 2008-07-08
接受日期: 2008-07-14
在线出版日期: 2008-08-18
目的: 筛选人肝细胞cDNA文库中与乙型肝炎病毒(HBV)全S蛋白相互作用蛋白的基因, 并反向验证HBV全S蛋白候选结合蛋白之间相互作用.
方法: 将全S基因定向克隆到酵母表达载体pDEST 32, 构建正向筛选的诱饵质粒并Western blot法验证其在酵母中的表达. 将诱饵质粒与人肝细胞cDNA文库质粒共同转化MaV203酵母细胞, 在人肝细胞cDNA文库筛选候选结合蛋白, 提取阳性菌落质粒测序, 并分析其生物学性质. 将筛选出的纤维蛋白原α链中下游序列及不同全S变异株基因, 分别定向克隆到pDEST32及pDEST22载体中, 利用Western blot法验证表达. 将诱饵质粒与猎物质粒共同转化MaV203酵母细胞, 以反向酵母双杂交方法验证初筛结果的可靠性及正确性.
结果: 正向的酵母双杂交实验, 经初筛和再转染实验纤维蛋白原α链可与HBV全S蛋白发生相互作用. 再以纤维蛋白原α链为靶基因设计诱饵质粒, 以四种变异的HBV全S蛋白为靶基因设计猎物质粒, 反向酵母双杂交法证实维蛋白原α链中下游可与不同全S变异体(总差异率2%)发生相互作用, 纤维蛋白原α链与全S蛋白的结合域可能为病毒蛋白的前268aa.
结论: 纤维蛋白原α链中下游可与HBV全S蛋白产生特异性结合, 其结合域可能与病毒蛋白的前268aa产生相互作用.
引文著录: 周飞, 任建林, 卢雅丕, 陈美娅, 许鸿志, 潘金水, 蔡稼燕, 董菁. 乙型肝炎病毒全S蛋白与纤维蛋白原α链的相互作用. 世界华人消化杂志 2008; 16(23): 2581-2586
Revised: July 8, 2008
Accepted: July 14, 2008
Published online: August 18, 2008
AIM: To screen the proteins interacting with the whole S protein of hepatitis B virus (HBV) from hepatocyte cDNA library by yeast two-hybrid system, and to validate interacting behavior between fibrinogen alpha chain and the whole S protein by reverse yeast two-hybrid.
METHODS: The whole S gene of HBV was cloned into yeast expression vector pDEST32 to construct a bait plasmid, which was verified by Western blot. The bait plasmid and prey plasmids inserted liver cDNA fragments were cotransformed into yeast cells MaV203 by Liac-mediated transformation. Diploid yeasts were plated on synthetic dropout nutrient medium to screen positive colonies. After extracting and sequencing of prey plasmids from positive colonies, the inserted sequences were bioinformatically analyzed. For reverse yeast two-hybrid, the bait plasmid expressing partial fibrinogen alpha chain and four prey plasmids expressing the whole S protein mutants were re-combined. The reconstituted bait plasmid was cotransformed into yeast cells MaV203 with the four prey plasmids, respectively. Diploid yeasts were plated on synthetic dropout nutrient medium and X-gal assay was performed to validate the interacting behavior.
RESULTS: By yeast two-hybrid technique, prey plasmids that were inserted partial gene coding 266-644 amino acid of fibrinogen alpha chain had a positive reaction with bait plasmid coding the whole S protein of HBV. By reverse yeast two-hybrid, fibrinogen alpha chain protein interacted with the four whole S protein mutants. The protein binding domain of the whole S protein might be the leading 268 amino acids.
CONCLUSION: Fibrinogen alpha chain protein may bind the whole S protein of HBV. The interacting domain is in the 266-644 amino acids of fibrinogen alpha chain and the frontal 268 amino acids of the whole S protein, respectively.
- Citation: Zhou F, Ren JL, Lu YP, Chen MY, Xu HZ, Pan JS, Cai JY, Dong J. Interaction between fibrinogen alpha chain and the whole S protein of hepatitis B virus. Shijie Huaren Xiaohua Zazhi 2008; 16(23): 2581-2586
- URL: https://www.wjgnet.com/1009-3079/full/v16/i23/2581.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i23.2581
胃乙型肝炎病毒(hepatitis B virus, HBV)是一种部分双链DNA嗜肝病毒, 不仅引起急、慢性病毒性肝炎, 而且与肝硬化(liver cirrhosis, LC)、肝细胞癌(hepatic cell carcinoma, HCC)的发生发展密切相关[1-3]. 以往研究认为HBV S区通过三个启始密码子(ATG)被人为的分为三个结构区, 前-S1, 前-S2和S区, 编码三种长度的包膜蛋白, 即大蛋白(LHBs)、中蛋白(MHBs)和主蛋白(SHBs). 最近董菁 et al的研究认为中国HBV部分病毒株的S区存在前前-S区, 即在前-S1之前存在一编码区域, 这提示HBV包膜蛋白可能存在第四种形式, 即全S蛋白, 其编码范围为前前-S区多肽和LHBs[4-5]. 由于HBV是通过其表达的病毒蛋白与肝细胞蛋白相互作用导致疾病的进展[6-8], 因此, 我们利用酵母双杂交技术寻找并证实可能与全S蛋白相互作用的人类肝细胞内蛋白, 以探讨全S蛋白功能, 进一步明确HBV可能存在的致病机制.
ProQuestTM酵母双杂交系统、人肝细胞cDNA文库(货号10422-020)、MaV203酵母高效感受态、酵母细胞质粒提取试剂盒等均购自Invitrogen公司. 酵母YPAD培养基、不含氨基酸的酵母氮碱基等购自Oxoid公司, 抗HIS、抗Pre S1、抗HBs鼠单克隆抗体购自Perfect公司. 内切酶、DNA聚合酶购自TaKaRa公司. 各种氨基酸及质粒提取试剂盒均购自北京博大泰克公司. 其他试剂为进口分装或国产分析纯.
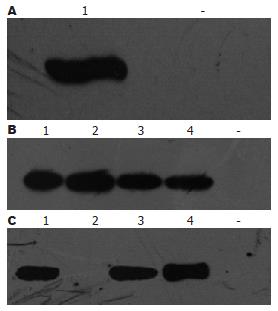
1.2.1 正向诱饵质粒载体的构建以及其表达的验证: 按Invitrogen公司操作指南, 以多聚酶链反应(polymerase chain reaction, PCR)法扩增HBV全S蛋白编码基因, 将本研究室自HBV感染者血清中克隆的HBV全S基因(GenBank: EU075334)定向克隆入酵母表达载体pDEST32中, DNA测序验证后命名该重组诱饵质粒为pDEST32-w216, Western blot方法验证诱饵质粒在酵母细胞中的表达.
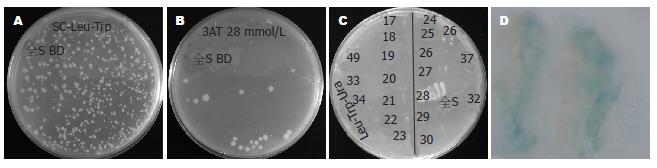
1.2.2 正向筛选靶蛋白候选结合蛋白: 按照Invitrogen公司酵母双杂交系统操作指南进行酵母双杂交筛选, 将诱饵质粒pDEST32-w216与包含肝细胞cDNA文库经过醋酸锂法转入MaV203酵母细胞中, 在SC-Leu-Trp、SC-Leu-Trp-His+3AT、SC-Leu-Trp-Ura等缺陷培养基以及X-gal分析等筛选出的阳性克隆, 将初步筛选获得的阳性克隆中的猎物质粒与pDEST32-w216进行再转染试验验证, 验证后将阳性克隆猎物质粒进行DNA测序, 测序结果在美国国立卫生院(NIH)网站进行BLAST生物信息学分析.
1.2.3 反向验证诱饵及猎物载体的构建: (1)诱饵载体的构建: 以正向筛选出的纤维蛋白原α链诱饵质粒为模板(GenBank号NP_068657), 根据测序结果设计引物, 上游引物5'-ATGGTCG ACTGGACGATTTCGATGATGAAGATACC-3'(SalⅠ); 下游引物5'-GTGCGGCCGCTAATG ATGATGATGATGATGAGAAGTGTGGAT ACCTCTGAC-3'(NotⅠ), 下游引物带有HIS标签. PCR条件: 94℃ 3 min, 94℃ 30 s, 58℃ 30 s, 72℃ 60 s, 经35个循环, 最后72℃ 7 min. PCR产物为含NotⅠ和SalⅠ酶切位点的纤维蛋白原α链蛋白基因, 双酶切后定向克隆到质粒pDEST32, DNA测序验证后命名为pDEST32-Fib. (2)猎物载体的构建: 以含有突变的全S序列质粒为模板, 其DNA序列可按下列GenBank号EU075334(S216, 即正向筛选时的靶基因序列)、EU082433(S245)、EU086837(S246)、EU086840(S249)寻找, 上游引物: 5'-ATGGTCGA CTGGATGCAGTTAATCATTACTTC-3'(SalⅠ), 下游引物: 5'-GTGCGGCCGCTAGGTTCT AATGTATACCCAAAGAC-3'(NotⅠ), PCR扩增-酶切后与pDEST22连接, DNA测序验证后分别命名为: pDEST22-w216、pDEST22-w245、pDEST22-w246和pDEST22-w249.
1.2.4 反向验证诱饵及猎物载体靶基因的表达: 将重组质粒pDEST32-Fib、pDEST22- w216、pDEST22-w245、pDEST22-w246和pDEST22-w249等质粒分别转入MaV203酵母细胞, 转化诱饵载体酵母细胞在亮氨酸营养缺乏液体培养基培养过夜, 转化猎物载体酵母细胞在色氨酸营养缺乏液体培养基培养过夜, 离心收集酵母细胞, 酵母裂解液裂解后, 保留上清备用.
Weatern blot方法验证诱饵蛋白表达: 按照常规操作处理样品后, 分别经120 g/L SDS-PAGE胶电泳, 经5%脱脂牛奶封闭后, 与1:1000抗HIS标签的鼠mAb孵育2 h, 洗脱后再与1:1000羊抗鼠二抗室温孵育2 h, 增强化学发光法进行放射自显影. 猎物蛋白表达的验证: 同诱饵蛋白验证, 仅将一抗替换为1:1000抗preS1或抗HBs鼠mAb.
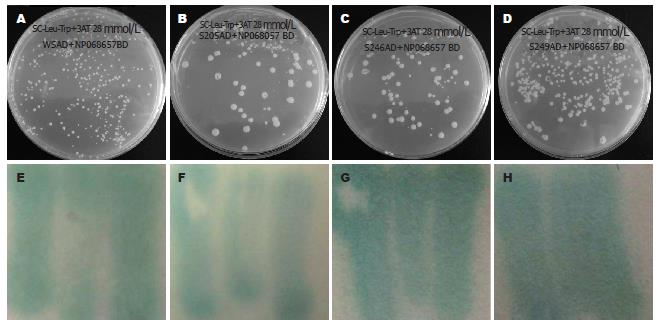
1.2.5 反向验证: 将构建好的重组诱饵及猎物质粒用醋酸锂法共转入高效MaV203酵母感受态细胞, 分别涂部在SC-Leu/-Trp, 及SC-Leu/-Trp/-His+28 mmol/L 3AT(抑制自激活)培养基上. 将生长在SC-Leu/-Trp/-His+3AT的阳性克隆划线到SC-Leu/-Trp/-Ura的培养基上, 最后将阳性菌落接种于YPAD培养皿影印于硝酸纤维素膜用液氮裂解后置于含X-gal的Z-buffer反应体系上检查a-半乳糖苷酶活性, 变成蓝色的为阳性菌落. 试验时交叉以pDEST32与pDEST22- w216、pDEST22-w245、pDEST22-w246和pDEST22-w249共转染, 或pDEST32-Fib与pDEST22共转染为阴性对照.
HBV全S基因为本实验室克隆并测序, GenBank号为: EU075334, C2亚基因型. 利用定向克隆技术, 将全S基因克隆到pDEST32中, 经验证后将诱饵质粒命名为pDEST32-w216(图1).
按照Invitrogen公司酵母双杂交操作指南方法, 我们将pDEST32-w216与肝细胞文库(克隆在pDEST22中)共转染MaV203酵母细胞, 之后利用不同缺陷培养基来筛选阳性克隆, 最后筛选出的阳性克隆可以表达His+、Ura+、LacZ报告基因(图2), 将阳性克隆送检测序. 正向筛选获得阳性克隆进行DNA测序, 经过BLAST, 确认其中一个阳性克隆内猎物质粒插入的cDNA序列含有部分纤维蛋白原α链序列, 即编码266-644aa部分的序列.
为进一步验证正向筛选结果的可靠性以及明确HBV全S蛋白与候选相互作用蛋白开放读码框(open reading frame, ORF)的正确性, 我们进行了反向酵母双杂交试验进行验证. 利用定向克隆技术, 将含HIS标签的纤维蛋白原α链基因克隆到pDEST32中, 经验证后将诱饵质粒命名为pDEST32-Fib(图3A).
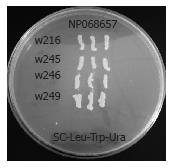
为验证相互作用的代表性, 我们选择了4个HBV全S基因为靶基因构建猎物质粒, 靶基因GenBank号分别为w216(EU075334)、w245(EU082433)、w246(EU086837)、w249(EU086840), 均为C2亚基因型. 利用定向克隆技术, 将全S基因克隆到pDEST22中, 经验证后将诱饵质粒命名为pDEST22-w216、pDEST22-w245、pDEST22-w246、pDEST22-w249(图3B-C).
试验所采用的不同的全S蛋白之间存在点替换突变, w216与w246、w249之间存在2.0%(9/445aa)的差异, 9个替换突变分别位于: 前前-S区1个, 前S1区下游2个, 前S2上游3个, 主蛋白区中游3个. w216与w245之间具有明显差异, 由于w245基因发生编码移行突变, w245前268aa(前前-S区至主蛋白上游)与w216之间仅有1个替换突变, w245在第269aa之后编码部分多聚酶序列, 但比较结果提示其蛋白具有一定结构相似性(图4).
按照Invitrogen公司酵母双杂交操作指南, 在交叉设计阴性对照基础上, 我们分别将诱饵质粒pDEST32-Fib与pDEST22-w216、pDEST22-w245、pDEST22-w246、pDEST22-w249共转染MaV203酵母细胞, 之后利用不同缺陷培养基来筛选阳性克隆, 阳性克隆可以表达His+、Ura+、LacZ报告基因(图5, 6).
既往研究认为HBV S区通过三个启始密码子(ATG)被人为的分为三个结构区, 前-S1, 前-S2和S区, 编码三种长度的包膜蛋白, 即大蛋白(LHBs)、中蛋白(MHBs)和主蛋白(SHBs). 最近的研究[2]认为中国HBV部分病毒株的S区中存在一前前S区, 即在前S1之前存在一编码区域, 这提示HBV包膜蛋白可能存在第四种形式[9]. 我们将前前S区至S终止密码子编码的蛋白命名为全S蛋白, 以其为靶蛋白, 利用酵母双杂交技术寻找并验证其与肝细胞相互作用的蛋白[10].
酵母双杂交方法是目前较为可靠的蛋白-蛋白相互作用研究手段之一[11], 国内学者也将该手段应用于HBV囊膜蛋白的结合蛋白的研究之中[12-14]. 我们采用的Invitrogen公司ProQuestTM酵母双杂交系统, 该系统特点是利用低复制载体控制猎物基因表达, 同时联合3个报告基因系统以最大程度的减少酵母细胞双杂交试验系统的假阳性. 按照该系统操作指南, 候选结合蛋白的筛选需要经过3个步骤: 初筛、再转染和反向杂交. 为排除初筛获得的阳性克隆的操作误差, 将阳性克隆中的猎物质粒提取后与诱饵质粒再次共转染, 只有初筛和再转染均能在三缺培养基中生长的阳性克隆才进行DNA测序验证. 虽然猎物质粒中插入的cDNA编码具有其原始序列的特异性, 但在插入pDEST22后也可能在表达过程中出现移位, 需要通过反向酵母双杂交试验验证. 反向杂交同时也将展示初筛获得的候选结合蛋白与正向筛选时猎物蛋白的突变体之间的可能的相互作用, 更加有利的证明相互作用[15]. 通过正向筛选获得的阳性克隆猎物质粒含有纤维蛋白原α链的中下游部分, 即266-644aa(全长644aa), 通过反向酵母双杂交实验可以得出以下结论: 以纤维蛋白原α链的266-644aa部分为诱饵质粒除了可以与表达原靶基因产物(EU075334)的猎物质粒发生良好的双杂交反应外, 还可以与2种发生替换突变(2%, 9/445aa)的HBV全S蛋白变异株pDEST22-w246、pDEST22-w249产生良好的相互作用, 这提示纤维蛋白原α链与HBV全S蛋白的相互作用不因点替换突变所改变, 提示相互作用可能具有普遍性. 靶基因w245编码的前268aa与w216(EU075334)仅有一个替换突变发生(135aa), 269aa下游为移行突变, 编码部分多聚酶序列, 但多聚酶序列与全S蛋白具有一定结构类似性. pDEST32-Fib与pDEST22-w245实验的阳性结果提示纤维蛋白原α链与HBV全S蛋白的具体结合域可能位于病毒蛋白的上游部分, 下游氨基酸序列的类似性可能对反向酵母双杂交的阳性结果产生影响.
纤维蛋白原由肝细胞分泌的糖蛋白, 为结构蛋白参与机体凝血过程, 分子由两对α链、β链及γ链组成, 每3条肽链(α、β、γ)绞合成索状, 形成两条索状肽链, 在N末端有二硫键使态个分子得到稳定. α及β肽链的N-端分别有一段16个及14个氨基酸的小肽, 称为纤维肽A及B[16]. Al Ghumlas et al[17]研究发现经过α-干扰素治疗后的慢性肝炎患者血清中的纤维蛋白原的表达较治疗前有所上升. Brennan et al[18]研究发现纤维蛋白原基因突变导致纤维蛋白原在肝细胞中的内质网聚积, 导致肝细胞的坏死, 最终导致肝硬化的发生. Liu et al[19]研究发现纤维蛋白原在肝细胞癌的表达下降. 而Fan et al[20]研究发现在肝细胞癌组织中表达上升, 尤其在转移灶组织高于其邻近的非癌组织. Yang et al[21]利用HBV的全X蛋白进行酵母双杂交肝文库的筛选, 也筛选出了纤维蛋白原β多肽, 这说明HBV编码的病毒蛋白可能通过与纤维蛋白原家族相互作用, 从而产生生物学作用. 我们筛选出的为纤维蛋白原α链也可能在肝细胞内与全S蛋白结合, 导致肝细胞内的纤维蛋白原的聚积, 肝细胞的坏死, 肝硬化的发生.
总之, 本研究通过对酵母双杂交初筛、再转染以及反向杂交实验, 初步证实了HBV全S蛋白的上游可能与纤维蛋白原α链中下游发生相互作用, 这一发现为该病毒蛋白可能存在的生物学作用提供研究思路. 为了进一步研究结果的可靠性, 我们将应用免疫共沉淀或哺乳动物细胞双杂交方法予以验证[22-23].
病毒蛋白-宿主蛋白相互作用是目前的研究热点之一. 本研究试图通过相对成熟的酵母双杂交技术, 对HBV全S蛋白的相互作用蛋白进行筛选和验证, 其目的是为了探讨在细胞内过量表达的囊膜蛋白干扰肝细胞功能、病毒蛋白的表达后加工等方面的可能性.
张晓岚, 教授, 河北医科大学第二医院消化内科; 陈红松, 副研究员, 北京大学人民医院肝病研究所
目前针对HBV X蛋白(HBx)的结合蛋白有较多研究, 国内成军教授的实验室针对前S区、中蛋白的候选结合蛋白进行了初步筛选, 但由于缺少多种技术手段的验证, 国内外没有针对HBV囊膜蛋白结合蛋白有所定论.
国内李丹 et al利用酵母双杂交技术对HBV前S1多肽结合蛋白进行了筛选并验证; 成军 et al报道了以前前S、中蛋白、截短型中蛋白为靶蛋白的酵母双杂交实验, 筛选出醛缩酶B等多个候选结合蛋白. 本文在酵母双杂交基础上, 进一步进行反向杂交实验, 不仅验证初筛结果的可靠性, 而且对可能的相互结合功能域进行初步探讨.
目前没有关于全S蛋白候选结合蛋白的筛检及验证报道. 本研究利用酵母双杂交系统操作性较好的特点, 首先自肝cDNA文库中筛选出全S蛋白可能的宿主结合蛋白-纤维蛋白原α链, 之后再以纤维蛋白原α链设计诱饵载体, 与全S蛋白的4种变异体进行反向酵母双杂交, 通过该手段验证初筛结果的可靠性, 并初步证实相互结合作用的普遍性.
本文通过酵母双杂交以及反向杂交实验, 初步证实纤维蛋白原α链中下游部分可能与HBV全S蛋白的中上游部分发生结合作用, 为进一步阐述HBV相关性肝纤维化提供了一条新的研究思路.
1 全S蛋白: 包含前前S、前S1、前S2和主蛋白的HBV完整囊膜蛋白.
2 酵母双杂交反向杂交实验: 仍利用酵母双杂交系统, 将正向筛选猎物质粒测序结果作为诱饵质粒靶基因, 以正向筛选诱饵质粒靶基因多种变异体为猎物质粒靶基因, 进行的验证性酵母双杂交实验, 该实验是一种有效的蛋白-蛋白相互作用的验证方法.
本研究创新性强, 层次分明, 思路清晰, 结果可信, 结论可靠, 具有很好的学术价值.
编辑: 李军亮 电编:何基才
| 1. | Ganem D, Prince AM. Hepatitis B virus infection--natural history and clinical consequences. N Engl J Med. 2004;350:1118-1129. [PubMed] [DOI] |
| 2. | Park NH, Song IH, Chung YH. Chronic hepatitis B in hepatocarcinogenesis. Postgrad Med J. 2006;82:507-515. [PubMed] [DOI] |
| 3. | Pollicino T, Squadrito G, Cerenzia G, Cacciola I, Raffa G, Craxi A, Farinati F, Missale G, Smedile A, Tiribelli C. Hepatitis B virus maintains its pro-oncogenic properties in the case of occult HBV infection. Gastroenterology. 2004;126:102-110. [PubMed] [DOI] |
| 6. | Lwa SH, Chen WN. Hepatitis B virus X protein interacts with beta5 subunit of heterotrimeric guanine nucleotide binding protein. Virol J. 2005;2:76. [PubMed] [DOI] |
| 7. | Nijhara R, Jana SS, Goswami SK, Rana A, Majumdar SS, Kumar V, Sarkar DP. Sustained activation of mitogen-activated protein kinases and activator protein 1 by the hepatitis B virus X protein in mouse hepatocytes in vivo. J Virol. 2001;75:10348-10358. [PubMed] [DOI] |
| 8. | Zhong S, Chan JY, Yeo W, Tam JS, Johnson PJ. Hepatitis B envelope protein mutants in human hepatocellular carcinoma tissues. J Viral Hepat. 1999;6:195-202. [PubMed] [DOI] |
| 10. | 周 飞, 任 建林, 卢 雅丕, 陈 美娅, 陈 建民, 刘 明, 张 波, 董 菁. 肝细胞cDNA文库中乙型肝炎病毒全S蛋白筛选结合蛋白基因. 中华肝脏病杂志. 2008;16:304-305. |
| 11. | Miller J, Stagljar I. Using the yeast two-hybrid system to identify interacting proteins. Methods Mol Biol. 2004;261:247-262. [PubMed] |
| 12. | 蔺 淑梅, 张 树林, 成 军, 刘 敏, 王 琳, 王 建军, 杨 倩, 黄 燕萍, 白 桂芹. 肝细胞cDNA文库中乙型肝炎病毒前-前-S蛋白结合蛋白基因筛选. 世界华人消化杂志. 2004;12:2907-2910. [DOI] |
| 15. | White MA. The yeast two-hybrid system: forward and reverse. Proc Natl Acad Sci U S A. 1996;93:10001-10003. [PubMed] [DOI] |
| 16. | 侯 韬. 纤维蛋白原结构与功能研究进展. 国外医学·输血及血液学分册. 2001;24:479-482. |
| 17. | Al Ghumlas AK, Abdel Gader AG, Al Faleh FZ. Natural anticoagulants and fibrinolytic activity following interferon therapy in chronic viral hepatitis. Blood Coagul Fibrinolysis. 2008;19:263-267. [PubMed] [DOI] |
| 18. | Brennan SO, Wyatt J, Medicina D, Callea F, George PM. Fibrinogen brescia: hepatic endoplasmic reticulum storage and hypofibrinogenemia because of a gamma284 Gly-->Arg mutation. Am J Pathol. 2000;157:189-196. [PubMed] |
| 19. | Liu Y, Zhu X, Zhu J, Liao S, Tang Q, Liu K, Guan X, Zhang J, Feng Z. Identification of differential expression of genes in hepatocellular carcinoma by suppression subtractive hybridization combined cDNA microarray. Oncol Rep. 2007;18:943-951. [PubMed] |
| 20. | Fan BL, Zhu WL, Zou GL, Luo GS, Xu CL, Zhao WX. [Cloning and identification of fibrinogen gamma polypeptide (FGG) gene differentially expressed in human hepatocellular carcinoma]. Ai Zheng. 2004;23:249-253. [PubMed] |
| 21. | Yang Q, Cheng J, Dong J, Zhang J, Zhang SL. Molecular epidemiological study on pre-X region of hepatitis B virus and identification of hepatocyte proteins interacting with whole-X protein by yeast two-hybrid. World J Gastroenterol. 2005;11:3473-3478. [PubMed] |
| 22. | Matsuzawa S, Reed JC. Yeast and mammalian two-hybrid systems for studying protein-protein interactions. Methods Mol Biol. 2007;383:215-225. [PubMed] [DOI] |
| 23. | Tanowitz M, von Zastrow M. Identification of protein interactions by yeast two-hybrid screening and coimmunoprecipitation. Methods Mol Biol. 2004;259:353-369. [PubMed] |