修回日期: 2008-04-02
接受日期: 2008-05-22
在线出版日期: 2008-06-08
HBV囊膜蛋白基因具有多个可变的起始编码位置, 囊膜蛋白可能包含前前-S、前-S1、前-S2以及主蛋白等区域, 不同长度的囊膜蛋白组分与肝细胞内蛋白的相互作用机制并不清楚. 本文着重探讨了现有的蛋白-蛋白相互作用的研究手段, 总结了目前宿主肝细胞可能与HBV囊膜蛋白相互作用的蛋白, 但目前获得的大部分结果需进一步验证.
引文著录: 周飞, 任建林, 董菁. 乙型肝炎病毒囊膜蛋白候选结合蛋白的研究进展. 世界华人消化杂志 2008; 16(16): 1788-1792
Revised: April 2, 2008
Accepted: May 22, 2008
Published online: June 8, 2008
There are at least 4 initial coding positions in S gene of hepatitis B virus (HBV), encoding pre-pre-S, pre-S1, pre-S2 as well as major protein. There are no definite evidences to prove that envelope protein will interact with human cellular protein. This review focused on research approaches to the existing protein - protein interaction and summarized proteins of liver cells that may interact with current HBV envelope protein. However, most of currently available results need further verification.
- Citation: Zhou F, Ren JL, Dong J. Advances of candidate binding protein to envelope protein of hepatitis B virus. Shijie Huaren Xiaohua Zazhi 2008; 16(16): 1788-1792
- URL: https://www.wjgnet.com/1009-3079/full/v16/i16/1788.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i16.1788
2005年世界卫生组织调查结果显示全世界约有4亿HBV感染者[1], HBV严重威胁着人类的健康. 近年来研究发现HBV编码的病毒蛋白在感染肝细胞方面起重要作用, 对肝细胞代谢有重要影响, 但病毒蛋白产生病理作用的生物学机制尚不明了. 本文对近年来HBV编码病毒囊膜蛋白及其结合蛋白相互作用的研究进展进行总结.
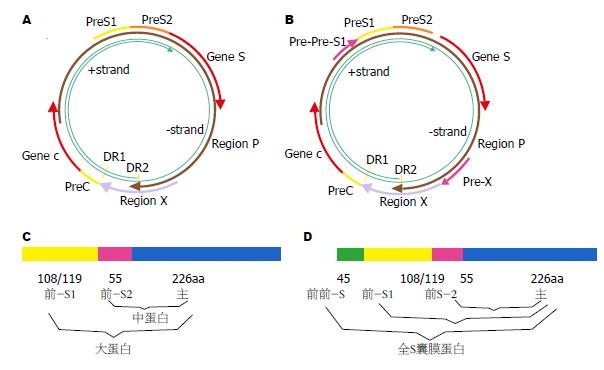
HBV基因组是一种部分双链DNA病毒, 具有独特而精密的结构, 全长约3200 bp. 1980年代初Galibert et al[2]通过对HBV基因组进行开放读码框(open reading frame, ORF)进行了界定分析, 确定了前C/C、P、S、X基因, 之后在S基因上游确定了前-S1、前-S2区[3]. 1983年Fujiyama et al[4]在其研究中被提及前-S区之前的ORF, 作者认为该ORF身份不明; Mimms et al[5]将Fujiyama研究的克隆分析后认为, 该段多肽是前-S1多肽的一部分, 但该段编码存在的广泛性未得到承认. 目前国内外仍沿用公认的前-S1、前-S2和S基因的分区方法(图1), 分别编码HBsAg包括小(SHBs), 中(MHBs)和大(LHBs)蛋白, 他们各自具有糖基化和非糖基化两种形式[6-9]. 近年来董菁 et al[10]围绕HBV准种的研究提供了我国大陆新的HBV分子流行病学资料, 认为Fujiyama et al发现的前-S1上游的编码序列在亚洲具有地域流行性特征, 故提出前前-S区这一概念, 并将我国的HBV部分病毒株的S区重新定义为前前-S、前-S1、前-S2及S区基因; 结合前-X流行病学资料, 新提出的HBV基因结构图(图1), 本文提到的全S蛋白即编码前前-S多肽与大蛋白的病毒蛋白, 具有一定的地域性.
HBV进入肝细胞的机制不明确, 可能是通过病毒蛋白与肝细胞胞膜蛋白(受体)相互作用, 或者是通过肝细胞胞饮作用进入细胞, 目前尚无定论. HBV囊膜蛋白是病毒与肝细胞膜病毒受体结合的最有可能的蛋白成分. HBV囊膜蛋白在被感染肝细胞内呈过度表达, 可能与宿主蛋白之间存在相互作用, 进而影响肝细胞功能. 下文探讨了目前常用的蛋白-蛋白相互作用的研究方法, 并对近年来囊膜蛋白结合蛋白的研究结果进行总结.
目前常用的蛋白-蛋白相互作用的研究方法主要有酵母双杂交技术(yeast two -hybrid)、哺乳动物细胞双杂交(mammalian two-hybrid)、噬菌体表面展示(phage display)技术、免疫共沉淀(Co-Immunoprecipitation)及谷胱甘肽(GST)-pull down等生物学技术. 下面就这5种分子生物学技术进行概述:
酵母双杂交技术是利用酵母双杂交系统研究细胞内蛋白之间相互作用的一种分子遗传学技术[11], 具有操作简便、不经蛋白质纯化即可获得配体蛋白的编码基因等特点, 可从cDNA文库和基因文库中直接筛选出蛋白间相互作用[12]. 该技术主要应用于: 验证蛋白-蛋白相互作用、确定蛋白-蛋白特异性相互作用的关键结构域、筛选文库以获得可能与已知蛋白存在特异相互作用的蛋白质等[13-17]. 该技术缺点在于: 不适于所有蛋白相互作用的研究、可能筛出假阳性的结果、酵母菌株中表达外源蛋白质常会带来毒性作用造成的假阴性结果[18-20].
哺乳动物细胞双杂交是在体内检测蛋白-蛋白相互作用的强有力方法, 其原理类似于酵母双杂交, 需要诱饵蛋白、猎物蛋白相互作用后才激活报告基因的表达, 只不过两者的宿主不同, 一个在哺乳动物细胞, 另一个在酵母细胞[21]. 哺乳动物细胞双杂交的优点在于, 所研究靶蛋白基本处于原来构型, 保持了诸如糖基化、磷酸化和酰基化等翻译后的修饰; 报告系统可以定量显示相互作用. 缺点在于, 细胞转化效率不稳定, 重复性差; 不适合文库的筛选. 哺乳动物细胞双杂交适用于蛋白的相互作用的确证研究, 或应用于识别涉及蛋白-蛋白相互作用的域/残基, 通常用于酵母双杂交筛选出的相互作用的蛋白进行验证[22-23].
噬菌体展示技术是一种基因表达产物与亲和选择相结合的技术, 该技术以改构的噬菌体为载体, 把待选基因片段以多肽/蛋白形式展示于噬菌体表面, 通过与另一个噬菌体展示库的相互作用, 可以导致待选基因表达产物选择性地与其可能具有相互作用的某个外源肽相结合, 从而研究该噬菌体所含外源基因的生物学功能[24-25]. 噬菌体展示技术优点在于, 序列的多样性, 其组合肽文库有着巨大的编码潜力; 可通过体外筛选组合肽文库得到与靶目标结合的多肽配基. 噬菌体展示技术缺点在于, 受噬菌体系统所固有的生理属性的限制, 其结果经常会得到非相关的多肽序列. 该技术主要应用于重组肽库(识别肽受体配基)的亲和筛选, 确定单克隆抗体的抗原表位, 选择酶作用底物以及筛选克隆的全套抗体[26-28].
免疫共沉淀技术是以抗体和抗原之间的专一性作用为基础的应用于研究蛋白质相互作用的一种方法[29], 可有效确定2种蛋白在完整细胞内生理性相互作用的方法. 其原理是: 当细胞在非变性条件下被裂解时, 完整细胞内存在的许多蛋白质-蛋白质间的相互作用被保留了下来, 之后以特异性抗体检测已知蛋白与其他蛋白的结合作用. 该方法优点在于, 相互作用的蛋白都是经翻译后修饰的, 处于天然状态; 蛋白的相互作用是在相对自然状态下进行的, 可以避免实验手段造成的影响; 可以分离天然状态的相互作用蛋白复合物[30-31]. 该方法缺点在于, 所检测的两种蛋白可能通过第3种蛋白在中间起桥梁作用而相互作用; 实验前需要获得至少1种蛋白的抗体. 该方法用于测定2种目标蛋白是否结合, 也用于确定一种已知蛋白的结合蛋白, 往往还需要联合质谱分析来判定结合蛋白的序列及性质. 适用于酵母双杂交相互作用蛋白的验证[32].
GST-pull down技术利用了融合表达蛋白的谷胱甘肽(GST)标签对GST偶联球珠的亲和性, 从非相互作用蛋白的溶液中纯化相互作用蛋白, 该技术在于探测蛋白在溶液中的相互作用, 而这种作用在膜性分析中可能是检测不到的[33]. 该技术缺点在于, 只用于确定体外的相互作用, 这种体外的相互作用需要进一步在体内通过单独的方法进行证实, 如免疫共沉淀; 对检测的未知蛋白必须有足够的量存在; 必须对每个蛋白质复合物的分析进行优化, 其中最重要的变量是发生相互作用的缓冲液在检测SDS聚丙烯酰胺凝胶出现阴性结果时需要进行变性/复性过程才有可能出现阳性结果. 该技术通常应用于确定融合蛋白与未知(或靶)蛋白间及证实探针蛋白与已知蛋白可疑的相互作用[34-36].
综上所述各种分子生物学技术各有其优缺点, 酵母双杂交和噬菌体展示技术主要适用于文库的筛选, 前者更适合进行蛋白-蛋白相互作用研究, 后者更适合抗体或多肽药物的筛检; 哺乳动物细胞双杂交、免疫共沉淀和GST-pull down技术不适用于结合蛋白的筛选, 更适合于验证特定蛋白之间相互作用关系. 在实际的研究中, 单一实验方法是不能完全确证蛋白之间的相互作用, 需要综合的运用, 各种方法相互验证[37-39].
由于HBV囊膜蛋白的结构复杂, 国内外多个研究组对HBV S基因分段编码多肽/蛋白与宿主细胞蛋白之间的相互作用总结如下. (1)前前-S蛋白结合蛋白: 蔺淑梅 et al[40]利用酵母双杂交技术, 以前前-S蛋白为诱饵, 对肝文库进行筛选, 发现了30种已知蛋白基因及8种未知蛋白, 如: 人类醛缩酶B, 人类金属硫蛋白2A在多个研究结果中出现, 但未见作者用其它方法进一步验证的报道. 该研究获得前前-S蛋白结合蛋白的结果, 只能提示前前-S蛋白可能与肝文库中的38种蛋白结合, 但特异性可能不高. (2)前-S1蛋白结合蛋白: Ryu et al[41]利用GST-pull down技术研究前-S1蛋白结合蛋白, 发现了p80(80 kDa)蛋白能和前S1蛋白结合, 但未见有后续研究报道; 李丹 et al[42-43]利用酵母双杂交技术初步证实了前-S1蛋白能与多肽相关复合体α亚单位(NACA)结合, 并利用免疫共沉淀技术进行了验证, 结果高度提示NACA可能与前-S1蛋白产生特异性结合, 研究结果可信度较高. 李丹 et al[44]继续利用酵母双杂交技术又发现了一个能与前-S1蛋白结合的蛋白, 为丝裂原活化蛋白激酶, 有待其进一步验证. 董菁 et al[45]利用噬菌体表面展示技术来研究前-S1蛋白的结合, 发现了神经胶质瘤抑制基因区候选基因2(GLTSCR2)能与前-S1蛋白结合, 未再进行深入研究, 故筛选出的结合肽有待验证. (3)前-S2蛋白结合蛋白: 陆荫英 et al[46]利用酵母双杂交技术, 以前-S2蛋白为诱饵, 对肝文库进行筛选, 发现10个能与前-S2蛋白的结合蛋白, 其中8个为已知功能蛋白, 2个为未知功能蛋白, 但未对其筛选结果进行验证. 其中的金属硫蛋2A基因及白蛋白为多个不同大小囊膜蛋白的研究结果中出现, 故需进一步验证. (4)前-S蛋白结合蛋白(前-S1蛋白和前-S2蛋白): Deng et al[47]利用噬菌体表面展示技术来寻找前-S蛋白的配体, 筛选出5个氨基酸序列能与前-S1结合, 未进一步验证. Deng et al[48]再利用噬菌体表面展示技术来寻找前-S蛋白的受体, 发现了脂蛋白脂肪酶A能与前-S蛋白结合, 并利用了GST-pull down技术进行了验证, 故可信度高. (5)中蛋白结合蛋白: 李志群 et al[49]利用酵母双杂交技术, 以中蛋白为诱饵, 对肝文库进行筛选, 研究结果提示了醛缩酶B和未知功能MBP1蛋白能与中蛋白结合, 但也未对其进一步研究. 其中的醛缩酶B为多个不同大小的囊膜蛋白研究结果中都有. (6)截短型中蛋白结合蛋白: 李志群 et al[50]利用酵母双杂交技术, 以羧基末端截短型中蛋白(MHBst167)为诱饵, 对肝文库进行筛选, 获得了7个已知功能蛋白能与羧基末端截短型中蛋白结合蛋白, 但未进一步研究. 其中筛选出的醛缩酶B结合蛋白也能与前前-S蛋白及中蛋白结合. (7)主蛋白结合蛋白: Toh et al[51]利用酵母双杂交分离的泛素系统(一种新型的膜蛋白酵母双杂交系统), 进行主蛋白的膜结合蛋白筛选, 获得了3个能与主蛋白相结合的蛋白; 分别为硫氧蛋白相关转膜蛋白2、连接相关蛋白复合体2、补体加速降解因子; 而Tan et al[52]利用噬菌体表面展示技术筛选出ETGAKPH多肽能与主蛋白特异位点结合. 两者的研究都未见后续报道, 尚待验证可信度不高. (8)全S蛋白结合蛋白: Bai et al[53]利用酵母双杂交技术对全S蛋白进行了筛选, 获得10个能与全S蛋白结合的蛋白, 并利用免疫共沉淀对其筛选出的蛋白进行了验证, 证明一个未知功能蛋白与全S蛋白特异性结合. 筛选结果中有白蛋白与以前S2蛋白为诱饵筛选的结果相同; 而筛选出的金属硫蛋白A2不仅与前S2蛋白为诱饵筛选的结果相同, 也与以前前-S蛋白为诱饵筛选出的结果也相同. 这说明白蛋白和金属硫蛋白A2可能与囊膜蛋白结合, 从而对肝细胞产生影响.
乙型肝炎病毒囊膜蛋白的研究取得一定成果, 筛选出的结果大部分为非特异蛋白, 其内部可能存在特定的空间结构可与囊膜蛋白相互结合, 从而影响肝细胞的新陈代谢. 从以上结果中未能找到能够与囊膜蛋白结合的而又明确的肝细胞膜受体. 李丹研究发现的丝裂原活化蛋白激酶信号转导通路, 可能为囊膜蛋白产生生物学作用的机制, 但未对其进行验证, 仍待进一步研究. 从以上研究结果看出, 只有李丹 et al的研究联合了两种以上的蛋白-蛋白相互作用方法, 故可信度高.
关于HBV囊膜蛋白的结合蛋白方面研究取得一定成果, 但主要问题在于上述大部分结果多是单向试验结果, 目前的结果中没有跨膜蛋白被证明与HBV囊膜蛋白具有明确的相互作用, 其肝细胞受体假说尚未明确, 仍需我们进一步去研究.
HBV的S基因表现出编码多样性, 其不同长度的编码产物可能通过蛋白-蛋白相互作用影响宿主细胞的代谢、分化、凋亡乃至肿瘤的产生.
张占卿, 主任医师, 上海市(复旦大学)公共卫生中心; 高润平, 教授, 吉林大学第一医院肝病科
国内成军、王小众 et al实验室较为系统的应用酵母双杂交方法筛选了HBV囊膜蛋白各个分区的宿主结合蛋白, 有部分结果予以多种技术手段加以验证, 结果较为可靠, 为进一步研究提出了新的思路.
本文系统阐述了目前HBV S基因的研究现状, 指出全S蛋白可能是一种重要的病毒蛋白形式, 主张加强对病毒蛋白-宿主蛋白相互作用研究, 并探讨病毒蛋白影响宿主细胞的生长、代谢及分化的具体途径.
本文收集并系统分析了目前HBV囊膜蛋白候选结合蛋白的研究方法及其初步结果, 为下一步的研究方向提出新的思路.
1 前前S编码区: 是指存在于前S1区之前的一段编码区域, 长度135 bp, 编码45 aa, 可能具有前导肽功能. 2 全S蛋白: 是指编码自前前S区至主蛋白的第4种HBV囊膜蛋白表达形式, 可能是亚洲流行病毒株的一种特异性表现.
本文内容新颖, 科学性较强, 思路清晰, 具有很好的学术价值.
编辑: 李军亮 电编:郭海丽
| 1. | Gish RG. Current treatment and future directions in the management of chronic hepatitis B viral infection. Clin Liver Dis. 2005;9:541-565. [PubMed] [DOI] |
| 2. | Galibert F, Mandart E, Fitoussi F, Tiollais P, Charnay P. Nucleotide sequence of the hepatitis B virus genome (subtype ayw) cloned in E. coli. Nature. 1979;281:646-650. [PubMed] [DOI] |
| 3. | Heermann KH, Goldmann U, Schwartz W, Seyffarth T, Baumgarten H, Gerlich WH. Large surface proteins of hepatitis B virus containing the pre-s sequence. J Virol. 1984;52:396-402. [PubMed] |
| 4. | Fujiyama A, Miyanohara A, Nozaki C, Yoneyama T, Ohtomo N, Matsubara K. Cloning and structural analyses of hepatitis B virus DNAs, subtype adr. Nucleic Acids Res. 1983;11:4601-4610. [PubMed] [DOI] |
| 5. | Mimms LT, Solomon LR, Ebert JW, Fields H. Unique preS sequence in a gibbon-derived hepatitis B virus variant. Biochem Biophys Res Commun. 1993;195:186-191. [PubMed] [DOI] |
| 6. | Chai N, Gudima S, Chang J, Taylor J. Immu-noadhesins containing pre-S domains of hepatitis B virus large envelope protein are secreted and inhibit virus infection. J Virol. 2007;81:4912-4918. [PubMed] [DOI] |
| 7. | Blanchet M, Sureau C. Analysis of the cytosolic domains of the hepatitis B virus envelope proteins for their function in viral particle assembly and infectivity. J Virol. 2006;80:11935-11945. [PubMed] [DOI] |
| 8. | Lambert C, Prange R. Posttranslational N-glycosylation of the hepatitis B virus large envelope protein. Virol J. 2007;4:45. [PubMed] [DOI] |
| 9. | Sureau C, Fournier-Wirth C, Maurel P. Role of N glycosylation of hepatitis B virus envelope proteins in morphogenesis and infectivity of hepatitis delta virus. J Virol. 2003;77:5519-5523. [PubMed] [DOI] |
| 11. | Fields S, Song O. A novel genetic system to detect protein-protein interactions. Nature. 1989;340:245-6. [PubMed] [DOI] |
| 12. | Osman A. Yeast two-hybrid assay for studying protein-protein interactions. Methods Mol Biol. 2004;270:403-422. [PubMed] |
| 13. | Miller J, Stagljar I. Using the yeast two-hybrid system to identify interacting proteins. Methods Mol Biol. 2004;261:247-262. [PubMed] |
| 14. | Wang H, Peters GA, Zeng X, Tang M, Ip W, Khan SA. Yeast two-hybrid system demonstrates that estrogen receptor dimerization is ligand-dependent in vivo. J Biol Chem. 1995;270:23322-23329. [PubMed] [DOI] |
| 15. | Legrain P, Selig L. Genome-wide protein interaction maps using two-hybrid systems. FEBS Lett. 2000;480:32-36. [PubMed] [DOI] |
| 16. | Walhout AJ, Vidal M. High-throughput yeast two-hybrid assays for large-scale protein interaction mapping. Methods. 2001;24:297-306. [PubMed] [DOI] |
| 17. | Maple J, Moller SG. Yeast two-hybrid screening. Methods Mol Biol. 2007;362:207-223. [PubMed] |
| 18. | Legrain P, Wojcik J, Gauthier JM. Protein--protein interaction maps: a lead towards cellular functions. Trends Genet. 2001;17:346-352. [PubMed] [DOI] |
| 19. | Vidalain PO, Boxem M, Ge H, Li S, Vidal M. Increasing specificity in high-throughput yeast two-hybrid experiments. Methods. 2004;32:363-370. [PubMed] [DOI] |
| 20. | Chen J, Chua HN, Hsu W, Lee ML, Ng SK, Saito R, Sung WK, Wong L. Increasing confidence of protein-protein interactomes. Genome Inform. 2006;17:284-297. [PubMed] |
| 21. | Uyttendaele I, Lemmens I, Verhee A, De Smet AS, Vandekerckhove J, Lavens D, Peelman F, Tavernier J. Mammalian protein-protein interaction trap (MAPPIT) analysis of STAT5, CIS, and SOCS2 interactions with the growth hormone receptor. Mol Endocrinol. 2007;21:2821-2831. [PubMed] [DOI] |
| 22. | Matsuzawa S, Reed JC. Yeast and mammalian two-hybrid systems for studying protein-protein interactions. Methods Mol Biol. 2007;383:215-225. [PubMed] |
| 23. | Lee JW, Lee SK. Mammalian two-hybrid assay for detecting protein-protein interactions in vivo. Methods Mol Biol. 2004;261:327-336. [PubMed] |
| 24. | Rodi DJ, Makowski L, Kay BK. One from column A and two from column B: the benefits of phage display in molecular-recognition studies. Curr Opin Chem Biol. 2002;6:92-96. [PubMed] [DOI] |
| 25. | Sidhu SS, Koide S. Phage display for engineering and analyzing protein interaction interfaces. Curr Opin Struct Biol. 2007;17:481-487. [PubMed] [DOI] |
| 26. | Sergeeva A, Kolonin MG, Molldrem JJ, Pasqualini R, Arap W. Display technologies: application for the discovery of drug and gene delivery agents. Adv Drug Deliv Rev. 2006;58:1622-1654. [PubMed] [DOI] |
| 27. | Hust M, Dubel S, Schirrmann T. Selection of recombinant antibodies from antibody gene libraries. Methods Mol Biol. 2007;408:243-255. [PubMed] |
| 28. | Rojas G, Talavera A, Munoz Y, Rengifo E, Krengel U, Angstrom J, Gavilondo J, Moreno E. Light-chain shuffling results in successful phage display selection of functional prokaryotic-expressed antibody fragments to N-glycolyl GM3 ganglioside. J Immunol Methods. 2004;293:71-83. [PubMed] [DOI] |
| 29. | Berggard T, Linse S, James P. Methods for the detection and analysis of protein-protein interactions. Proteomics. 2007;7:2833-2842. [PubMed] [DOI] |
| 30. | Masters SC. Co-immunoprecipitation from transfected cells. Methods Mol Biol. 2004;261:337-350. [PubMed] |
| 31. | Elion EA, Wang Y. Making protein immuno-precipitates. Methods Mol Biol. 2004;284:1-14. [PubMed] |
| 32. | Tanowitz M, von Zastrow M. Identification of protein interactions by yeast two-hybrid screening and coimmunoprecipitation. Methods Mol Biol. 2004;259:353-369. [PubMed] |
| 33. | Brymora A, Valova VA, Robinson PJ. Protein-protein interactions identified by pull-down experiments and mass spectrometry. Curr Protoc Cell Biol. 2004;Chapter 17:Unit 17.5. [PubMed] |
| 34. | Nagaya T, Seo H. Functional analysis of nuclear hormone receptors. Nippon Rinsho. 1998;56:1688-1692. [PubMed] |
| 35. | Kishimoto A, Ogura T, Esumi H. A pull-down assay for 5' AMP-activated protein kinase activity using the GST-fused protein. Mol Biotechnol. 2006;32:17-21. [PubMed] [DOI] |
| 36. | Craig TJ, Ciufo LF, Morgan A. A protein-protein binding assay using coated microtitre plates: increased throughput, reproducibility and speed compared to bead-based assays. J Biochem Biophys Methods. 2004;60:49-60. [PubMed] [DOI] |
| 37. | Ledee DR, Tripathi BK, Zelenka PS. The CDK5 activator, p39, binds specifically to myosin essential light chain. Biochem Biophys Res Commun. 2007;354:1034-1039. [PubMed] [DOI] |
| 38. | Liu JP, Liu NS, Yuan HY, Guo Q, Lu H, Li YY. Human homologue of SETA binding protein 1 interacts with cathepsin B and participates in TNF-Induced apoptosis in ovarian cancer cells. Mol Cell Biochem. 2006;292:189-195. [PubMed] [DOI] |
| 39. | Swick L, Kapatos G. A yeast 2-hybrid analysis of human GTP cyclohydrolase I protein interactions. J Neurochem. 2006;97:1447-1455. [PubMed] [DOI] |
| 40. | 蔺 淑梅, 张 树林, 成 军, 刘 敏, 王 琳, 王 建军, 杨 倩, 黄 燕萍, 白 桂芹. 肝细胞cDNA文库中乙型肝炎病毒前-前-S蛋白结合蛋白基因筛选. 世界华人消化杂志. 2004;12:2907-2910. [DOI] |
| 41. | Ryu CJ, Cho DY, Gripon P, Kim HS, Guguen-Guillouzo C, Hong HJ. An 80-kilodalton protein that binds to the pre-S1 domain of hepatitis B virus. J Virol. 2000;74:110-116. [PubMed] |
| 43. | Li D, Wang XZ, Ding J, Yu JP. NACA as a potential cellular target of hepatitis B virus preS1 protein. Dig Dis Sci. 2005;50:1156-1160. [PubMed] [DOI] |
| 47. | Deng Q, Zhuang M, Kong YY, Xie YH, Wang Y. Screening for PreS specific binding ligands with a phage displayed peptides library. World J Gastroenterol. 2005;11:4018-4023. [PubMed] |
| 48. | Deng Q, Zhai JW, Michel ML, Zhang J, Qin J, Kong YY, Zhang XX, Budkowska A, Tiollais P, Wang Y. Identification and characterization of peptides that interact with hepatitis B virus via the putative receptor binding site. J Virol. 2007;81:4244-4254. [PubMed] [DOI] |
| 50. | 李 志群, 马 英骥, 成 军. 酵母双杂交技术筛选肝细胞中与羧基末端截短型乙型肝炎表面抗原中蛋白MHBst167蛋白结合蛋白的研究. 胃肠病学和肝病学杂志. 2006;15: 138-140. |
| 51. | Toh QC, Tan TL, Teo WQ, Ho CY, Parida S, Chen WN. Identification of cellular membrane proteins interacting with hepatitis B surface antigen using yeast split-ubiquitin system. Int J Med Sci. 2005;2:114-117. [PubMed] |
| 52. | Tan WS, Tan GH, Yusoff K, Seow HF. A phage-displayed cyclic peptide that interacts tightly with the immunodominant region of hepatitis B surface antigen. J Clin Virol. 2005;34:35-41. [PubMed] [DOI] |
| 53. | Bai GQ, Cheng J, Zhang SL, Huang YP, Wang L, Liu Y, Lin SM. Screening of hepatocyte proteins binding to complete S protein of hepatitis B virus by yeast-two hybrid system. World J Gastroenterol. 2005;11:3899-3904. [PubMed] |