修回日期: 2008-04-01
接受日期: 2008-05-20
在线出版日期: 2008-06-08
恶性肿瘤严重危害着人类的健康. 尽管肿瘤的病因学研究取得了一定进展, 肿瘤的临床处置依然面临挑战, 包括手术切除、化学药物治疗和放射治疗在内的标准治疗模式不能对所有患者都奏效. 如何选择合适的治疗方案和准确评估患者的预后, 临床医生经常感到困惑. 针对信号通路重要蛋白的分子靶向治疗的结果显示, 不同肿瘤患者具有独特的分子表型特征并决定了肿瘤治疗的疗效. 有效治疗肿瘤需要量体裁衣式地设计出个体化的治疗方案. 通过"组"学研究发现了许多肿瘤细胞特异的分子表型, 而针对这些表型的个体化治疗将是未来肿瘤治疗的方向.
引文著录: 何平, 赵晓航. 细胞重要分子表型与肿瘤个体化治疗的研究进展. 世界华人消化杂志 2008; 16(16): 1781-1787
Revised: April 1, 2008
Accepted: May 20, 2008
Published online: June 8, 2008
Malignant tumor impairs human's health severely. Although progresses have been made in tumor's etiology, clinical management of tumor is still confronted by challenges. The standard treatments against tumor, including surgery, chemotherapy and radiotherapy, are not effective for all patients. The clinicians often feel confused while selecting appropriate therapeutic pattern and judging patients' prognosis. The clinical outcome of target-protein-based cancer therapy revealed that personalized molecular signature determined individualized curative effect. Effective management of tumor needs patient-tailored design. The so-called "omic" researches accelerate the studies in specific molecule phenotype of tumor cells, and the personalized therapy against such individualized malignant phenotype will represent the future direction in tumor treatment.
- Citation: He P, Zhao XH. Advances in tumor-related molecular phenotypes and personalized therapy. Shijie Huaren Xiaohua Zazhi 2008; 16(16): 1781-1787
- URL: https://www.wjgnet.com/1009-3079/full/v16/i16/1781.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i16.1781
肿瘤已成为我国城镇人口第一死因, 严重危害人类健康. 尽管对肿瘤的病因学研究和以手术切除、放射治疗和化学药物治疗为主的综合治疗取得一定进展, 肿瘤治疗仍然面临巨大挑战[1]. 对不同肿瘤类型和病理分期的个体采用相同的治疗方案也许是疗效不佳的原因之一, 个体差异与疗效的关系越来越受到临床医生和研究者的重视[2-6]. 如果肿瘤治疗能够做到"因人而异、量体裁衣"似的个体化, 将能提高疗效、避免过度治疗和降低患者经济负担, 减少医疗资源的浪费[7-8]. 研究发现, 肿瘤细胞信号传导通路及其调节网络存在个体化差异[9-15]. 而这种个体癌细胞分子水平的差异决定了治疗效果的差异, 所以根据肿瘤个体分子表型的特征选择治疗方案可能是个体化治疗的一个方向. 高通量的"组学"技术适合全面的描述个体分子表型并发现肿瘤治疗的"新靶点", 针对细胞异常信号传导通路中关键作用分子的肿瘤靶向治疗逐渐呈现个体化趋势[16-18].
细胞信号传导通路是由一系列级联放大的酶促反应被激活的. 激酶底物蛋白的磷酸化是信号传递的驱动力. 由此, 阐明特定细胞信号通路的激活程度, 了解该信号通路中关键激酶底物被磷酸化的状态十分重要[19]. 研究证实, 细胞信号传导通路的异常活化是导致肿瘤发生发展的重要原因之一[20]. 通过比较肿瘤患者样本不同时间点、不同病程阶段、治疗前后的关键激酶底物磷酸化水平, 可以了解特定细胞信号传导通路中关键激酶活性水平, 间接了解个体生理或病理状态. 因此, 了解肿瘤细胞内关键细胞信号传导通路功能状态的网络图, 即关键靶点分子的表达水平和磷酸化程度, 将会成为肿瘤个体化治疗的一种新依据[9].
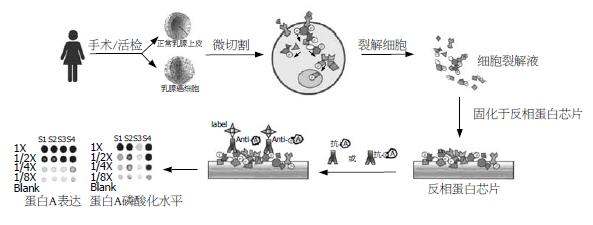
Petricoin et al[21]分析了45例乳腺癌和9例良性乳腺病患者的癌组织和正常乳腺上皮中激酶底物磷酸化水平及其信号传导通路的磷酸化状态. 乳腺癌手术切除或乳腺细针穿刺活检取材样本, 经显微切割获取癌细胞和正常乳腺上皮. 全细胞裂解蛋白按不同稀释度被固化于反相蛋白质芯片上. 用抗特定激酶底物蛋白抗体和抗该蛋白磷酸化抗体与固化于芯片上的靶蛋白杂交, 同时检测这些信号传导通路中特定激酶底物的表达和磷酸化水平(图1). 如图所示, 蛋白A在4个样本(S1、S2、S3和S4)中表达水平是一致的, 但磷酸化程度不同, 在样本3(S3)中蛋白A的磷酸化水平明显低于来自其他个体的样本. 结果显示, 细胞信号传导通路的活跃程度存在显著个体差异; 每个个体的肿瘤细胞呈现各不相同的激酶活性及其相应被活化的信号通路. 尽管慢性粒细胞性白血病、间质瘤等某些肿瘤存在共同的细胞信号通路异常活化或功能障碍, 如c-kit家族信号传递异常活化[22-23], 但多数散发的乳腺癌和某些上皮来源性恶性肿瘤是由复杂的、各不相同的细胞信号传导网络失调所引起的.
Irish et al[24]对急性粒细胞白血病(acute myeloid leukemia, AML)化疗前、后细胞样本做了多参数流式细胞学分析, 发现不同个体白血病细胞中STAT和MAPK等细胞重要信号传导通路的激酶底物磷酸化程度不尽相同. 急性粒细胞白血病中常见两条平行的信号通路的异常活化[25], 即STAT通路[26]和Ras/MAPK通路[27]. 研究发现, STATs, 如STAT3和STAT5, 在AML中组成性活化[28-29], 但是在AML患者的癌细胞中还未发现基础STAT磷酸化水平与白血病发生之间的因果联系. fms样酪氨酸激酶3(fms-like tyrosine kinase 3, Flt3)是以上2条通路的上游分子[30], 其异常改变见于30%的AML患者, 而且Flt3还是AML不良预后的重要指示分子[31]. 在细胞系中已发现突变且活化的Flt3可以激活STAT和Ras/MAPK通路[32]. 然而, 也有研究发现Stat5磷酸化的基础水平在Flt3野生型和突变型的AML患者癌细胞并无统计学差异[33]. 细胞系与患者原始样本之间的相反观察结果提示, 关键蛋白在信号传递网络中的作用模式而非绝对表达量(或是基础磷酸化状态)更能反应癌症的信号传递状态. 基于以上考虑, Irish et al以已知发生Flt3突变和其他细胞遗传学异常改变的AML患者为研究对象, 应用细胞内磷酸化特异流式细胞仪检测来源于患者外周血白血病细胞内的多个磷酸化、活化信号传递分子, 并绘制磷酸化蛋白功能网络图. 结果发现, AML不同遗传和临床表型有着不同的磷酸化蛋白信号传导网络特征. 根据这种特征可以划分同一个体的不同癌细胞亚群, 可以将AML患者分为对化疗反应和预后显著不同的四个亚类, 并最终确定AML中与Flt3突变、其他遗传学改变、细胞表面标志物CD15表达以及对化疗敏感表型密切相关的磷酸化蛋白表达谱. 由此可见, 肿瘤细胞信号传导异常具有个性化特征.
随着对肿瘤细胞异常信号传导通路的了解, 那些在肿瘤细胞中起关键调节作用的分子逐渐成为肿瘤靶向治疗潜在的"靶标", 例如细胞增殖、分化、迁移、血管形成和细胞周期调节的关键调节因子[34-35]. 针对这些信号通路关键分子的肿瘤靶向药物按照化学性质通常可分为单克隆抗体类药物和小分子抑制剂类药物. 单克隆抗体类药物可以直接和间接地杀伤肿瘤细胞. 抗体直接阻断或激活靶信号分子及其受体, 抑制细胞周期或DNA修复[36], 抑制血管形成[37], 增强受体内吞[38]. 同时, 抗体类药物还可以通过激活免疫效应细胞, 通过补体依赖的细胞毒性反应(complement-dependent cytotoxicity, CDC)和抗体依赖的细胞毒性反应(antibody-dependent cellular cytotoxicity, ADCC)间接清除肿瘤细胞[39]. 此类药物根据其人源化的程度又可分为嵌合抗体、人源化抗体和完全人抗体. 小分子抑制物可直接抑制信号传导通路中关键分子的活性, 从而抑制肿瘤生长. 其中最常见的是蛋白酪氨酸激酶的小分子抑制物, 他们大多是ATP的类似物, 可竞争性地结合激酶, 抑制激酶的活性[40]. 表1列举了目前应用于肿瘤临床治疗的主要分子靶向药物.
| 药物名称 | 商品名 | 分子靶点 | 药物性质 | 针对肿瘤类型 |
| 西妥昔(cetuximab) | 爱必妥(Erbitux) | EGFR | mAb | 结直肠癌、头颈鳞状细胞癌 |
| 曲妥珠(trastuzumab) | 赫赛汀(Herceptin) | ERBB2 | mAb | 乳腺癌 |
| 贝伐(bevacizumab) | 阿瓦斯丁(Avastin) | VEGF | mAb | 结直肠癌 |
| 利妥昔(rituximab) | 美罗华(Rituxan) | CD20 | mAb | B细胞淋巴瘤 |
| 替伊莫(ibritumomab tiuxetan) | 泽娃灵(Zevalin) | CD20 | mAb | B细胞淋巴瘤 |
| 托西莫(tositumomab-I131) | Bexxar | CD20 | mAb | B细胞淋巴瘤 |
| 吉妥(gemtuzumab ozogamicin) | 麦罗塔(Mylotarg) | CD33 | mAb | 急性粒细胞白血病 |
| alemtuzumab | 坎帕斯( Campath) | CD52 | mAb | B细胞慢性淋巴细胞白血病 |
| 伊马替尼(imatinib mesylate) | 格列卫(Gleevec) | BCR-ABL、KIT、PDGFR | SMI | 胃肠间质瘤、慢性粒细胞白血病 |
| 吉非替尼(gefitinib) | 易瑞沙(Iressa) | EGFR | SMI | 非小细胞肺癌 |
| 艾罗替尼(erlotinib) | 特罗凯(Tarceva) | EGFR | SMI | 非小细胞肺癌、胰腺癌 |
| 舒尼替尼(sunitinib) | Sutent | VEGFR、KIT、PDGFR、FLT3 | SMI | 胃肠间质瘤、肾癌 |
| 索拉非尼(sorafenib) | 多吉美(Nexavar) | B-Raf、EGFR、VEGFR2、PDGFR | SMI | 肾癌 |
| 硼替佐米(bortezomib) | 万珂(Velcade) | 28S protease | SMI | 多发性骨髓瘤 |
临床研究提示, 对于特定肿瘤患者, 应用这种针对特异分子靶点的靶向治疗通常能够有效地控制肿瘤, 但是也存在对此类药物不敏感的肿瘤个体. 表皮生长因子受体(epidermal growth factor receptor, EGFR)的异常活化与多种肿瘤的发生、发展密切相关[41-43]. EGFR的酪氨酸激酶结构域是其活性所必需的, 针对这一结构域的小分子特异抑制物-吉非替尼(gefitinib)和艾罗替尼(erlotinib)已被应用于晚期非小细胞肺癌(non-small-cell lung cancer, NSCL)的临床治疗[34-35,39-40]. 然而, 80%以上的非小细胞肺癌对这两种抑制剂并无反应[44-47], 提示只有部分人群肿瘤细胞内存在EGFR的异常传导通路[48]. 研究提示, EGFR基因突变[49-50]、扩增[46,51-52]以及EGFR蛋白表达升高[52]与吉非替尼和艾罗替尼的药物敏感性呈正相关[53-55]. 同时, K-ras基因的突变也与以上两种药物的抵抗性呈正相关[56]. 然而, 也有研究发现, EGFR基因的突变与艾罗替尼的药物疗效无关[46]. 此外, 发现对于EGFR基因突变几率低的男性、吸烟和肺鳞癌人群, 艾罗替尼也可以延长患者的总体生存时间[57]. 这种看似矛盾的研究结果提示, 个体化分子特征决定个体疗效. 实际上, 每一个肿瘤个体都存在独特的疾病相关分子表达谱[10,15], 某类治疗方案可能仅对部分存在特异分子靶点的个体有效. 因此, 针对肿瘤个体特定分子表达谱的个体化治疗(personalized therapy)是肿瘤靶向治疗的方向[6].
"组学"技术是研究生物系统功能状态的有力工具, 应用该技术可以综合评价肿瘤患者的个体差异. 以基因芯片为代表的基因组学技术可分析肿瘤个体的基因表达谱, 结合病理分期和生存资料将患者分成几个亚类[58-59], 便于进行有针对性的个体化治疗. 然而, 基因转录水平的改变不能完全反映蛋白质表达水平的异常, 尤其是蛋白质的翻译后修饰、蛋白质相互作用和细胞信号传导通路的改变. 通过蛋白质组学分析, 可以展示特定细胞表达的全部蛋白质及其功能状态, 通过检测信号通路中重要功能蛋白的表达和活性, 区分肿瘤细胞特定分子表型与肿瘤靶向治疗的疗效关系, 以实现肿瘤治疗的个体化[60-64].
Taguchi et al[65]应用基质辅助激光解吸质谱(matrix assisted laser desorption/Ionization-mass spectrometry, MALDI-MS)研究了吉非替尼/艾罗替尼治疗非小细胞肺癌的敏感性, 从外周血分离、鉴定了一种基于8个特征性蛋白谱型, 该蛋白谱型能够区分对EGFR抑制剂治疗敏感的人群. 研究分为建立模型和验证两阶段. 首先, 采集139例未接受吉非替尼或艾罗替尼治疗的非小细胞肺癌外周血样本, 进行MALDI-MS分析. 从两个研究所分别采集了原始质谱数据, 经分析得到可预测临床结局的基于8个特征谱型的算法. 这些特征谱型用于区分两个研究所用药后不同临床结局人群的一致性为97.1%. 进而, 用已建立模型分别验证67和96个接受吉非替尼或艾罗替尼治疗的两组患者和158例未接受该药物治疗的患者(对照组). 吉非替尼治疗组病例被预测为"良好预后"和"不良预后"的生存时间分别是207 d和92 d; 艾罗替尼治疗组患者被预测为"良好预后"和"不良预后"的生存时间分别是306 d和107 d. 未接受治疗的对照组, 这8个特征谱型预测为"良好预后"和"不良预后"患者的生存时间无显著差异. 因此, 这种算法可以区分对小分子抑制剂吉非替尼和艾罗替尼有无反应的人群、判断预后, 在治疗前预测哪些人接受吉非替尼/艾罗替尼治疗后可能有良好的疗效和预后, 指导临床医师有针对性地用药.
Okano et al[66]应用双向差异凝胶电泳(two dimensional difference gel electrophoresis, 2D-DIGE)分析了对吉非替尼敏感和不敏感肺腺癌组织差异表达蛋白. 共鉴定87个差异蛋白, 经过数据分析确定其中9个蛋白的组合可以准确区分对吉非替尼敏感和不敏感的肺腺癌人群. 心型脂肪酸结合蛋白经酶联免疫吸附试验(ensyme linked immunosorbent assay, ELISA)验证在对吉非替尼不敏感的肺腺癌人群中表达下降. 由于EGFR的突变与吉非替尼敏感性有关[49-50], 该研究小组还分析了这些差异蛋白与EGFR突变的相关性, 确定了与EGFR突变紧密相关的12个蛋白组合. 这些差异蛋白有可能成为对肺腺癌吉非替尼靶向治疗疗效预测的候选标志分子, 从而指导吉非替尼的个体化用药.
抗神经母细胞瘤的靶向治疗中广泛应用了拓扑异构酶抑制剂依托泊苷(etoposide). 拓扑异构酶可逆地与DNA结合, 改变DNA三级结构, 在DNA复制过程中发挥重要作用[67]. 依托泊苷是拓扑异构酶Ⅱ的选择性抑制剂, 通过干扰其介导的DNA再连接而稳定拓扑异构酶Ⅱ-断裂DNA复合物, 从而抑制DNA复制[68]. 依托泊苷的耐药性主要源于药物诱导的肿瘤细胞凋亡能力的下降. 应用二维电泳(two dimensional electrophoresis, 2DE)技术, Urbani et al[69]在依托泊苷耐药的神经母细胞瘤细胞中发现一组高表达蛋白, 如peroxiredoxin 1、β半乳糖苷可溶性凝集素结合蛋白、Vimentin、热休克蛋白27和异质性核糖核蛋白K. 此外, 在依托泊苷耐药细胞克隆中还发现了表达水平明显下降的蛋白, 即dUTP焦磷酸酶. 这些蛋白抵抗依托泊苷的作用机制还不十分清楚, 也可能是药物诱导的产物, 他们有望成为预测神经母细胞瘤化疗敏感性的候选标志分子. Shin et al[70]用类似的差异蛋白质组学技术筛查了对5氟尿嘧啶(5-FU)化疗敏感/不敏感的结肠癌细胞的差异蛋白表达谱. 发现, 在对5-FU不敏感的结肠癌细胞中线粒体F1F0-ATP合成酶的α亚基表达明显减少. 进一步分析发现在5-FU不敏感的结肠癌细胞中ATP合成酶亚基、ATP合成酶活性以及细胞内ATP含量都有所下降. 提示ATP合成酶的下调可能会导致细胞对5-FU化疗不敏感, 因此, 对于组织中ATP合成酶低表达的结肠癌来说, 5-FU化疗可能不是首选的化疗方案.
Allal et al[71]用比较蛋白质组学技术策略从17例直肠癌患者肿瘤组织分离、鉴定了放疗抵抗相关蛋白. 首先, 于治疗前采集直肠癌组织标本, 然后进行术前放疗, 放疗6 wk后患者接受手术治疗与临床反应评估. 从对治疗敏感或抵抗的肿瘤组织提取的总蛋白, 经2DE分离和MALDI-TOF-MS/MS串联质谱鉴定, 获得差异表达蛋白. 其中, 角蛋白Ⅰ型蛋白、notch 2蛋白类似物以及DNA修复蛋白RAD51L3等蛋白的差异表达与放疗敏感性相关; 而tropomodulin、热休克蛋白42、β-tubulin、annexin V和calsenilin等则与放疗抵抗相关. 所鉴定的这些对放疗敏感或抵抗相关蛋白及其组合不但可以增加对放疗杀伤肿瘤细胞分子机制的认识, 还可以在放疗前辅助选择适应治疗的个体, 避免无效和/或过度治疗.
肿瘤生物学的个体化差异决定了肿瘤靶向治疗的个体化. 只有从每个肿瘤患者的个体入手, 分析其个体特性, 有针对性的"量体裁衣", 才是肿瘤治疗有效和经济的途径. 肿瘤细胞信号传导异常具有个性化特征, 个体化治疗疗效优于传统的单一治疗模式. 然而, 肿瘤个体化治疗是肿瘤治疗的理想境界. 达到这个目标, 在今天看来并非易事, 可能的原因有: 缺乏检测个体化分子特征的工具, 缺乏对潜在指示个体化疗效分子标志的后期验证, 无法合理解释这些标志在肿瘤发生发展过程中发挥的确切作用, 缺乏利用这些个体化分子特征进一步指导临床治疗的经验等. 其中重要的原因是人类目前还远没有搞清楚细胞癌变的分子机制. 随着生物学研究技术的改进和人类对肿瘤发生发展机制认识的不断深入, 肿瘤治疗将会逐步接近个体化.
实际上, 在手术切除、放射治疗和化学药物治疗这3种治疗肿瘤的方法中, 最能体现针对细胞重要表型进行个体化干预的治疗手段是药物治疗. 其理想的治疗模式是: 临床医师在用药前采集肿瘤患者的体液标本(包括血液、脑脊液和尿液等)并通过手术或穿刺技术采集肿瘤组织; 科研人员通过基因组学、蛋白质组学或代谢组学研究确认个体化的分子表型, 并将信息反馈给临床医师; 最后临床医师将这些个体化的分子表型信息与患者的临床症状、影像学和病理学的报告结果结合起来, 确定适合不同个体自身情况的用药方案.
肿瘤严重危害人类健康. 尽管对肿瘤的病因学研究和综合治疗取得一定进展, 肿瘤临床处置依然面临巨大挑战. 对不同肿瘤类型和病理分期的个体采用相同的治疗方案也许是疗效不佳的原因之一.
李淑德, 主任医师, 中国人民解放军第二军医大学长海医院消化内科; 黄文林, 教授, 中山大学附属肿瘤医院, 中山大学肿瘤防治中心
目前肿瘤个体化研究的热点是应用高通量的"组"学技术描绘个体化的分子特征. 针对这些特征设计个体化靶向治疗方案, 并应用于肿瘤的临床治疗.
Marko-Varga et al综述了目前最先进的蛋白质组学研究方法的特点, 并进一步介绍这些技术方法进行个体化治疗的实例. 提出建立广泛的统计分析可以更好地理解个体差异的来源(如蛋白表达谱的差异), 有助于对临床结局做出正确的判断.
本文根据国内外最新资料, 介绍针对肿瘤患者个体化分子表型的靶向治疗最新进展, 提出建立在个体化分子特征基础上的用药模式.
针对肿瘤个体化分子表型的个体化治疗将是未来肿瘤治疗的方向之一.
反相蛋白质芯片(RPA): 是将待检测的样品固化在芯片的每个点上, 这样一张芯片可以同时固化成百上千的不同患者的样品或细胞裂解液. 在该体系中, 每张芯片一次同一种检测蛋白(如抗体)杂交, 该蛋白同时在不同的样品得以检测, 从而在不同样品间进行平行比较.
本文选题紧密结合临床亟待解决的难题, 起点高, 论证较充分, 参考文献新, 行文流畅, 具有较好的可读性.
编辑: 李军亮 电编:何基才
| 2. | Calvo KR, Liotta LA, Petricoin EF. Clinical proteomics: from biomarker discovery and cell signaling profiles to individualized personal therapy. Biosci Rep. 2005;25:107-125. [PubMed] [DOI] |
| 3. | Verrills NM. Clinical proteomics: present and future prospects. Clin Biochem Rev. 2006;27:99-116. [PubMed] |
| 4. | Jain KK. Personalized medicine. Curr Opin Mol Ther. 2002;4:548-558. [PubMed] |
| 5. | Petricoin E, Wulfkuhle J, Espina V, Liotta LA. Clinical proteomics: revolutionizing disease detection and patient tailoring therapy. J Proteome Res. 2004;3:209-217. [PubMed] [DOI] |
| 6. | Liotta LA, Kohn EC, Petricoin EF. Clinical proteomics: personalized molecular medicine. JAMA. 2001;286:2211-2214. [PubMed] [DOI] |
| 7. | Dalton WS, Friend SH. Cancer biomarkers--an invitation to the table. Science. 2006;312:1165-1168. [PubMed] [DOI] |
| 9. | Petricoin EF, Zoon KC, Kohn EC, Barrett JC, Liotta LA. Clinical proteomics: translating benchside promise into bedside reality. Nat Rev Drug Discov. 2002;1:683-695. [PubMed] [DOI] |
| 10. | Liotta L, Petricoin E. Molecular profiling of human cancer. Nat Rev Genet. 2000;1:48-56. [PubMed] [DOI] |
| 11. | Paweletz CP, Charboneau L, Bichsel VE, Simone NL, Chen T, Gillespie JW, Emmert-Buck MR, Roth MJ, Petricoin III EF, Liotta LA. Reverse phase protein microarrays which capture disease progression show activation of pro-survival pathways at the cancer invasion front. Oncogene. 2001;20:1981-1989. [PubMed] [DOI] |
| 12. | VanMeter A, Signore M, Pierobon M, Espina V, Liotta LA, Petricoin EF 3rd. Reverse-phase protein microarrays: application to biomarker discovery and translational medicine. Expert Rev Mol Diagn. 2007;7:625-633. [PubMed] [DOI] |
| 13. | Speer R, Wulfkuhle J, Espina V, Aurajo R, Edmiston KH, Liotta LA, Petricoin EF 3rd. Development of reverse phase protein microarrays for clinical applications and patient-tailored therapy. Cancer Genomics Proteomics. 2007;4:157-164. [PubMed] |
| 14. | Liotta LA, Espina V, Mehta AI, Calvert V, Rosenblatt K, Geho D, Munson PJ, Young L, Wulfkuhle J, Petricoin EF 3rd. Protein microarrays: meeting analytical challenges for clinical applications. Cancer Cell. 2003;3:317-325. [PubMed] [DOI] |
| 15. | Grubb RL, Calvert VS, Wulkuhle JD, Paweletz CP, Linehan WM, Phillips JL, Chuaqui R, Valasco A, Gillespie J, Emmert-Buck M. Signal pathway profiling of prostate cancer using reverse phase protein arrays. Proteomics. 2003;3:2142-2146. [PubMed] [DOI] |
| 16. | Smith L, Lind MJ, Welham KJ, Cawkwell L; Cancer Biology Proteomics Group. Cancer proteomics and its application to discovery of therapy response markers in human cancer. Cancer. 2006;107:232-241. [PubMed] [DOI] |
| 17. | Petricoin EF, Liotta LA. Clinical applications of proteomics. J Nutr. 2003;133:2476S-2484S. [PubMed] |
| 18. | Wulfkuhle JD, Edmiston KH, Liotta LA, Petricoin EF 3rd. Technology insight: pharmacoproteomics for cancer--promises of patient-tailored medicine using protein microarrays. Nat Clin Pract Oncol. 2006;3:256-268. [PubMed] [DOI] |
| 19. | Thor AD, Liu S, Edgerton S, Moore D 2nd, Kasowitz KM, Benz CC, Stern DF, DiGiovanna MP. Activation (tyrosine phosphorylation) of ErbB-2 (HER-2/neu): a study of incidence and correlation with outcome in breast cancer. J Clin Oncol. 2000;18:3230-3239. [PubMed] |
| 21. | Petricoin EF 3rd, Bichsel VE, Calvert VS, Espina V, Winters M, Young L, Belluco C, Trock BJ, Lippman M, Fishman DA, Sgroi DC, Munson PJ, Esserman LJ, Liotta LA. Mapping molecular networks using proteomics: a vision for patient-tailored combination therapy. J Clin Oncol. 2005;23:3614-3621. [PubMed] [DOI] |
| 22. | Hochhaus A. Imatinib mesylate (Glivec, Gleevec) in the treatment of chronic myelogenous leukemia (CML) and gastrointestinal stromal tumors (GIST). Ann Hematol. 2004;83 Suppl 1:S65-S66. [PubMed] |
| 23. | Nakajima M, Toga W. Tyrosine kinase inhibitor as a therapeutic drug for chronic myelogenous leukemia and gastrointestinal stromal tumor. Nippon Yakurigaku Zasshi. 2003;122:482-490. [PubMed] [DOI] |
| 24. | Irish JM, Hovland R, Krutzik PO, Perez OD, Bruserud Ø, Gjertsen BT, Nolan GP. Single cell profiling of potentiated phospho-protein networks in cancer cells. Cell. 2004;118:217-228. [PubMed] [DOI] |
| 25. | Stirewalt DL, Meshinchi S, Radich JP. Molecular targets in acute myelogenous leukemia. Blood Rev. 2003;17:15-23. [PubMed] [DOI] |
| 26. | Smithgall TE, Briggs SD, Schreiner S, Lerner EC, Cheng H, Wilson MB. Control of myeloid differentiation and survival by Stats. Oncogene. 2000;19:2612-2618. [PubMed] [DOI] |
| 27. | Platanias LC. Map kinase signaling pathways and hematologic malignancies. Blood. 2003;101:4667-4679. [PubMed] [DOI] |
| 28. | Xia Z, Baer MR, Block AW, Baumann H, Wetzler M. Expression of signal transducers and activators of transcription proteins in acute myeloid leukemia blasts. Cancer Res. 1998;58:3173-3180. [PubMed] |
| 29. | Benekli M, Xia Z, Donohue KA, Ford LA, Pixley LA, Baer MR, Baumann H, Wetzler M. Constitutive activity of signal transducer and activator of transcription 3 protein in acute myeloid leukemia blasts is associated with short disease-free survival. Blood. 2002;99:252-257. [PubMed] [DOI] |
| 30. | Gilliland DG, Griffin JD. Role of FLT3 in leukemia. Curr Opin Hematol. 2002;9:274-281. [PubMed] [DOI] |
| 31. | Gilliland DG, Griffin JD. The roles of FLT3 in hematopoiesis and leukemia. Blood. 2002;100:1532-1542. [PubMed] [DOI] |
| 32. | Hayakawa F, Towatari M, Kiyoi H, Tanimoto M, Kitamura T, Saito H, Naoe T. Tandem-duplicated Flt3 constitutively activates STAT5 and MAP kinase and introduces autonomous cell growth in IL-3-dependent cell lines. Oncogene. 2000;19:624-631. [PubMed] [DOI] |
| 33. | Pallis M, Seedhouse C, Grundy M, Russell N. Flow cytometric measurement of phosphorylated STAT5 in AML: lack of specific association with FLT3 internal tandem duplications. Leuk Res. 2003;27:803-805. [PubMed] [DOI] |
| 34. | Krause DS, Van Etten RA. Tyrosine kinases as targets for cancer therapy. N Engl J Med. 2005;353:172-187. [PubMed] [DOI] |
| 35. | Ciardiello F, Tortora G. EGFR antagonists in cancer treatment. N Engl J Med. 2008;358:1160-1174. [PubMed] [DOI] |
| 36. | Pietras RJ, Fendly BM, Chazin VR, Pegram MD, Howell SB, Slamon DJ. Antibody to HER-2/neu receptor blocks DNA repair after cisplatin in human breast and ovarian cancer cells. Oncogene. 1994;9:1829-1838. [PubMed] |
| 37. | Izumi Y, Xu L, di Tomaso E, Fukumura D, Jain RK. Tumour biology: herceptin acts as an anti-angiogenic cocktail. Nature. 2002;416:279-280. [PubMed] [DOI] |
| 38. | Harding J, Burtness B. Cetuximab: an epidermal growth factor receptor chemeric human-murine monoclonal antibody. Drugs Today (Barc). 2005;41:107-127. [PubMed] [DOI] |
| 39. | Imai K, Takaoka A. Comparing antibody and small-molecule therapies for cancer. Nat Rev Cancer. 2006;6:714-727. [PubMed] [DOI] |
| 40. | Baselga J. Targeting tyrosine kinases in cancer: the second wave. Science. 2006;312:1175-1178. [PubMed] [DOI] |
| 41. | Citri A, Yarden Y. EGF-ERBB signalling: towards the systems level. Nat Rev Mol Cell Biol. 2006;7:505-516. [PubMed] [DOI] |
| 42. | Hynes NE, Lane HA. ERBB receptors and cancer: the complexity of targeted inhibitors. Nat Rev Cancer. 2005;5:341-354. [PubMed] [DOI] |
| 43. | Bublil EM, Yarden Y. The EGF receptor family: spearheading a merger of signaling and therapeutics. Curr Opin Cell Biol. 2007;19:124-134. [PubMed] [DOI] |
| 44. | Fukuoka M, Yano S, Giaccone G, Tamura T, Nakagawa K, Douillard JY, Nishiwaki Y, Vansteenkiste J, Kudoh S, Rischin D. Multi-institutional randomized phase II trial of gefitinib for previously treated patients with advanced non-small-cell lung cancer (The IDEAL 1 Trial) [corrected]. J Clin Oncol. 2003;21:2237-2246. [PubMed] [DOI] |
| 45. | Kris MG, Natale RB, Herbst RS, Lynch TJ Jr, Prager D, Belani CP, Schiller JH, Kelly K, Spiridonidis H, Sandler A, Albain KS, Cella D, Wolf MK, Averbuch SD, Ochs JJ, Kay AC. Efficacy of gefitinib, an inhibitor of the epidermal growth factor receptor tyrosine kinase, in symptomatic patients with non-small cell lung cancer: a randomized trial. JAMA. 2003;290:2149-2158. [PubMed] [DOI] |
| 46. | Tsao MS, Sakurada A, Cutz JC, Zhu CQ, Kamel-Reid S, Squire J, Lorimer I, Zhang T, Liu N, Daneshmand M. Erlotinib in lung cancer - molecular and clinical predictors of outcome. N Engl J Med. 2005;353:133-144. [PubMed] [DOI] |
| 47. | Miller VA, Riely GJ, Zakowski MF, Li AR, Patel JD, Heelan RT, Kris MG, Sandler AB, Carbone DP, Tsao A. Molecular characteristics of bronchioloalveolar carcinoma and adenocarcinoma, bronchioloalveolar carcinoma subtype, predict response to erlotinib. J Clin Oncol. 2008;26:1472-1478. [PubMed] [DOI] |
| 48. | Dancey JE, Freidlin B. Targeting epidermal growth factor receptor--are we missing the mark? Lancet. 2003;362:62-64. [PubMed] [DOI] |
| 49. | Lynch TJ, Bell DW, Sordella R, Gurubhagavatula S, Okimoto RA, Brannigan BW, Harris PL, Haserlat SM, Supko JG, Haluska FG. Activating mutations in the epidermal growth factor receptor underlying responsiveness of non-small-cell lung cancer to gefitinib. N Engl J Med. 2004;350:2129-2139. [PubMed] [DOI] |
| 50. | Paez JG, Jänne PA, Lee JC, Tracy S, Greulich H, Gabriel S, Herman P, Kaye FJ, Lindeman N, Boggon TJ. EGFR mutations in lung cancer: correlation with clinical response to gefitinib therapy. Science. 2004;304:1497-1500. [PubMed] [DOI] |
| 51. | Cappuzzo F, Hirsch FR, Rossi E, Bartolini S, Ceresoli GL, Bemis L, Haney J, Witta S, Danenberg K, Domenichini I. Epidermal growth factor receptor gene and protein and gefitinib sensitivity in non-small-cell lung cancer. J Natl Cancer Inst. 2005;97:643-655. [PubMed] |
| 52. | Hirsch FR, Varella-Garcia M, Bunn PA Jr, Franklin WA, Dziadziuszko R, Thatcher N, Chang A, Parikh P, Pereira JR, Ciuleanu T, von Pawel J, Watkins C, Flannery A, Ellison G, Donald E, Knight L, Parums D, Botwood N, Holloway B. Molecular predictors of outcome with gefitinib in a phase III placebo-controlled study in advanced non-small-cell lung cancer. J Clin Oncol. 2006;24:5034-5042. [PubMed] [DOI] |
| 53. | Sharma SV, Bell DW, Settleman J, Haber DA. Epidermal growth factor receptor mutations in lung cancer. Nat Rev Cancer. 2007;7:169-181. [PubMed] [DOI] |
| 54. | Wheatley-Price P, Shepherd FA. Epidermal growth factor receptor inhibitors in the treatment of lung cancer: reality and hopes. Curr Opin Oncol. 2008;20:162-175. [PubMed] |
| 55. | Eberhard DA, Giaccone G, Johnson BE. Biomarkers of response to epidermal growth factor receptor inhibitors in Non-Small-Cell Lung Cancer Working Group: standardization for use in the clinical trial setting. J Clin Oncol. 2008;26:983-994. [PubMed] [DOI] |
| 56. | Pao W, Wang TY, Riely GJ, Miller VA, Pan Q, Ladanyi M, Zakowski MF, Heelan RT, Kris MG, Varmus HE. KRAS mutations and primary resistance of lung adenocarcinomas to gefitinib or erlotinib. PLoS Med. 2005;2:e17. [PubMed] [DOI] |
| 57. | Clark GM, Zborowski DM, Santabarbara P, Ding K, Whitehead M, Seymour L, Shepherd FA. Smoking history and epidermal growth factor receptor expression as predictors of survival benefit from erlotinib for patients with non-small-cell lung cancer in the National Cancer Institute of Canada Clinical Trials Group study BR.21. Clin Lung Cancer. 2006;7:389-394. [PubMed] |
| 58. | Bublil EM, Yarden Y. The EGF receptor family: spearheading a merger of signaling and therapeutics. Curr Opin Cell Biol. 2007;19:124-134. [PubMed] [DOI] |
| 59. | Rosenwald A, Wright G, Chan WC, Connors JM, Campo E, Fisher RI, Gascoyne RD, Muller-Hermelink HK, Smeland EB, Giltnane JM. The use of molecular profiling to predict survival after chemotherapy for diffuse large-B-cell lymphoma. N Engl J Med. 2002;346:1937-1947. [PubMed] [DOI] |
| 60. | Aguilar-Mahecha A, Hassan S, Ferrario C, Basik M. Microarrays as validation strategies in clinical samples: tissue and protein microarrays. OMICS. 2006;10:311-326. [PubMed] [DOI] |
| 61. | Azad NS, Rasool N, Annunziata CM, Minasian L, Whiteley G, Kohn EC. Proteomics in clinical trials and practice: present uses and future promise. Mol Cell Proteomics. 2006;5:1819-1829. [PubMed] [DOI] |
| 62. | Caprioli RM. Deciphering protein molecular signatures in cancer tissues to aid in diagnosis, prognosis, and therapy. Cancer Res. 2005;65:10642-10645. [PubMed] [DOI] |
| 63. | Petricoin EF, Liotta LA. Proteomic approaches in cancer risk and response assessment. Trends Mol Med. 2004;10:59-64. [PubMed] [DOI] |
| 64. | Hasan RK, Wulfkuhle JD, Liotta LA, Petricoin EF. Studentjama. Molecular technologies for personalized cancer management. JAMA. 2004;291:1644-1645. [PubMed] [DOI] |
| 65. | Taguchi F, Solomon B, Gregorc V, Roder H, Gray R, Kasahara K, Nishio M, Brahmer J, Spreafico A, Ludovini V. Mass spectrometry to classify non-small-cell lung cancer patients for clinical outcome after treatment with epidermal growth factor receptor tyrosine kinase inhibitors: a multicohort cross-institutional study. J Natl Cancer Inst. 2007;99:838-846. [PubMed] [DOI] |
| 66. | Okano T, Kondo T, Fujii K, Nishimura T, Takano T, Ohe Y, Tsuta K, Matsuno Y, Gemma A, Kato H. Proteomic signature corresponding to the response to gefitinib (Iressa, ZD1839), an epidermal growth factor receptor tyrosine kinase inhibitor in lung adenocarcinoma. Clin Cancer Res. 2007;13:799-805. [PubMed] [DOI] |
| 67. | Berger JM, Gamblin SJ, Harrison SC, Wang JC. Structure and mechanism of DNA topoisomerase II. Nature. 1996;379:225-232. [PubMed] [DOI] |
| 68. | Wang JC. DNA topoisomerases. Annu Rev Biochem. 1996;65:635-692. [PubMed] [DOI] |
| 69. | Urbani A, Poland J, Bernardini S, Bellincampi L, Biroccio A, Schnölzer M, Sinha P, Federici G. A proteomic investigation into etoposide chemo-resistance of neuroblastoma cell lines. Proteomics. 2005;5:796-804. [PubMed] [DOI] |
| 70. | Shin YK, Yoo BC, Chang HJ, Jeon E, Hong SH, Jung MS, Lim SJ, Park JG. Down-regulation of mitochondrial F1F0-ATP synthase in human colon cancer cells with induced 5-fluorouracil resistance. Cancer Res. 2005;65:3162-3170. [PubMed] |
| 71. | Allal AS, Kähne T, Reverdin AK, Lippert H, Schlegel W, Reymond MA. Radioresistance-related proteins in rectal cancer. Proteomics. 2004;4:2261-2269. [PubMed] [DOI] |