修回日期: 2008-03-01
接受日期: 2008-05-02
在线出版日期: 2008-05-08
目的: 观察SMYD3(SET and MYND-domain containing 3)基因沉默后c-Myc的表达及对HepG2细胞凋亡的影响.
方法: 构建针对SMYD3的shRNA干扰质粒Pgenesil-1-s1、Pgenesil-1-s2和阴性对照质粒Pgenesil-1-hk, 同时设空白对照组, 采用Lipofectamine2000脂质体介导转染法转染质粒. 转染后24、48、72 h, RT-PCR检测HepG2细胞SMYD3和c-Myc的表达情况. 流式细胞术法检测各组细胞的凋亡.
结果: SMYD3、c-Myc基因在HepG2细胞中强表达. RT-PCR显示Pgenesil-1-s1、Pgenesil-1-s2转染组与阴性对照质粒转染组Pgenesil-1-hk转染24、48、72 h后相比, SMYD3基因表达均明显受到抑制(F = 67.46, P<0.01; F = 176.79, P<0.01; F = 175.28, P<0.01), 同时c-Myc表达下调(三组之间: F = 11.58, P = 0.009; F = 126.41, P<0.01; F = 261.25, P<0.01). Pgenesil-1-s1、Pgenesil-1-s2转染组细胞早期凋亡率与Pgenesil-1-hk转染组(LSD-t = -13.58, -12.62, 均P<0.01)、空白组(LSD-t = -18.62, -17.67, 均P<0.01)相比有显著性差异.
结论: RNA干扰技术特异性沉默HepG2细胞SMYD3基因后, 抑制了c-Myc的表达, 促进了HepG2细胞的凋亡.
引文著录: 刘鑫, 陈立波, 叶进, 江军, 何军, 徐鋆耀, 钱伟. shRNA干扰SMYD3对肝癌细胞c-Myc表达及凋亡的影响. 世界华人消化杂志 2008; 16(13): 1373-1377
Revised: March 1, 2008
Accepted: May 2, 2008
Published online: May 8, 2008
AIM: To observe the c-Myc gene expression and apoptosis of HepG2 cells after SET and MYND-domain containing 3 (SMYD3) silence induced by short hairpin RNA.
METHODS: Three short hairpin RNA interference plasmids targeting SMYD3 were prepared as 3 groups: Pgenesil-1-s1 (with interfering effect), Pgenesil-1-s2 (with interfering effect), and Pgenesil-1-hk (negative control plasmid, without interfering effect). Meanwhile, an empty control group was also designed. Transfection was performed using the Lipofectmine2000 liposome. Reverse transcription-polymerase chain reaction (RT-PCR) was employed to detect the expression of SMYD3 and c-Myc gene 24, 48 and 72 h after transfection. Flow cytometry (FCM) was used to detect cell apoptosis in each group.
RESULTS: SMYD3 and c-Myc gene were strongly expressed in HepG2 cells. The expression of SMYD3 gene was significantly inhibited after 24-, 48- and 72-h transfection (F = 67.46, P < 0.01; F = 176.79, P < 0.01; F = 175.28, P < 0.01). At the same time, c-Myc gene mRNA expression was down-regulated in the Pgenesil-1 transfected group as compared with that in the Pgenesil-1-hk group (F = 11.58, P = 0.009; F = 126.41, P < 0.01; F = 261.25, P < 0.01). Moreover, the early apoptosis rate was significantly higher in the Pgenesil-1-s1 or Pgenesil-1-s2 group than that in the Pgenesil-1-hk group (LSD-t = -13.58, -12.62; both P < 0.01) and the empty control group (LSD-t = -18.62, -17.67; P < 0.01).
CONCLUSION: Short hairpin RNA interference targeting SMYD3 may inhibit the expression of c-Myc gene in HepG2 cells, thus promoting the apoptosis of HepG2 cells.
- Citation: Liu X, Chen LB, Ye J, Jiang J, Xu JY, Qian W. Effects of shRNA-induced SMYD3 silence on c-Myc expression and apoptosis of HepG2 cells. Shijie Huaren Xiaohua Zazhi 2008; 16(13): 1373-1377
- URL: https://www.wjgnet.com/1009-3079/full/v16/i13/1373.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i13.1373
组蛋白修饰指通过对组蛋白特定残基进行翻译后修饰, 如甲基化、乙酰化和磷酸化等, 调控染色体的结构、调节基因的转录激活和抑制等[1-3]. 组蛋白甲基化是组蛋白修饰的一个重要方式, 对基因转录的调控具有重要作用. 例如组蛋白H3的第9赖氨酸残基(H3K9)的甲基化具有抑制基因转录的作用, 而组蛋白H3的第4赖氨酸残基(H3K4)的甲基化具有激活基因转录的功能[4-6]. SMYD3是一种具有组蛋白甲基化功能的蛋白, 能够使H3K4发生2倍(di-)和3倍(tri-)的甲基化, 激活下游基因的转录, 能够抑制肿瘤细胞的凋亡、促进细胞增殖、促进细胞黏附和迁移等[7-9]. 我们根据RNA干扰原理[10-11], 以强表达SMYD3和c-Myc基因的HepG2细胞为研究对象, 设计、构建、筛选针对SMYD3基因的shRNA, 在体外研究肝癌细胞中SMYD3对c-Myc基因作用以及对肝癌细胞凋亡的影响, 探讨shRNA沉默SMYD3表达治疗肝癌的可行性.
肝癌细胞株HepG2购自中国科学院上海细胞库, 细胞在含100 mL/L小牛血清(美国Gibco公司)的DMEM培养基、20 mL/L CO2、37℃条件下培养. 分为Pgenesil-l-sl转染组、Pgenesil-1-s2和Pgenesil-1-hk, 空白组. 由徐鋆耀博士赠送构建好并经酶切鉴定的重组质粒Pgenesil-1-s1、Pgenesil-1-s2、Pgenesil-1-hk. 重组质粒图谱请参考文献[12]. Pgenesil-1-s1、Pgenesil-1-s2分别干扰SMYD3 mRNA 302-323、267-288之间核苷酸Pgenesil-1-s1正义5'-GATCCATACTGTAGTGCTAAGTGTTTCAAGACGACACACTTAGCACTACAGTATTTTTTTGTCGACA-3', 反义5'-AGCTTGTCGACAAAAAAATACTGTAGTGCTAAGTGTCGTCTTGAAACACTTAGCACTACAGTATG-3'; Pgenesil-l-s2正义5'-GATCCGCTGATGCGATGCTCTCAGTTCAAGACGCTGAGAGCATCGCATCAGCTTTTTTGTCGACA-3'. 反义5'-AGCTTGTCGACAAAAAAGCTGATGCGATGCTCTCAGCGTCTTGAACTGAGAGCATCGCATCAGCG-3'; Pgenesil-1-hk不针对任何SMYD3 mRNA序列, 正义: 5'-GATCCGACTTCATAAGGCGCATGCTTCAAGACGGCATGCGCCTTATGAAGTCTTTTTTGTCGACA-3', 反义: 5'-AGCTTGTCGACAAAAAAGACTTCATAAGGCGCATGCCGTCTTGAAGCATGCGCCTTATGAAGTCG-3'. 采用Lipofectamine2000脂质体(美国Invitrogen公司)进行转染, 转染前调整HepG2细胞密度为2×1010/L, 以每孔2 mL接种于6孔板, 待细胞增至50%时, 将质粒、脂质体复合物转染至细胞中, 具体操作参照说明书.
1.2.1 HepG2细胞中SMYD3和c-Myc基因mRNA表达的测定: 用TRIzol提取HepG2细胞的总mRNA, 逆转录反应: DEPC水5.5 μL, mRNA 2 μg, Oligo(dt)2 μg, dNTP(10 mmol/L)2.0 μL, MMLV(200 MU/L)1.0 μL, Rnasin(40 MU/L)0.5 μL, 5×Buffer 4.0 μL, 经42℃ 1 h, 95℃ 5 min反应后, 收集cDNA -20℃保存. SMYD3、c-Myc、β-actin引物用Primer5.0设计, 由上海英俊生物技术有限公司合成. SMYD3上游: 5'-TATCCTCCAGACTCCGTTCG-3', 下游: 5'-TCCATACTTGCTCATCACCAG-3', PCR产物为562 bp; c-Myc上游: 5'-GGGGCTTTATCTAACTCGCG-3', 下游: 5'-CTATGGGCAAAGTTTCGTG-3', PCR产物为222 bp; β-actin上游: 5'-GTGGACATCCGCAAAGAC-3'. 下游: 5'-AAAGGGTGTAACGCAACTAA-3', PCR产物为192 bp. PCR反应体系为: DEPC水16.4 μL、10×Buffer 2.5 μL, 25 mmol/L MgCl2 2.0 μL, dNTP(10 mmol/L)1.0 μL, cDNA 2.0 μL, Taq酶(83.35 mkat/L)0.5 μL, 上游引物、下游引物(50 umol/L)各0.3 μL. PCR仪进行反应, 参数设置为: 94℃ 5 min, 变性94℃ 30 s, 退火SMYD3、c-Myc均为53℃ 30 s、β-actin为56℃ 30 s, 延伸72℃ 30 s, 循环35次, 最后72℃延伸7 min. 分别取5 μL PCR产物加样至12 g/L琼脂糖凝胶上电泳, 用凝胶图像成像系统拍摄并分析结果.
1.2.2 HepG2细胞凋亡率的检测: 瞬时转染24 h后, 收集各组细胞, 加AnnexinⅤ-PE和7-AAD荧光染料(晶美生物工程有限公司), 具体操作按操试剂盒说明书进行. 1 h内流式细胞仪(FACSsort, Becton Dinkinson)检测细胞早期凋亡率.
统计学处理 数据以mean±SD表示, 采用SPSS13.0软件进行方差分析(ANOVA)LSD-t检验.
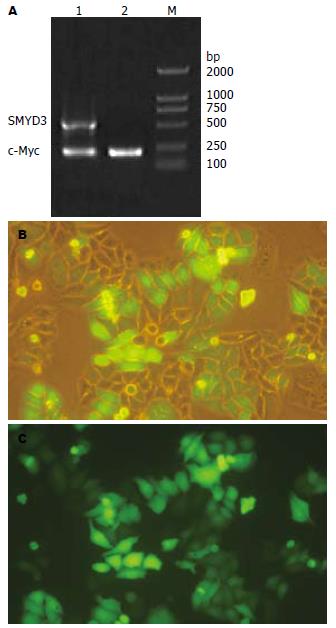
RT-PCR结果显示SMYD3、c-Myc基因在HepG2细胞中强表达(图1A)HepG2细胞转染24 h后荧光(图1B-C).
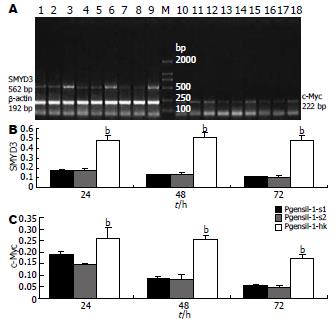
粒转染HepG2细胞24、48、72 h后, 分别提取RNA, 通过RT-PCR检测各组中两个基因的表达情况, 结果示: shRNA转染后, SMYD3基因表达均明显受抑, 同时c-Myc表达下调. 24、48、72 h各组SMYD3 mRNA及c-Myc mRNA的光密度值/β-actin mRNA的光密度值, 各时间点Pgenesil-1-s1组、Pgenesil-1-s2组SMYD3基因表达较Pgenesil-1-hk组均明显下调, 差异具有统计学意义(24 h: 三组之间, 总F = 67.46, s1与hk组LSD-t = -0.300, s2与hk组LSD-t = -0.300, P均<0.01; 48 h: 三组之间, 总F = 176.79, s1与hk组LSD-t = -0.378, s2与hk组LSD-t = -0.370, P均<0.01; 72 h: 三组之间, 总F = 175.28, s1与hk组LSD-t = -0.376, s2与hk组LSD-t = -0.380, P均<0.01); 各时间点Pgenesil-1-s1组、Pgenesil-1-s2组c-Myc基因表达较Pgenesil-1-hk组也都明显降低, 有统计学差异(24 h: 三组之间, 总F = 11.58, P = 0.009, s1与hk组LSD-t = -0.068, P = 0.029, s2与hk组LSD-t = -0.113, P = 0.003; 48 h: 三组之间, 总F = 126.41, s1与hk组LSD-t = -0.175, s2与hk组LSD-t = -0.178, P均<0.01; 72 h: 三组之间, 总F = 261.25, s1与hk组LSD-t = -0.117, s2与hk组LSD-t = -0.125, P均<0.01, 图2).
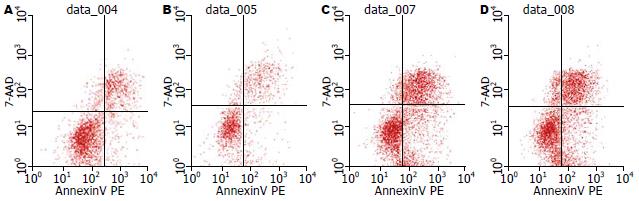
Pgenesil-1-s1、Pgenesil-1-s2转染组细胞早期凋亡率分别为27.16%±0.57%、26.20%±0.71%两组间无明显差异(LSD-t = -0.957, P>0.05), 与Pgenesil-1-hk转染组13.58%±0.50%(LSD-t分别为-13.58、-12.62, P均<0.01)和空白组8.53%±0.14%(LSD-t 分别为-18.62、-17.67, P均<0.01)相比, 差异均有显著性. Pgenesil-1-s1、Pgenesil-1-s2转染组早期凋亡明显增加(图3).
组蛋白甲基化转移酶能够使DNA组蛋白的氨基酸残基发生甲基化, 不但使染色质的结构发生变化, 而且更为重要的是能够从表观遗传学水平介导下游基因的转录激活或转录抑制. 新近发现的SMYD3基因所编码的蛋白是一种能够催化H3K4发生2倍和3倍的甲基化, 介导下游基因的转录激活, 在胆管癌、乳腺癌和肝癌组织中高表达, 与肿瘤的发生发展关系密切[12-14]. SMYD3基因有两个重要的结构域SET和zf-MYND, SET具有甲基化转移酶功能[15-16], SMYD3的SET结构域能特异性催化H3K4发生甲基化, zf-MYND能够特异地与靶基因启动子区域的基因序列5'-CCCTCC-3'和5'-GGAGGG-3'相结合, zf-MYND结合靶基因后, SET结构域发挥其H3K4甲基化转移酶功能使靶基因启动子H3K4发生2倍(di-)和3倍(tri-)的甲基化, 激活靶基因转录[7,17-18].
c-Myc基因是一个十分重要的原癌基因, 在许多恶性肿瘤中被激活, 肿瘤组织中c-Myc基因的表达显著增加[19]. 研究发现通过shRNA干扰肝癌细胞HepG2细胞中c-Myc基因的表达, 可以明显抑制细胞的增殖, 促进HepG2细胞的凋亡[20-21]. 但是c-Myc基因被转录激活的机制复杂, 目前研究认为, 他是Wnt/β-catenin的靶基因之一, β-catenin与T细胞转录因子4/淋巴增强因子(TCF-4/LEF)等结合形成复合物后将c-Myc基因激活[22-24], 但是该复合物对c-Myc基因表达的具体调控机制还不清楚.
我们通过应用短发夹RNA Pgenesil-1-s1、Pgenesil-1-s2和阴性对照质粒Pgenesil-1-hk, 成功转染HepG2细胞24、48、72 h后, 结果显示Pgenesil-1-s1组、Pgenesil-1-s2组SMYD3表达明显受抑制, 而Pgenesil-1-hk组SMYD3的表达水平没有明显改变, 各组的β-actin没有明显变化, 说明了shRNA干扰的有效性和特异性. 经Pgenesil-1-s1、Pgenesil-1-s2转染的HepG2细胞, AnnexinⅤ-PE和7-AAD荧光染料双标后, 流式细胞仪检测发现早期凋亡明显增加, 同时RT-PCR测得c-Myc基因表达明显下降, 这意示着SMYD3基因的沉默可以使肝癌细胞凋亡增加, SMYD3可能通过直接或间接激活c-Myc基因, 促进肝癌细胞的增殖和肿瘤发展. 所谓直接激活指SMYD3通过其组蛋白甲基化功能使c-Myc基因的启动子区域的H3K4发生了2倍或3倍的甲基化, 从而使c-Myc基因激活, 因为在c-Myc基因的启动子区域存在5'-CCCTCC-3'和5'-GGAGGG-3'序列[25]; 间接激活指SMYD3对c-Myc基因的上游基因进行调控, 影响c-Myc基因的表达, 其具体机制将是我们今后研究的重点. 结果也提示设计和应用针对SMYD3的特异性shRNA治疗肝癌的可行性, 为肝癌的治疗提供新的方向.
SMYD3是一种具有组蛋白甲基化功能的蛋白, 能够使H3K4发生2倍(di-)和3倍(tri-)的甲基化, 激活下游基因的转录. c-Myc基因是一个原癌基因, 有多种癌的发生密切相关, 但其表达调控机制不十分清楚.
陈建荣, 副教授, 南通大学第二附属医院急诊医学中心
2004年, Hama-moto et al首次报道SMYD3具有组蛋白甲基酶活性, 能结合靶基因DNA启动子序列5'-CCCTCC-3'、 5'-GGAGGG-3'激活靶基因的表达, 与多种肿瘤发生发展有关.
本文首次探讨肝癌HepG2细胞SMYD3可能通过调控c-Myc基因参与肝癌的发生发展.
本研究表明应用RNA干扰技术下调HepG2细胞中SMYD3的表达, 可调控c-Myc基因的表达, 促进肿瘤细胞凋亡, 为将来RNA干扰技术应用于临床提供依据, 并充实了c-Myc基因表达调控的理论基础.
1 表观遗传学: 是指在基因的DNA 序列没有发生改变的情况下, 基因功能发生了可遗传的变化, 并最终导致了表型的变化.
2 组蛋白修饰: 指通过对组蛋白特定残基进行翻译后修饰, 如甲基化、乙酰化、磷酸化等, 调控染色体的结构、
调节基因的转录激活和抑制等.
本研究具有一定的新颖性和先进性, 结论明确有科学价值, 能较好地反映我国基础研究的先进水平.
编辑: 潘伯荣 电编:何基才
| 1. | An W. Histone acetylation and methylation: combinatorial players for transcriptional regulation. Subcell Biochem. 2007;41:351-369. [PubMed] |
| 2. | Peterson CL, Laniel MA. Histones and histone modifications. Curr Biol. 2004;14:R546-R551. [PubMed] |
| 3. | Ito T. Role of histone modification in chromatin dynamics. J Biochem. 2007;141:609-614. [PubMed] [DOI] |
| 4. | Peters AH, Mermoud JE, O'Carroll D, Pagani M, Schweizer D, Brockdorff N, Jenuwein T. Histone H3 lysine 9 methylation is an epigenetic imprint of facultative heterochromatin. Nat Genet. 2002;30:77-80. [PubMed] [DOI] |
| 5. | Santos-Rosa H, Schneider R, Bannister AJ, Sherriff J, Bernstein BE, Emre NC, Schreiber SL, Mellor J, Kouzarides T. Active genes are tri-methylated at K4 of histone H3. Nature. 2002;419:407-411. [PubMed] [DOI] |
| 6. | Wang H, Cao R, Xia L, Erdjument-Bromage H, Borchers C, Tempst P, Zhang Y. Purification and functional characterization of a histone H3-lysine 4-specific methyltransferase. Mol Cell. 2001;8:1207-1217. [PubMed] [DOI] |
| 7. | Hamamoto R, Furukawa Y, Morita M, Iimura Y, Silva FP, Li M, Yagyu R, Nakamura Y. SMYD3 encodes a histone methyltransferase involved in the proliferation of cancer cells. Nat Cell Biol. 2004;6:731-740. [PubMed] [DOI] |
| 8. | Luo XG, Ding Y, Zhou QF, Ye L, Wang SZ, Xi T. SET and MYND domain-containing protein 3 decreases sensitivity to dexamethasone and stimulates cell adhesion and migration in NIH3T3 cells. J Biosci Bioeng. 2007;103:444-450. [PubMed] [DOI] |
| 9. | Xu JY, Chen LB, Xu JY, Yang Z, Wei HY, Xu RH. Inhibition of SMYD3 gene expression by RNA interference induces apoptosis in human hepatocellular carcinoma cell line HepG2. Ai Zheng. 2006;25:526-532. [PubMed] [DOI] |
| 11. | Devi GR. siRNA-based approaches in cancer therapy. Cancer Gene Ther. 2006;13:819-829. [PubMed] [DOI] |
| 12. | Xu JY, Chen LB, Xu JY, Yang Z, Xu RH, Wei HY. Experimental research of therapeutic effect on hepatocellular carcinoma of targeting SMYD3 gene inhibition by RNA interference. Zhonghua Waike Zazhi. 2006;44:481-484. [PubMed] |
| 13. | Hamamoto R, Silva FP, Tsuge M, Nishidate T, Katagiri T, Nakamura Y, Furukawa Y. Enhanced SMYD3 expression is essential for the growth of breast cancer cells. Cancer Sci. 2006;97:113-118. [PubMed] [DOI] |
| 14. | Frank B, Hemminki K, Wappenschmidt B, Klaes R, Meindl A, Schmutzler RK, Bugert P, Untch M, Bartram CR, Burwinkel B. Variable number of tandem repeats polymorphism in the SMYD3 promoter region and the risk of familial breast cancer. Int J Cancer. 2006;118:2917-2918. [PubMed] [DOI] |
| 15. | Qian C, Zhou MM. SET domain protein lysine methyltransferases: Structure, specificity and catalysis. Cell Mol Life Sci. 2006;63:2755-2763. [PubMed] [DOI] |
| 16. | Marmorstein R. Structure of SET domain proteins: a new twist on histone methylation. Trends Biochem Sci. 2003;28:59-62. [PubMed] [DOI] |
| 17. | Kunizaki M, Hamamoto R, Silva FP, Yamaguchi K, Nagayasu T, Shibuya M, Nakamura Y, Furukawa Y. The lysine 831 of vascular endothelial growth factor receptor 1 is a novel target of methylation by SMYD3. Cancer Res. 2007;67:10759-10765. [PubMed] [DOI] |
| 18. | Liu C, Fang X, Ge Z, Jalink M, Kyo S, Bjorkholm M, Gruber A, Sjoberg J, Xu D. The telomerase reverse transcriptase (hTERT) gene is a direct target of the histone methyltransferase SMYD3. Cancer Res. 2007;67:2626-2631. [PubMed] [DOI] |
| 19. | Vita M, Henriksson M. The Myc oncoprotein as a therapeutic target for human cancer. Semin Cancer Biol. 2006;16:318-330. [PubMed] [DOI] |
| 20. | Lin CP, Liu JD, Chow JM, Liu CR, Liu HE. Small-molecule c-Myc inhibitor, 10058-F4, inhibits proliferation, downregulates human telomerase reverse transcriptase and enhances chemosensitivity in human hepatocellular carcinoma cells. Anticancer Drugs. 2007;18:161-170. [PubMed] [DOI] |
| 22. | Cui J, Zhou X, Liu Y, Tang Z, Romeih M. Wnt signaling in hepatocellular carcinoma: analysis of mutation and expression of beta-catenin, T-cell factor-4 and glycogen synthase kinase 3-beta genes. J Gastroenterol Hepatol. 2003;18:280-287. [PubMed] [DOI] |
| 23. | Huang WS, Wang JP, Wang T, Fang JY, Lan P, Ma JP. ShRNA-mediated gene silencing of beta-catenin inhibits growth of human colon cancer cells. World J Gastroenterol. 2007;13:6581-6587. [PubMed] [DOI] |
| 24. | Zhao DH, Hong JJ, Guo SY, Yang RL, Yuan J, Wen CY, Zhou KY, Li CJ. Aberrant expression and function of TCF4 in the proliferation of hepatocellular carcinoma cell line BEL-7402. Cell Res. 2004;14:74-80. [PubMed] [DOI] |
| 25. | DesJardins E, Hay N. Repeated CT elements bound by zinc finger proteins control the absolute and relative activities of the two principal human c-myc promoters. Mol Cell Biol. 1993;13:5710-5724. [PubMed] |