修回日期: 2007-09-30
接受日期: 2007-10-28
在线出版日期: 2007-11-28
目的: 探讨HBV基因型、C区基本核心启动子(BCP)及前C(PC)区变异与拉米夫定抗病毒治疗后HBV DNA反弹的关系.
方法: 应用多引物对巢式PCR法, PCR-序列分析法, 检测拉米夫定治疗27例乙型肝炎患者(治疗组), 以及19例从未用过抗病毒治疗患者(对照组)的HBV基因型PC区, BCP的突变位点.
结果: 27例HBV DNA反弹的患者9例检出G1896A变异率高于对照组(33.33% vs 5.26%, P<0.05), 4例检出C1856T变异(14.81%). 治疗组4份治疗前标本未检出G1896A、C1856T和BCP变异. 与对照组比较, 治疗组PC(G1896A)及BCP(A1762T+G1764A)双变异的患者中B基因型的构成比增高, 分别为75%和50%, C基因型的构成比下降, 分别为25%和50%. 其中在BCP(A1762T+G1764A)变异患者中B、C基因型构成比与对照组比较有显著性差异(P<0.05). 4例HBV DNA反弹患者治疗前未检出有基因变异, 治疗后有2例检出变异, BCP变异1例, BCP+PC变异1例. 27例HBV DNA反弹患者BCP变异4例, PC变异2例, BCP+PC变异8例.
结论: BCP(T1762/A1764)变异、PC区(G1896A)变异可能与拉米夫定治疗后HBV DNA反弹有关. 病毒变异导致的HBV DNA反弹可以是单基因变异引起, 也可以是多个基因联合变异引起, 拉米夫定治疗后B基因型患者更易发生A1762T+G1764A变异.
引文著录: 苏明华, 江建宁, 周元平, 陈茂伟, 刘志红. 乙型肝炎病毒基因型、BCP及PC区变异与拉米夫定治疗后HBV DNA反弹的关系. 世界华人消化杂志 2007; 15(33): 3507-3513
Revised: September 30, 2007
Accepted: October 28, 2007
Published online: November 28, 2007
AIM: To investigate the relationship of HBV gene mutations with HBV DNA rebound after lamivudine therapy.
METHODS: Twenty-seven hepatitis B patients with HBV DNA rebound after lamivudine therapy (therapy group) and 19 patients without antiviral therapy (control group) were investigated. HBV genotype was detected by nested PCR with multiple pair primers. Mutations of basal core promoter (BCP) and precore (PC) were identified by PCR gene sequence analysis in sera of 27 patients after HBV DNA rebound, and in sera of 4 patients before therapy in the therapy group and in sera of 19 patients in the control group.
RESULTS: The ratio of BCP (A1762T + G1764A) mutations in serum of patients with HBV DNA rebound was 44.44% (12/27) higher than 26.32% (5/19) in the control group, although not significantly. The mutation was not detected in the sera of 4 patients before therapy. The ratio of the PC (G1896A) mutation in serum of patients with HBV DNA rebound was 33.33% (9/27) higher than 5.26% (1/19) in the control group (P < 0.01). The mutation was not detected in the sera of 4 patients before therapy. The ratio of the C1856T mutation was 14.81% (4/27) higher than 10.53%(2/19) in the control group, but not significantly. It was not detected in the sera of 4 patients before therapy. In patients with the BCP mutation (A1762T + G1764A), the constituent ratio of genotype B was 75% higher and that of genotype C was 25% lower than those in the control group (both P < 0.05). In patients with mutation PC (G1896A), the constituent ratio of genotype B was 50% higher, and that of genotype C was 50% lower than those in the control group ( P > 0.05). Gene mutations were not detected in four patients before lamivudine therapy but BCP and BCP + PC mutations occurred in two patients after HBV rebound. In 27 patients with HBV DNA rebound, there were BCP mutations in four, PC mutation in two, and BCP + PC mutation in eight.
CONCLUSION: BCP (T1762/A1764) and PC (G1896A) mutations may be involved in HBV DNA rebound after lamivudine therapy. The mutations occur in both single and multiple gene groups. The A1762T + G1764A mutation occurs more frequently in patients with genotype B after lamivudine therapy.
- Citation: Su MH, Jiang JN, Zhou YP, Chen MW, Liu ZH. Relationship of HBV genotype and bcp and pc mutations with HBV DNA rebound after lamivudine therapy. Shijie Huaren Xiaohua Zazhi 2007; 15(33): 3507-3513
- URL: https://www.wjgnet.com/1009-3079/full/v15/i33/3507.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i33.3507
慢性乙型肝炎是最常见的感染性疾病之一, 全世界约有3.5亿人感染乙型肝炎病毒(HBV)[1], 其中75%在亚洲[2]. 我国是乙型病毒性肝炎的流行大国, 其中HBV携带者几乎占了人口的10%, 即1.2亿人. 其中约有15%-40%的感染者发展为不同类型的肝病, 如活动性肝炎、肝硬化、甚至原发性肝癌. 目前认为持续病毒复制是慢肝发展到肝硬化、重肝的主要原因. 因此抗病毒治疗是阻止和延缓病程发展的重要治疗措施. 拉米夫定于1999年开始在国内广泛应用于治疗乙肝, 其疗效已得到多数学者肯定. 但在治疗的过程中出现了许多问题, 如用药疗程不确定, 用药时间长会出现基因变异, 停药后又会出现HBV DNA反弹等. HBV是一种嗜肝DNA病毒, 基因组约含3200个bp, 其长链有4个开放性读框(ORF), 即S区、C区、P区和X区. 由于在HBV的复制过程中有逆转录过程的参与, 因而HBV较其他DNA病毒更容易发生变异. HBV基因型可以分为A-H八种[3], 不同的基因型可能有不同的病毒变异特点. 国内外已公认拉米夫定治疗后出现的耐药导致HBV DNA反弹与YMDD变异密切相关, 对于拉米夫定治疗后是否还可有其他基因区的变异, 目前国外文献报道很少, 国内无这方面的研究. 为了探讨HBV基因型、BCP及PC变异及变异位点与拉米夫定抗病毒治疗的关系, 以及拉米夫定治疗后HBV DNA反弹时是否存在联合变异的现象, 我们做了以下研究.
1.1.1 病例来源: 来源于2001-12/2004-12广西医科大学第一附属医院传染病科门诊及住院患者.
1.1.2 研究对象入选标准: 治疗组: (1)慢性乙型肝炎或者乙肝后肝硬化患者, 诊断标准符合2000年"全国第十次病毒性肝炎学术会议"修订的病毒性肝炎诊断标准[4]; (2)治疗前HBV DNA拷贝数≥1.0×107拷贝/L, 谷丙转氨酶(ALT>1.5倍正常值)反复异常>6 mo; (3)用拉米夫定治疗(100 mg/d)达到病毒血应答(HBV DNA<1.0×106拷贝/L)超过半年; (4)疗程至少1年; (5)治疗前均未用过抗病毒药物; (6)患者依从性好, 能按要求定期回访. 所有患者追踪至HBV DNA反弹时即HBV DNA由阴性转为阳性. 对照组: 从未用过抗病毒药物治疗的慢性乙型肝炎或者乙肝后肝硬化患者. 两组患者治疗前HBV DNA拷贝数相近, 平均6.7log和6.56log.
1.1.3 患者剔除标准: (1)合并有抗HCV, 抗HDV, 抗HIV阳性; (2)乙肝病毒携带者、急性肝炎、重型肝炎、肝癌患者; (3)HBV DNA拷贝数<1.0×107拷贝/L; (4)用免疫抑制剂治疗者.
1.1.4 入选对象的一般资料: 符合入选标准的治疗组患者27例, 其中男19例, 女8例, 年龄20-58(平均35.26)岁, 乙肝后肝硬化3例, 慢性乙肝24 例, 其中HBeAg(+)18例, HBeAg(-)9例. 用药疗程12-33 mo, 平均19.99 mo. 对照组患者19例, 男16例, 女3例, 年龄20-58(平均33.63) 岁, 乙肝后肝硬化3例, 慢性乙肝16例, 其中HBeAg(+)13例, HBeAg(-)6例. 两组中>35岁的患者占67.39%.
1.1.5 试剂和仪器: Tap DNA合成酶由TaKaRa宝生物(大连)科技有限公司提供. 10 mmol/L dNTP, 50 bp DNA Marker由广州东盛生物科技公司提供. 美国TheromP×2 Thermal cycler, 美国Bio-Rad公司GelDoc 2000凝胶电泳成像分析系统.
检测治疗前、治疗后每个月和HBV DNA阴转后每3 mo的肝功能、HBV DNA和 HBsAg, HBsAb, HBeAg, HBeAb, HBcAb标记物.
1.2.1 血清的处理: 共收集到50份血清标本: 治疗组治疗前4份, 治疗后HBV DNA反弹时的标本27份, 对照组收集到19份. 将收集到的血清置于 -20℃冰箱中贮存.
1.2.2 HBV: DNA的提取: 采用酚: 氯仿抽提法, 将收集到的血清500 μL置于1.5 mL离心管内, 加等量饱和苯溶液, 混匀, 3500 r/min离心5 min. 取上清液置于另一1.5 mL离心管内, 加等量氯仿•异戊醇溶液, 混匀, 3500 r/min离心5 min; 取上清液置于另一1.5 mL离心管内, 加入2.5倍无水乙醇, 轻轻摇晃离心管, 1000 r/min离心10 min, 弃上清液, 底部即为析出的DNA. 加入750 mL/L冰冻乙醇400 μL洗涤, 12 000 r/min离心15 min, 去全部液体. 待干燥后加30 μLTAE缓冲液溶解, 置4℃冰箱待测.
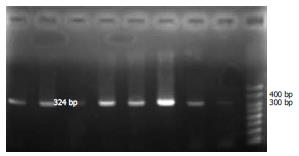
1.2.3 检测项目: 检测46例患者的HBV基因型、BCP, PC基因的变异. HBV基因型引物根据HBV DNApre-S和S基因区保守序列设计[5]. BCP, PC基因引物设计: 检索GenBank收录的HBV基因全序列(基因号为AY123041), 采用Premier5.0引物设计软件对HBV病毒基因组保守区序列设计引物, BCP, PC(sense)GCATGGAGACCACCGTGAAC(nt1607-1627), (antisense)CAAATTCTTTATACGGGTCAAT(nt1930-1908), 产物324 bp, 由上海生工生物工程有限公司合成.
1.2.4 检测方法: HBV DNA用荧光定量PCR法, HBsAg, HBsAb, HBeAg, HBeAb, HBcAb用ELISA法, 均由广西医科大学第一附属医院检验中心完成. HBV DNA<1.0×106拷贝/L定为阴性.
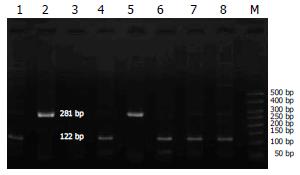
1.2.5 HBV基因型的检测: (1)采用基因型特异多引物对巢式PCR法: 第一轮PCR: 反应体系50 μL即ddH2O 34.6 μL, 10×PCR 缓冲液 8 μL, 10 mmol/L dNTP 1.0 μL, 浓度为10 μmol/L的正义、反义引物各1.0 μL, 5×103 kU/L Taq DNA聚合酶0.4 μL, DNA模板4 μL. 反应参数为: 预变性94℃ 5 min, 循环参数为变性94℃ 1 min, 退火55℃ 1 min, 延伸72℃ 1 min, 共34个循环, 最后72℃延伸10 min的扩增条件放入PCR仪中扩增. 第二轮PCR: 分两组进行, 第一组反应体系的引物为A, B, C型特异性引物, 第二组反应体系的引物为D, E, F型特异性引物. 第一组反应体系25 μL即ddH2O 15.3 μL, 10×PCR 缓冲液4 μL, 10 mmol/L dNTP 0.5 μL, 浓度为10 μmol/L的A to C正义引物1.5 μL, A, B, C型特异性引物反义引物各0.5 μL, 5×103 kU/L Taq DNA聚合酶0.2 μL, 第一轮PCR扩增产物2 μL. 第二组反应体系亦为25 μL, 除引物为10 μmol/L的D to F正义引物1.5 μL, D, E, F型特异性引物反义引物各0.5 μL外, 其余均与第一组反应体系相同, 反应参数均与第一轮PCR相同. (2)HBV基因型的判定: 分别取第二轮两组PCR的扩增产物10 μL加2 μL 6×Loading dye 混合在15 g/L琼脂糖上样, 同时加入50 bp Marker对照, 100 V 40 min电泳后用凝胶电泳成像分析系统观察并照相. 通过产物片段大小判定待测样品的基因型, 即68bp-A基因型, 281bp-B基因型, 122bp-C基因型, 119bp-D基因型, 167bp-E基因型, 97bp-F基因型(图1). (3)BCP, PC区变异的检测: 采用PCR-序列分析法. PCR扩增反应体系25 μL: ddH2O 17.3 μL, 10×PCR缓冲液4 μL, 10 mmol/L dNTP 0.5 μL, 浓度为10 μmol/L的正义、反义引物各0.5 μL, 5×103 kU/L Taq DNA聚合酶0.2 μL, DNA模板2 μL. 反应参数为: 预变性94℃ 5 min, 循环参数为变性94℃ 1 min, 退火55℃(C区为52℃) 1 min, 延伸72℃ 1 min, 共34个循环, 最后72℃延伸10 min. (4)基因测序: 上述扩增的产物10 μL加2 μL 6×Loading dye 混合在15 g/L琼脂糖上样, 同时加入50 bp Marker对照, 110 V 40 min电泳后用凝胶电泳成像分析系统观察并照相. BCP(包括PC区)的PCR扩增产物长度为324 bp, (图2)将扩增出的样品送上海鼎安生物科技有限公司经纯化后进行正义序列测定. 测序结果采用Analysis(Vect NTI suite 8.0)对比软件A-F基因型标准株核苷酸序列进行对比分析, 检测各区的变异位点. HBV P, C区的测序结果经翻译成氨基酸序列后与各区标准的氨基酸序列作对比分析.
统计学处理 全部数据采用SPSS13.0统计软件包进行统计处理, 率的比较用四格表的确切概率法.
BCP变异的检测结果, A1762T, G1764A均是同时出现, 治疗后BCP(A1762T, G1764A)变异检出率(44.44%)高于对照组(26.32%), 但无统计学差异. 同时在未治疗标本中还可检出7例有G1721A, 5例T1753C, 4例G1757A, 6例A1775G位点变异. 其中3例A1762T, G1764A 双变异同时合并有G1721A, T1753C, G1757A的变异. G1721A, T1753C, G1757A, A1775G位点的变异, 治疗组与对照组比较无统计学意义(表1).
| 分组 | n | A1762T+G1764A | G1721A | T1753C | G1757A | A1775G | G1896A | C1856T |
| 对照组 | 19 | 5(26.32) | 6(31.58) | 4(21.05) | 3(0.00) | 5(0.00) | 1(5.26) | 2(10.53) |
| 治疗组 | ||||||||
| 治疗前 | 4 | 0(0.00) | 1(25.00) | 1(25.00) | 1(25.00) | 1(25.00) | 0 | 0 |
| 治疗后 | 27 | 12(44.44) | 8(29.63) | 0(0.00) | 3(11.11) | 4(14.81) | 9(33.33)a | 4(14.81) |
PC区可检出G1896A及C1856T变异位点, G1896A变异检出率治疗组高于对照组(P<0.05). C1856T变异治疗组与对照组比较(P>0.05, 表1, 2).
| 分组/变异 | n | B基因型(%) | C基因型(%) | B+C混合型(%) |
| 对照组 | 19 | 6(31.58) | 13(68.42) | |
| 治疗组 | 27 | 10(37.04) | 16(59.26) | 1(3.70) |
| BCP | 12 | 9(75.00) | 3(25.00) | |
| PC | 10 | 5(50.00) | 5(50.00) |
与对照组比较, 治疗组PC(G1896A)及BCP(A1762T+G1764A)双变异的患者中B基因型的构成比增高, C基因型的构成比下降, 其中在BCP(A1762T+G1764A)变异患者中B, C基因型构成比与对照组比较有显性差异(P<0.05, 表2).
4例HBV DNA反弹患者治疗前未检出有基因变异, 治疗后有2例检出变异, BCP变异1例, BCP+PC变异1例.
27例HBV DNA反弹患者BCP变异4例、PC变异2例、BCP+PC变异8例.
近15年的研究, 对HBV基因变异已有了较深入的了解. 研究发现, 病毒变异可能会导致病毒本身的复制、转录和表达水平的改变, 从而影响慢性乙型肝炎的临床诊断及治疗, 特别是核苷类似物耐药性的发现具有重要的临床价值. 拉米夫定治疗慢性乙型肝炎的主要缺陷之一是HBV对其易产生耐药性, 且随着治疗时间的延长, 耐药率明显增加.亚洲的临床研究数据显示, 拉米夫定治疗1-4年的耐药率分别为14%, 38%, 49%和66%[6-8]. 目前认为[9], HBV对核苷类似物耐药性的产生与HBV P基因变异有关, 以C区酪氨酰(Y)、蛋氨酰(M)、天冬氨酰(D)、天冬氨酰(D)氨基酸(YMDD)基因的变异最为重要. 在我们的一项研究中(待发表), 在HBV DNA反弹的27例患者中, YMDD变异的检出率占48%, 仍有52%患者无YMDD变异. 我们认为除YMDD外, 还存在其他引起拉米夫定治疗HBV DNA反弹的因素. BCP变异即基本核心启动子变异, 定位于1744-1851 nt之间, 大部分与X基因区重叠. 他可引起基本启动子与转录因子的结合减少, 导致前C和C mRNA的转录减少, 从而造成前C区蛋白的减少, 但不影响HBV其他RNA的转录和核心蛋白或多聚酶蛋白的翻译. 由于减少了HBV前C区蛋白对HBV复制的抑制作用, BCP变异株的复制能力有所增强. BCP中最重要、最常见的突变是1762 nt A→T, 1764 nt G→A, 两者常同时出现, 称为双突变. 其在HBeAg阴性的HBV长期携带者中高度流行, 但在HBeAg阳性者中却较罕见, BCP的变异在转录水平上影响HBeAg的形成. 体外试验证实BCP变异有利于病毒的复制[10]. Kim[11]研究100例慢性乙型肝炎(HBeAg阳性50例)发现BCP(T1762/A1764)变异, 在HBeAg阳性和阴性患者中均存在. Sendi et al[12]研究30例HBeAg阴性慢性乙型肝炎发现BCP(T1762/A1764)变异的占20%, T1764G1766变异的占33%, 认为BCP常见变异位点在T1764和G1766, 而且常常联合A1757的变异, 与病毒高复制及转氨酶升高有关. BCP变异对核苷类似物治疗的影响. Chen et al[13]认为T1762/A1764(无A1896)变异有益于拉米夫定治疗后HBeAg转阴. Tacke et al[14]的研究认为BCP变异不影响耐药性, 但可使病毒的复制能力增强及可增强耐拉米夫定的变异株复制能力. 本研究检出BCP变异的位点有A1762T+G1764A, G1721A, T1753C, G1757A, A1775G, T1762+A1764双变异仍是BCP常见的变异位点. G1721A, T1753C, G1757A, A1775G的变异在治疗组和对照组均存在, 且变化不大, 可能不影响拉米夫定治疗的疗效. 是否影响乙型肝炎的临床表现有待进一步研究. BCP(T1762/A1764)变异在治疗组HBV DNA反弹的患者检出率(44.44%)高于对照组(26.32%), 但统计学上无差异, 可能与样本量少有关. 我们认为在拉米夫定这种抗病毒药物的压力下, 亦可导致BCP变异, BCP变异增强耐拉米夫定的变异株复制能力, 导致HBV DNA反弹.
前C基因的突变影响HBeAg的表达. 1989年Carman et al[15]首次在乙肝患者HBV基因组中, 用PCR技术证明前C区核昔酸(nt)1896G--A的变异, 使密码子TGG变为TAG, 产生一个终止密码, 使前C蛋白的翻译终止, 从而使HBeAg不能合成. 前C区基因变异对核苷类似物疗效的影响. Reiichiro et al[16]观察54例慢性乙型肝病患者用拉米夫定治疗至少1年, 结果提示有前C区变异的效果好. Chen et al[13]研究74例HBeAg阳性的慢性肝病患者用拉米夫定治疗9-12 mo, 结果提示在治疗后和HBeAg阴转的34例完全应答的患者T1762/A1764变异增多而A1896变异减少. 40例无应答患者中可出现1773, 1802, 1803, 1845, 1850和1858核苷酸位点的变异, 认为T1762/A1764(无A1896)变异有益于拉米夫定治疗后HBeAg转阴. 1773, 1802, 1803, 1845, 1850和1858核苷酸位点的变异是患者无应答的原因. Shin et al[17]研究认为1896位的突变是拉米夫定长期治疗后低应答的原因. 我们的结果显示, PC(G1896A)变异的发生率(9/27, 33.33%)在拉米夫定治疗后HBV DNA反弹的患者中明显高于对照组(1/19, 5.26%)(P<0.05). 因此, 我们认为PC(G1896A)变异的出现也是HBV DNA反弹的原因之一. 同时我们还检测到C1856T位点的变异, 但治疗组与对照组比较(P>0.05). C1856T位点的变异可能与HBV DNA反弹关系不大.
从治疗组治疗前后的4例患者看, 治疗前均未检出基因变异, 治疗后有3例患者检出基因变异, 其中两例为联合变异. 我们认为, 病毒变异导致的HBV DNA反弹的基因变异是多种形式的, 可以是单基因变异引起, 也可是多个基因联合变异引起.因此, 临床上在检测拉米夫定耐药变异时, 不能只单纯检测YMDD, 还应考虑检测其他基因变异.
HBV的基因型在1988年由Okamoto et al[18]提出. 目前发现的HBV基因型可以分为A-H八种[19-21], 并呈现一定的地理区域性分布特征. 在中国大陆地区, 主要为B和C基因型, 其中南方以B基因型为主, 北方以C基因型为主[22]. 我们采用基因型特异多引物对巢式PCR法经过两次PCR扩增, 从PCR产物片段的大小直接判断HBV的基因型. 本研究共检测46例患者血清, 发现B基因型16例(34.78%), C基因型29例(63.04%), B+C基因型1例, 未检测出D, E, F, G基因型. 我们的结果与国内的相关报道有差异[22], 可能与所选择患者的年龄多数在35岁以上有关.
不同基因型HBV对抗病毒药物的应答不一样, HBV基因型对拉米夫定抗病毒治疗的影响, 目前看法不一. 国内王永忠 et al通过对68例接受核苷类药物拉米夫定治疗的慢性乙肝患者的跟踪观察, 发现拉米夫定治疗后HBV C基因型感染者反弹比例明显低于B基因型感染者. 而香港Chan et al[23]及Kao et al[24]的研究均认为基因型对HBeAg血清转换无影响. Westland et al[25]在adefovir dipivoxil Ⅲ期临床试验的研究中认为HBV基因型并不影响阿得福韦的抗病毒疗效. 我们结果显示, 对照组B基因型构成比为31.58%(6/19), C基因型构成比为68.42%(13/19). 治疗组B基因型构成比为37.04%(10/27), C基因型构成比为59.26%(16/27). 两组患者B, C基因型的构成比比较(P>0.05). 我们认为感染B, C基因型的患者, 对用拉米夫定治疗后出现HBV DNA反弹无差别. 关于这一结果, 由于样本量不够大, 仍须扩大样本量进一步证实.
不同的基因型与某些变异相关, 如前C区1896变异是基因型依赖的. 在B, C, D和E型的G1896与T1858配对的干襻结构不稳定, 1896的G-A变异使干襻结构趋于稳定, 故前C终码变异率较高[26]. 而A和F型的1858位核苷酸为C, 能与1896位核苷酸G形成稳定的结构, 较少发生变异[27]. 所以, 前C区1896变异的流行率与基因型相关. 台湾学者[28]通过对200例慢性乙肝及肝硬化患者的研究发现, C基因型BCP(T1762/A1764)变异率较高, 出现T1762/A1764变异的患者更易发展为肝硬化、HCC. Chan, Lindh, Orito, Kao et al[29-32]的研究也认为BCP(T1762/A1764)变异C基因型较B基因型常见. 也有学者认为T1762/A1764变异伴随前C区变异常见于A基因型患者[33]. Liu et al[34]观察了45名慢性肝炎患者, 发现1858位核苷酸为C的基因型(A, F基因型)较1858位核苷酸为T的基因型(B, C, D, E基因型)更易出现BCP区变异. 也有人认为BCP变异在A, D基因型是相似的, 而前C区的变异常见于D基因型[12]. 我们的研究结果显示, 与对照组比较, 治疗组PC(G1896A)及BCP(A1762T+G1764A)双变异的患者中B基因型的构成比增高, 分别为75%和50%; C基因型的构成比下降, 分别为25%和50%. 其中在BCP(A1762T+ G1764A)变异患者中B, C基因型构成比与对照组比较有显性差异(P<0.05), 即BCP(A1762T+G1764A)双变异在B基因型中常见.
拉米夫定治疗后HBV DNA反弹可能与BCP, PC变异有关, 而B基因型患者更易发生BCP变异.
国内外已公认拉米夫定治疗后出现的耐药导致HBV DNA反弹与YMDD变异密切相关. 对于拉米夫定治疗后是否还可有其他基因区的变异, 以及是否存在联合变异、基因型与变异的关系, 为探讨这一问题本文做了以下研究.
文献中研究拉米夫定治疗后出现的基因区变异主要为YMDD变异, 但是否还存在有其他基因区的变异, 目前国外文献报道很少, 国内罕见这方面的研究. 本文除YMDD变异外侧重于多个基因区的联合变异(BCP、PC区), 并且设立了对照组(未用拉米夫定治疗)及治疗前后的对照.
国内汤守兵et al研究认为BCP变异可影响病毒复制, BCP变异可能与肝病的严重程度存在着相关性.侯阮沛et al认为乙型肝炎病毒前C区和BCP区突变能显著影响HBeAg量的表达, 从而导致乙型肝炎病毒致病能力的变化.
本文证实了YMDD变异是拉米夫定治疗后出现耐药导致HBV DNA反弹的主要原因, 而且提示BCP(A1762T /G1764A)变异、前C区G1896A变异是病毒耐药的另一机制. 病毒变异导致的HBV DNA反弹可以是单基因变异或多基因联合变异引起.
病毒变异导致的HBVDNA反弹的基因变异是多种形式的, 可以是单基因变异引起, 也可是多个基因联合变异引起. 临床上在检测拉米夫定耐药变异时, 不能只单纯检YMDD, 还应考虑检测其他基因变异(如BCP, PC区变异).
本文研究方法设计合理, 方法科学, 数据可信, 具有较强的参考价值.
编辑: 孙学英 电编:马文华
| 1. | Marques AR, Straus SE. Advances in the treatment of chronic hepatitis B virus infection. Rev Med Virol. 1998;8:223-234. [PubMed] |
| 2. | Gust ID. Epidemiology of hepatitis B infection in the Western Pacific and South East Asia. Gut. 1996;38 Suppl 2:S18-23. [PubMed] |
| 3. | Norder H, Courouce AM, Magnius LO. Complete genomes, phylogenetic relatedness, and structural proteins of six strains of the hepatitis B virus, four of which represent two new genotypes. Virology. 1994;198:489-503. [PubMed] |
| 5. | Naito H, Hayashi S, Abe K. Rapid and specific genotyping system for hepatitis B virus corresponding to six major genotypes by PCR using type-specific primers. J Clin Microbiol. 2001;39:362-364. [PubMed] |
| 6. | Liaw YF, Leung NW, Chang TT, Guan R, Tai DI, Ng KY, Chien RN, Dent J, Roman L, Edmundson S. Effects of extended lamivudine therapy in Asian patients with chronic hepatitis B. Asia Hepatitis Lamivudine Study Group. Gastroenterology. 2000;119:172-180. [PubMed] |
| 7. | Leung NW, Lai CL, Chang TT, Guan R, Lee CM, Ng KY, Lim SG, Wu PC, Dent JC, Edmundson S. Extended lamivudine treatment in patients with chronic hepatitis B enhances hepatitis B e antigen seroconversion rates: results after 3 years of therapy. Hepatology. 2001;33:1527-1532. [PubMed] |
| 8. | Lok AS, McMahon BJ. Chronic hepatitis B: update of recommendations. Hepatology. 2004;39:857-861. [PubMed] |
| 9. | Stuyver LJ, Locarnini SA, Lok A, Richman DD, Carman WF, Dienstaq JL, Schinazi RF. Nomenclatu re for antiviral-resistant human hepatitis B virus mutations in the polymerase region. Hepatology. 2001;33:751-757. [PubMed] |
| 10. | Laras A, Koskinas J, Hadziyannis SJ. In vivo suppression of precore mRNA synthesis is associated with mutations in the hepatitis B virus core promoter. Virology. 2002;295:86-96. [PubMed] |
| 11. | Kim HJ, Yoo BC. The prevalence and clinical significance of precore and core promoter mutations in Korean patients with chronic hepatitis B virus infection. Taehan Kan Hakhoe Chi. 2002;8:149-156. [PubMed] |
| 12. | Sendi H, Mehrab-Mohseni M, Zali MR, Norder H, Magnius LO. T1764G1766 core promoter double mutants are restricted to Hepatitis B virus strains with an A1757 and are common in genotype D. J Gen Virol. 2005;86:2451-2458. [PubMed] |
| 13. | Chen CH, Lee CM, Lu SN, Changchien CS, Wang JC, Wang JH, Hung CH, Hu TH. Comparison of sequence changes of precore and core promoter regions in HBeAg-positive chronic hepatitis B patients with and without HBeAg clearance in lamivudine therapy. J Hepatol. 2006;44:76-82. [PubMed] |
| 14. | Tacke F, Gehrke C, Luedde T, Heim A, Manns MP, Trautwein C. Basal core promoter and precore mutations in the hepatitis B virus genome enhance replication efficacy of Lamivudine-resistant mutants. J Virol. 2004;78:8524-8535. [PubMed] |
| 15. | Carman WF, Jacyna MR, Hadziyannis S, Karayiannis P, McGarvey MJ, Makris A, Thomas HC. Mutation preventing formation of hepatitis B e antigen in patients with chronic hepatitis B infection. Lancet. 1989;2:588-591. [PubMed] |
| 16. | Kuwahara R, Kumashiro R, Murashima S, Ogata K, Tanaka K, Hisamochi A, Hino T, Ide T, Tanaka E, Koga Y. Genetic heterogeneity of the precore and the core promoter region of genotype C hepatitis B virus during lamivudine therapy. J Med Virol. 2004;72:26-34. [PubMed] |
| 17. | Shin JW, Chung YH, Choi MH, Kim JA, Ryu SH, Jang MK, Kim IS, Park NH, Lee HC, Lee YS. Precore stop codon mutation of hepatitis B virus is associated with low breakthrough rate following long-term lamivudine therapy. J Gastroenterol Hepatol. 2005;20:844-849. [PubMed] |
| 18. | Okamoto H, Tsuda F, Sakugawa H, Sastrosoewignjo RI, Imai M, Miyakawa Y, Mayumi M. Typing hepatitis B virus by homology in nucleotide sequence: comparison of surface antigen subtypes. J Gen Virol. 1988;69:2575-2583. [PubMed] |
| 19. | Norder H, Courouce AM, Magnius LO. Complete genomes, phylogenetic relatedness, and structural proteins of six strains of the hepatitis B virus, four of which represent two new genotypes. Virology. 1994;198:489-503. [PubMed] |
| 20. | Magnius LO, Norder H. Subtypes, genotypes and molecular epidemiology of the hepatitis B virus as reflected by sequence variability of the S-gene. Intervirology. 1995;38:24-34. [PubMed] |
| 21. | Stuyver L, De Gendt S, Van Geyt C, Zoulim F, Fried M, Schinazi RF, Rossau R. A new genotype of hepatitis B virus: complete genome and phylo-genetic relatedness. J Gen Virol. 2000;81:67-74. [PubMed] |
| 23. | Chan HL, Wong ML, Hui AY, Chim AM, Tse AM, Hung LC, Chan FK, Sung JJ. Hepatitis B virus genotype has no impact on hepatitis B e antigen seroconversion after lamivudine treatment. World J Gastroenterol. 2003;9:2695-2697. [PubMed] |
| 24. | Kao JH, Liu CJ, Chen DS. Hepatitis B viral genotypes and lamivudine resistance. J Hepatol. 2002;36:303-304. [PubMed] |
| 25. | Westland C, Delaney W 4th, Yang H, Chen SS, Marcellin P, Hadziyannis S, Gish R, Fry J, Brosgart C, Gibbs C, Miller M, Xiong S. Hepatitis B virus genotypes and virologic response in 694 patients in phase III studies of adefovir dipivoxil1. Gastroenterology. 2003;125:107-116. [PubMed] |
| 26. | Mayerat C, Mantegani A, Frei PC. Does hepatitis B virus(HBV)genotype influence the clinical outcome of HBV infection? J Viral Hepat. 1999;6:299-304. [PubMed] |
| 27. | Locarnini S. Molecular virology of hepatitis B virus. Semin Liver Dis. 2004;24 Suppl 1:3-10. [PubMed] |
| 28. | Kao JH. Hepatitis B virus genotypes and hepato-cellular carcinoma in Taiwan. Intervirology. 2003;46:400-407. [PubMed] |
| 29. | Chan HL, Hussain M, Lok AS. Different hepatitis B virus genotypes are associated with different mutations in the core promoter and precore regions during hepatitis B e antigen seroconversion. Hepatology. 1999;29:976-984. [PubMed] |
| 30. | Lindh M, Hannoun C, Dhillon AP, Norkrans G, Horal P. Core promoter mutations and genotypes in relation to viral replication and liver damage in East Asian hepatitis B virus carriers. J Infect Dis. 1999;179:775-782. [PubMed] |
| 31. | Orito E, Mizokami M, Sakugawa H, Michitaka K, Ishikawa K, Ichida T, Okanoue T, Yotsuyanagi H, Iino S. A case-control study for clinical and molecular biological differences between hepatitis B viruses of genotypes B and C. Japan HBV Genotype Research Group. Hepatology. 2001;33:218-223. [PubMed] |
| 32. | Kao JH, Chen PJ, Lai MY, Chen DS. Basal core promoter mutations of hepatitis B virus increase the risk of hepatocellular carcinoma in hepatitis B carriers. Gastroenterology. 2003;124:327-334. [PubMed] |
| 33. | Hunt CM, McGill JM, Allen MI, Candreay LD. Clinical relevance of hepatitis B viral mutations. Hepatology. 2000;31:1037-1044. [PubMed] |
| 34. | Liu CJ, Kao JH, Lai MY, Chen PJ, Chen DS. Precore/core promoter mutations and genotypes of hepatitis B virus in chronic hepatitis B patients with fulminant or subfulminant hepatitis. J Med Virol. 2004;72:545-550. [PubMed] |