修回日期: 2007-09-13
接受日期: 2007-09-28
在线出版日期: 2007-09-28
目的: 改建布鲁氏菌自杀载体.
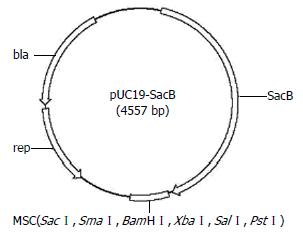
方法: 用NdeⅠ将pKOBEG-SacB质粒中的反向筛选基因sacB切下来, 与同样酶切处理的pUC19质粒连接, 得到pUC19-SacB, 经测序和蔗糖筛选实验证实.
结果: 成功将SacB从pKOBEG-SacB中切下来并插入到pUC19质粒中, 构建得到了pUC19-SacB. 测序和蔗糖筛选实验证实, 插入的sacB基因具有较好的反筛功能. 我们利用该质粒, 成功的构建了布鲁氏菌Ⅳ型分泌系统的突变株, 改建的载体能高效的应用于布鲁氏菌染色体的精确修饰.
结论: 构建了一个新的自杀质粒pUC19-SacB, 该质粒能用于布鲁氏菌无痕缺失突变株的构建.
引文著录: 王玉飞, 陈泽良, 乔凤, 汪舟佳, 杜昕颖, 苑锡铜, 黄留玉. 布鲁氏菌自杀载体的改建及其在突变株构建中的应用. 世界华人消化杂志 2007; 15(27): 2934-2937
Revised: September 13, 2007
Accepted: September 28, 2007
Published online: September 28, 2007
AIM: To developed a new suicide plasmid, pUC19-SacB, for Brucella mutant construction.
METHODS: The counter-selection gene sacB, released from plasmid pKOBEG-SacB by digestion with NdeⅠ, was inserted into the same site of plasmid pUC19 to produce pUC19-SacB. This plasmid was then applied to the construction of a Brucella mutant. DNA sequencing and sucrose selection were used.
RESULTS: The counter-selection gene sacB was successfully released from pKOBEG-SacB and cloned into pUC19 to produce pUC19-SacB. Using DNA sequencing and sucrose selection, the pUC19-SacB plasmid was shown to have counter-selection capability. Using pUC19-SacB, we successfully constructed an unmarked deletion mutant of the VirB operon, showing that this plasmid can be efficiently used to modify the Brucella chromosome.
CONCLUSION: The successfully constructed new suicide plasmid pUC19-SacB can be used to construct a Brucella unmarked mutant.
- Citation: Wang YF, Chen ZL, Qiao F, Wang ZJ, Du XY, Yuan XT, Huang LY. Development of a new suicide plasmid and its application to Brucella mutant construction. Shijie Huaren Xiaohua Zazhi 2007; 15(27): 2934-2937
- URL: https://www.wjgnet.com/1009-3079/full/v15/i27/2934.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i27.2934
布鲁氏菌病(Brucellosis)是由布鲁氏菌(Brucella)引起的一种人畜共患病, 在世界各地都有广泛流行[1]. 在我国, 布鲁氏菌病曾在一段时间内得到了较好的控制, 人间和畜间布鲁氏菌病的发病率下降到了一个较低的水平. 但是, 由于没有较好的监测畜间疫情并及时采取控制措施, 近年来, 布鲁氏菌病在我国有重新抬头的趋势. 一直以来, 致病机制研究都是布鲁氏菌研究的重点和热点[2]. 目前, 病原菌的致病机制研究主要是通过失活或缺失特定的基因, 然后分析突变株各种表型的变化, 从而解析目标基因的功能[3].
因此, 突变株的构建是致病机制研究的前提. 在以往的研究中, 常以克隆载体pUC19等作为自杀载体来构建布鲁氏菌的插入失活突变株, 而精确的缺失则是采用经典的自杀载体构建的, 如pCVD442等[4]. 但是, 由于这类载体的分子操作需要在特殊的宿主菌中进行, 并且克隆的效率较低, 突变株的构建往往比较困难, 已成为后基因组时代大规模功能研究的一个限速步骤[5-6]. 为了进一步提高布鲁氏菌突变株的构建效率, 我们对布鲁氏菌的常用自杀载体pUC19质粒进行了改建, 在其中插入反向筛选基因sacB, 得到pUC19-SacB. 并且, 利用该质粒成功的构建Ⅳ型分泌系统编码基因virB的无痕缺失突变株.
布鲁氏菌B. melitensis 55 009购自中国药品生物制品鉴定所, DH5α和pUC19质粒及pKOBEG-SacB质粒均为本室保存[7]. DNA胶回收试剂盒及质粒提取试剂盒购自Promega公司, 各种限制性内切酶均购自TaKaRa公司, T4 DNA连接酶购自NEB公司.
1.2.1 自杀质粒pUC19-SacB的改建: pKOBEG-SacB质粒用NdeⅠ进行酶切后, 胶回收sacB片段, 与同样酶切处理的pUC19质粒连接, 得到pUC19-SacB. 对质粒进行PCR和酶切鉴定, 并送Invitrogen公司进行测序. 测序正确后, 为进一步验证sacB基因的有效性, 将携带有pUC19-SacB质粒的DH5α在无抗性培养基中培养4 h, 稀释后分别等量涂布含有氨苄青霉素和含有50 g/L蔗糖的平板. 比较两块平板上的克隆数, 如果蔗糖板上的克隆数远远低于抗性板, 则说明sacB基因是有效的.
1.2.2 利用pUC19-SacB载体构建Ⅳ型分泌系统缺失突变株: 1.2.2.1 缺失引物的设计 设计两对缺失引物分别用于扩增Ⅳ型分泌系统启动子的上游和下游片段, 并通过融合PCR将这两个片段融合在一起, 从而得到缺失启动子区域511bp(-510-+1)的缺失突变盒. 此外, 设计一对引物PRO-F和PRO-R用于确定启动子的缺失; 一对引物针对31 kDa蛋白基因, 用于鉴定布鲁氏菌; 一对引物针对pUC19-SacB质粒上的sacB基因, 用于确定质粒的消除. 设计的引物见表1.
| 引物名 | 引物序列(5'-3') |
| IVB-N-F | CTGCGAAGCTTGCAAATTCCCGTCCGGTTCG |
| IVB-N-R | GAGGACAAGGAATGGCACCACGACGCAGGACGGAAAGGAC |
| IVB-C-F | GTCCTTTCCGTCCTGCGTCGTGGTGCCATTCCTTGTCCTC |
| IVB-C-R | CGACCGGAATTCGAAGCCGCCCGTAAAGTTGC |
| PRO-F | GAGCGGCTGGAACTGCAAAC |
| PRO-R | GACCAACCGCCCACCAACGAC |
| 31KD-F | GGCGGCAAGTTCAAGCATC |
| 31KD-R | GGTCGGTGTAGAGGTATTCC |
| sacB-F | TCGCATTATCCGAACCATCC |
| sacB-R | CACCCAGTCCCAGACGAAGC |
1.2.2.2 缺失突变载体的构建 用IVB-N-F和IVB-N-R扩增Ⅳ型分泌系统启动子的上游片段, 用IVB-C-F和IVB-C-R扩增下游片段, 切胶回收PCR产物, 等量混合, 稀释后作为第二轮PCR反应的模板. 第二轮扩增的引物为IVB-N-F和IVB-C-R, 扩增产物切胶回收, 得到缺失突变盒IVB-NC. 用HindⅢ和EcoRⅠ对IVB-NC进行双酶切, 然后与构建好的自杀载体pUC19-SacB质粒连接, 得到缺失突变载体pUCSacB-IVB.
1.2.2.3 缺失突变体的构建与鉴定 将0.8 μg pUCSacB-IVB质粒与100 μL布鲁氏菌感受态细胞充分混合以后, 预冷10 min, 随后将混合物加入冷却的电击杯狭缝中, 电击转化参数为1.8 KV/cm, 400 Ω. 电击以后, 迅速向电击杯中加入1 mL SOC培养基, 转移至试管中37℃振摇20 h, 随后涂含100 mg/L氨苄青霉素的TSA平板, 37℃培养3-4 d, 对长出的抗性克隆进行菌落PCR鉴定. PCR鉴定的靶标基因包括布鲁氏菌特异的31 kDa蛋白的基因、质粒DNA的sacB基因和突变盒的IVB片段, 并用PRO-F和PRO-R鉴定启动子的缺失情况. 四者均能扩增出预期大小的片段即为阳性重组克隆. 为获得无痕缺失突变体, 将重组克隆转接至无抗性的TSB液体培养基中, 培养5 h后进行倍比稀释, 涂布蔗糖平板, 37℃培养3-4 d, 随机挑取16个蔗糖抗性克隆, 用上述4对引物进行鉴定. sacB基因扩增结果为阴性, 而其余3对引物均能扩出预期大小片段的克隆即为我们所需的无痕缺失突变体.
为了提高布鲁氏菌遗传操作的效率, 我们对pUC19质粒进行了改建, 在该质粒中插入一个反向筛选基因, 使其能用于染色体的精确修饰. 用NdeⅠ内切酶从pKOBEG-SacB质粒中切下sacB基因, 胶回收后与同样酶切处理的pUC19质粒连接, 转入大肠杆菌DH5α中, PCR、酶切鉴定以及测序结果表明sacB正确插入到了pUC19质粒中, 我们成功的构建了自杀载体pUC19-SacB. 图1是改建得到的pUC19-SacB的质粒图谱.
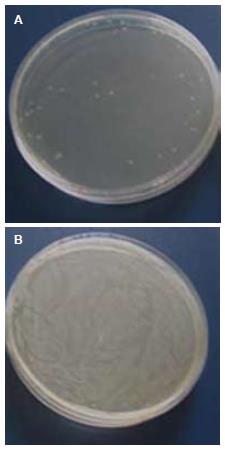
观察氨苄青霉素平板和蔗糖平板上的克隆数, 发现蔗糖平板上的克隆数远远低于抗性平板的克隆数, 大约为0.01%(图2), 表明改建的pUC19-SacB质粒中的sacB基因具有非常高的反向筛选能力.
为了验证pUC19-SacB自杀质粒的功能, 我们利用pUC19-SacB成功的构建了布鲁氏菌Ⅳ型分泌系统编码基因virB的无痕缺失突变株. 按照材料方法中描述的步骤, 我们构建了virB的缺失突变载体, 将其通过电转化导入布鲁氏菌中, 经过两轮筛选, 成功的筛选到了virB启动子缺失的无痕缺失突变株. PCR和DNA测序结果证实, virB无痕缺失突变株构建正确.
布鲁氏菌是一个比较特殊的胞内寄生菌, 他没有质粒, 而且大多其他来源的质粒也不能在布鲁氏菌中复制. 据此推理,很多质粒都可作为自杀载体来构建布鲁氏菌的突变株. pUC19是一种常见的克隆载体, 主要用于布鲁氏菌插入突变株的构建[8]. 虽然其重组效率较高, 但是插入失活的策略不能用于分析染色体特定区域的功能, 特别是分析操纵子中的个别基因的功能, 因为插入的外源载体序列会影响基因其他部分的功能, 从而不能确定表型的变化是否真实[9-10]. 对于这类情况, 需要缺失目的基因编码框的一部分, 并且不能带有载体序列, 即必须构建无痕缺失突变株. 一般来说, 细菌无痕缺失突变株的构建主要是利用带有反向筛选基因的自杀质粒来实现的[11]. 我们在前期的研究中采用经典的自杀质粒来构建无痕缺失突变株. 但是, 这些自杀载体存在一定的不足, 如: 分子操作需要在特殊的宿主菌中进行、重组的效率较低, 因此, 布鲁氏菌无痕缺失突变株的构建往往比较困难. sacB作为反向筛选基因, 在革兰氏阴性菌突变株的构建中应用非常广泛. 表达该蛋白的革兰氏阴性菌在含有蔗糖的培养基中生长时, 会将蔗糖分解为对细菌有毒的产物, 从而杀死表达该蛋白的细菌. 因此, sacB作为反筛基因常与抗性基因等正向筛选基因联合, 用于构建细菌的无痕缺失突变株[12]. pEX18Gm是一种含有反向筛选基因sacB的克隆载体[13], 在前期的研究中, 我们利用该载体作为自杀载体成功的构建了布鲁氏菌的突变株. 但是, 我们在应用该载体时发现了一些问题, 利用蔗糖抗性消除质粒得到的无痕缺失突变株由于一些不清楚的原因在体外培养一段时间后部分克隆又丢失了蔗糖抗性. 为此, 在本研究中, 我们对布鲁氏菌常用质粒pUC19进行了改建, 在该质粒中插入一个反向筛选基因sacB, 构建了pUC19-SacB. 蔗糖敏感性实验证实构建的载体反向筛选效率很高. 我们利用改建的载体, 成功的构建了virB无痕缺失突变株, 这不仅证实了pUC19-SacB自杀质粒的功能, 也为深入研究VirB操纵子的功能奠定了基础. 此外, 利用改建的载体, 我们还构建了多个其他基因的无痕缺失突变株, 包括双组分系统BvrR/BvrS的突变株和Omp25基因的突变株. 这些突变株的稳定性较好, 没有发生蔗糖抗性丢失的情况. 试验结果表明, pUC19-SacB不仅保持了pUC19的高效重组效率, 而且反筛效率也很高, 是一个非常高效的自杀质粒, 可以为布鲁氏菌基因功能的研究提供了一个有力的分析工具. 有了快速高效的构建突变株的方法, 必将大大加速布鲁氏菌基因功能研究的步伐.
布鲁氏菌危害严重, 在世界各地都有广泛流行. 一直以来, 致病机制研究都是布鲁氏菌研究的重点, 而突变株的构建是致病机制研究的前提.
克隆载体pUC19主要用于布鲁氏菌插入突变株的构建, 而精确的缺失则是采用经典的自杀载体来构建的, 如pCVD442等. 但是, 这类载体的分子操作比较困难.
为了提高布鲁氏菌遗传操作的效率, 本文对pUC19质粒进行了改建, 在该质粒中插入一个反向筛选基因, 使其能用于染色体的精确修饰.
本文构建pUC19-SacB载体, 并证实其反向筛选效率很高, 且利用其构建布鲁氏菌的突变株, 技术方法先进, 有一定的参考价值和指导意义.
编辑: 程剑侠 电编:李军亮
| 1. | Pappas G, Akritidis N, Bosilkovski M, Tsianos E. Brucellosis. N Engl J Med. 2005;352:2325-2336. [PubMed] |
| 2. | Cutler SJ, Whatmore AM, Commander NJ. Brucellosis-new aspects of an old disease. J Appl Microbiol. 2005;98:1270-1281. [PubMed] |
| 3. | Baek SH, Rajashekara G, Splitter GA, Shapleigh JP. Denitrification genes regulate Brucella virulence in mice. J Bacteriol. 2004;186:6025-6031. [PubMed] |
| 4. | Ekaza E, Teyssier J, Ouahrani-Bettache S, Liautard JP, Kohler S. Characterization of Brucella suis clpB and clpAB mutants and participation of the genes in stress responses. J Bacteriol. 2001;183:2677-2681. [PubMed] |
| 5. | Burkhardt S, Jimenez de Bagues MP, Liautard JP, Kohler S. Analysis of the behavior of eryC mutants of Brucella suis attenuated in macrophages. Infect Immun. 2005;73:6782-6790. [PubMed] |
| 6. | Michaux-Charachon S, Jumas-Bilak E, Allardet-Servent A, Bourg G, Boschiroli ML, Ramuz M, O'Callaghan D. The Brucella genome at the beginning of the post-genomic era. Vet Microbiol. 2002;90:581-585. [PubMed] |
| 7. | Derbise A, Lesic B, Dacheux D, Ghigo JM, Carniel E. A rapid and simple method for inactivating chromosomal genes in Yersinia. FEMS Immunol Med Microbiol. 2003;38:113-116. [PubMed] |
| 8. | den Hartigh AB, Sun YH, Sondervan D, Heuvelmans N, Reinders MO, Ficht TA, Tsolis RM. Differential requirements for VirB1 and VirB2 during Brucella abortus infection. Infect Immun. 2004;72:5143-5149. [PubMed] |
| 9. | Boschiroli ML, Ouahrani-Bettache S, Foulongne V, Michaux-Charachon S, Bourg G, Allardet-Servent A, Cazevieille C, Liautard JP, Ramuz M, O'Callaghan D. The Brucella suis virB operon is induced intracellularly in macrophages. Proc Natl Acad Sci USA. 2002;99:1544-1549. [PubMed] |
| 10. | Ortiz-Martin I, Macho AP, Lambersten L, Ramos C, Beuzon CR. Suicide vectors for antibiotic marker exchange and rapid generation of multiple knockout mutants by allelic exchange in Gram-negative bacteria. J Microbiol Methods. 2006;67:395-407. [PubMed] |
| 11. | Hoang TT, Karkhoff-Schweizer RR, Kutchma AJ, Schweizer HP. A broad-host-range Flp-FRT recombination system for site-specific excision of chromosomally-located DNA sequences: application for isolation of unmarked Pseudomonas aeruginosa mutants. Gene. 1998;212:77-86. [PubMed] |
| 12. | Reyrat JM, Pelicic V, Gicquel B, Rappuoli R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infect Immun. 1998;66:4011-4017. [PubMed] |
| 13. | Choi KH, Schweizer HP. An improved method for rapid generation of unmarked Pseudomonas aeruginosa deletion mutants. BMC Microbiol. 2005;5:30. [PubMed] |