修回日期: 2007-09-13
接受日期: 2007-09-28
在线出版日期: 2007-09-28
目的: 通过筛选原发性肝癌血清蛋白结合蛋白, 探究肝癌的分子发病机制.
方法: 应用噬菌体展示技术, 以肝癌患者的血清作为固相筛选分子, 对T7 Select 噬菌体人肝细胞cDNA文库进行5轮"吸附-洗脱-扩增"的筛选过程, 经噬斑的聚合酶链式反应(PCR)扩增后, 对阳性PCR产物直接进行序列测定和同源性分析.
结果: 噬菌体经富集后, 随机挑取48个噬斑进行PCR扩增, 得到5种大小不同的PCR片断, 序列测定后经序列同源性分析, 结果发现与肝癌患者血清蛋白结合的蛋白有以下5种: 人核仁螺旋体磷酸化蛋白NOLC1, 人高度迁移基核小体结合域1(HMGN1), 人依赖ATP的DNA连接酶1(LIG1), 人小EDRK-丰富因子2(SERF2)和A-kinase anchor protein 9 isoform.
结论: 用噬菌体人肝cDNA文库筛选得到了肝癌血清蛋白结合蛋白, 为进一步阐明肝癌的发生及发病机制奠定了基础.
引文著录: 刘志英, 魏红山, 张黎颖, 成军. 应用噬菌体展示技术筛选原发性肝癌血清结合蛋白. 世界华人消化杂志 2007; 15(27): 2914-2917
Revised: September 13, 2007
Accepted: September 28, 2007
Published online: September 28, 2007
AIM: To screen for serum binding proteins in patients with primary hepatocellular carcinoma and to investigate the tumor molecular mechanisms of the primary hepatocellular carcinoma.
METHODS: Serum protein of the primary hepatocellular carcinoma was used as the selective molecule and the T7 select phage display system from a human liver cDNA library was screened through adsorption, elution and amplification for 5 cycles. After polymerase chain reaction of bacteriophage plaques, the DNA fragments of the screened clones were sequenced, and the amino acid sequences of target proteins were compared with the GenBank protein sequence database using basic local alignment search tool (BLAST).
RESULTS: After amplification for 5 cycles, 5 positive clones were obtained from 48 randomly screened clones. The sequences of positive clones were investigated for homology. Homo sapiens nucleolar and coiled-body phosphoprotein 1 (NOLC1), Homo sapiens high-mobility group nucleosome binding domain 1 (HMGN1), Homo sapiens ligase I (LIG1), A-kinase anchor protein 9 isoform, and Homo sapiens small EDRK-rich factor 2 (SERF2) were identified as proteins binding to the serum protein in patients with primary hepatocellular carcinoma.
CONCLUSION: The current results of screening for proteins binding to serum protein in patients with primary hepatocellular carcinoma are useful for further studies of the molecular mechanisms of primary hepatocellular carcinoma.
- Citation: Liu ZY, Wei HS, Zhang LY, Cheng J. Screening for serum binding proteins in subjects with primary hepatocellular carcinoma using a phage display system. Shijie Huaren Xiaohua Zazhi 2007; 15(27): 2914-2917
- URL: https://www.wjgnet.com/1009-3079/full/v15/i27/2914.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i27.2914
原发性肝癌是我国最常见的恶性肿瘤之一, 占癌症死亡率的第2位. 导致肝癌的主要因素包括HBV和HCV慢性感染, 食物中的黄曲霉毒素及嗜酒等. 多种遗传和后天因素参与了HCC的分子发病机制, 如p53抑癌基因的突变和WNT信号转导途径的激活等[1], 肝癌的分子发病机制是当前肝癌研究的重点所在.
噬菌体展示技术是一种基因表达产物和亲和选择相结合的技术, 他以改构的噬菌体为载体, 把待选基因片段定向插入噬菌体外壳蛋白质基因区, 使外源多肽或蛋白质表达并展示于噬菌体表面, 进而通过亲和富集法表达有特异肽或蛋白质的噬菌体. cDNA 文库的表达筛选系统除利用λ噬菌体、酵母双杂交系统外, 噬菌体表面展示技术也可达到同样的目的[2-4]. 本研究中, 我们用正常人血清做筛选对照, 用人肝细胞cDNA文库对肝癌患者血清蛋白进行特异性筛选, 为研究肝癌的发生与发病机制提供了线索.
T7Select人肝细胞cDNA 文库、受体菌BLT5615(Novagen 公司), 10份人肝癌血清标本均来自我院住院肝癌患者, 符合第4届全国肝癌学术会议制订的肝癌诊断标准; 另取10份健康人血清做正常对照, 血清经静脉采血获得.
1.2.1 文库扩增: 动物术前禁食12 h, 自将BLT5615 接种于3 mL LB/Amp, 220 r/min, 37℃过夜; 在3 mL LB/Amp内加入振摇细菌30 μL, 将细菌浓度摇至A600 = 0.5, 加入1 mol/ L IPTG 3 μL诱导, 37℃振摇30 min 后加入噬菌体文库5 μL; 37℃振摇1-3 h直到观察到细菌裂解; 8000 g离心10 min, 将上清移至另一无菌管中, 4℃保存.
1.2.2 生物筛选: 分别将10份等量肝癌患者血清和健康人血清混匀后分装成100 μL/份, 贮存在-20℃冰箱内. 取混合好的肝癌血清80 μL, 加入NaHCO3(pH9.6)包被液320 μL中, 混匀, 包被24孔聚丙烯酰胺培养板, 4℃放置48 h; 加入T7 Select人肝细胞 cDNA 文库250 μL, 4℃过夜. 吸弃上清液, 加入1×TBST(成分为三羟甲基氨基甲烷-HCl-吐温20, pH 7.5) 缓冲液1 mL洗液漂洗5次, 10 g/L SDS洗脱液400 μL室温孵育20 min, 反复吹打洗涤包被板, 将洗脱液移至另一无菌管中. 将洗脱噬菌体10 μL加入对数生长期的细菌培养液3 mL, 37℃振摇培养, 直到看到细菌裂解. 8000 g离心10 min, 将上清移到一新管, 4℃保存备下一轮筛选用. 再取混合后的正常血清80 μL, 加入包被液320 μL震荡混匀, 包被于24孔板4℃ 48 h后, 1×TBST洗涤5遍, 加入前一步细菌裂解液用于筛选. 上述两步生物筛选为1轮. 每轮筛选后, 均做噬斑分析. 按上述步骤重复筛选5 次. 计算产出率, 产出率 = 洗脱的噬菌体数量/所用的噬菌体数量.
1.2.3 噬斑PCR扩增及分析: 选择第5轮正常对照10-8稀释梯度的30个阳性噬斑, 进行噬菌体裂解, 裂解液进行PCR扩增, 引物序列: 5'-TTATCGGATAACCCTTTATA-3'(上游), 5'-CCCTCAAGACCCGTTTAGAG-3'(下游), 一部分PCR产物用10 g/L琼脂糖凝胶电泳. 另一部分PCR产物直接送上海生工公司进行序列测定, 序列结果发到美国国立卫生院GenBank数据库(http://www.ncbi.nlm.nih.gov), 由BLAST n软件完成.
以肝癌患者血清为固相分子, 对人类正常肝脏噬菌体cDNA细胞文库进行5轮"包被-结合-洗脱-扩增"筛选, 噬菌体的富集结果见表1.
| 筛选次数 | 投入量(nfu/L) | 产出量(nfu/L) | 产出率(%) |
| 1 | 2×109 | 8×1010 | 40 |
| 2 | 3×1010 | 6×1010 | 2 |
| 3 | 3×1010 | 4×1010 | 1.3 |
| 4 | 2×1010 | 8×109 | 0.4 |
| 5 | 6×109 | 3×109 | 0.5 |
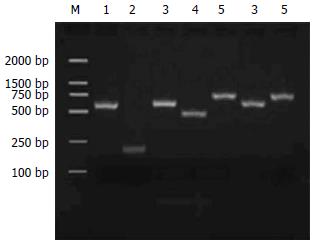
经过5轮筛选, 随机挑选30个噬斑为模板进行PCR扩增, 序列测定和同源性分析后, 得到5个与肝癌患者血清蛋白结合的蛋白 (图1, 表2).
| 同源序列编码蛋白 | GenBank收录号 | 核苷酸位置(nt) | 同源性 |
| NOLC1 | NM_004741.1 | 1219-1869 | 99% |
| LIG1 | NM_000234.1 | 107-530 | 99% |
| HMGN1 | NM_004965 | 128-668 | 100% |
| A-kinase anchor protein 9 isoform | NT_007933 | 529-749 | 99% |
| SERF2 | NM-001018108.1 | 28-485 | 100% |
肝癌是世界上最常见的恶性肿瘤之一, 迄今为止, 尚没有发现行之有效的治疗方案, 肝癌的诊断和治疗是肿瘤研究领域的一个主要焦点. 本研究中, 我们用正常人血清做筛选对照, 用噬菌体表面展示技术筛选了肝癌患者血清特异结合蛋白, 我们发现了包括NOLC1、HMGN1、Homo sapiens ligase I、SERF2、A-kinase anchor protein 9 isoform在内的5种肝癌血清特异结合蛋白.
有效的修复DNA的损伤是保证基因表达忠实性和防止突变而导致疾病的关键因素. HMGN1是一个核仁结合蛋白, 属于核小体结合高速迁移蛋白家族(A family of High Mobility Group Proteins that bind to Nucleosomesn, HMGN)含量丰富的蛋白, 能稳定染色质的二级结构, 可以修复紫外线诱导的染色质DNA损伤[5].
Birger et al[5]提出HMGN1的缺失可导致细胞增殖和生长发生紊乱, 通过打乱细胞的生长控制机制使裸鼠对一些致癌因素更为敏感并发生恶性转化. 最近研究表明, HMGN1在细胞核内是动态表达, 与细胞的新陈代谢有关. HMGN1蛋白和核仁的相互作用可改变染色质的结构, 并且能调节各种与DNA相关的包括转录的细胞核作用[6]. 本研究中, 我们用肝癌患者血清做固相包被物, 筛选出与肝癌患者血清蛋白特异结合蛋白HMGN1, 根据已报道的HMGN1的功能, 推测肝癌患者血清里某种或某几种蛋白与核仁的HMGN1蛋白发生了特异作用, 导致了HMGN1的维护染色质结构的功能失常而导致了细胞非正常的生长和增殖作用. 该蛋白的发现为进一步研究肝癌的发病机制提供了重要的线索, 但是何种蛋白还需进一步验证和考核.
人核仁磷酸化蛋白NOLC1, 又名hNopp140, 是一个高度磷酸化的哺乳动物蛋白, 参与核仁的发生, 调节rDNA的转录[7]; 国内研究小组曾报道为HCV NS5A反式激活蛋白, 命名为NS5ATP13[8], 进一步用基因表达谱芯片技术对HCV NS5ATP13的研究证实, 上调的基因中包括细胞生长、炎症发生、肿瘤发生的相关调节基因[9]. 陈京龙 et al[10]发现隐源性肝炎患者血清中NS5ATP13的表达上调, Jin et al[11]用抗癌药阿霉素做固相包被物, 用T7肝细胞cDNA文库筛选与其特异结合蛋白, 证实hNopp140的C端能与阿霉素特异结合, 提示阿霉素有可能破坏hNopp140在细胞内的功能. 本研究中用肝癌患者血清做固相包被物, 同样用T7人肝细胞cDNA文库筛选技术, 再次发现NOLC1的C末端能与肝癌血清发生特异结合. 结合近期国内外关于该领域的研究, 提示NOLC1与肝癌的发生、发展或者治疗具有密切的关系, 但其生物学作用还有待深入的研究探讨.
在筛选出的已知功能蛋白中, 还有人DNA连接酶I(Homo sapiens ligase Ⅰ, LIG1), LIG1编码DNA 连接酶Ⅰ, 其功能是参与DNA的复制和碱基修复过程, LIG1突变可导致DNA连接酶I的缺乏进而导致免疫缺陷并且能提高DNA对损伤抗原的敏感度[12]. Landi et al[13]报道参与DNA错配修复的LIG1的基因多态性与肺癌的发生有很强的相关性; 同时Narayan et al[14]用DNA微阵列的方法发现宫颈癌患者体内LIG1基因表达明显上调. 结合本研究初步筛选结果, 提示LIG1与癌细胞的增殖有着密切的联系.
此外本实验还发现了SERF2(Small EDRK-rich factor 2, 小EDRK丰富因子2)和A-kinase anchor protein 9 isoform等肝癌血清特异结合蛋白, 但在肝癌发展中的具体作用还需进一步研究.
肝癌是世界上最常见的恶性肿瘤之一, 迄今为止, 尚没有发现行之有效的治疗方案, 肝癌的诊断和治疗是肿瘤研究领域的一个主要焦点.
国内研究小组曾报道HCV NS5A反式激活蛋白, 命名为NS5ATP13,进一步用基因表达谱芯片技术对HCV NS5ATP13的研究证实上调的基因中包括细胞生长、炎症发生、肿瘤发生的相关调节基因, 陈京龙 et al发现隐源性肝炎患者血清中NS5ATP13的表达上调, Jin et al用抗癌药阿霉素做固相包被物, 用 T7 肝细胞cDNA文库筛选与其特异结合蛋白, 证实hNopp140的C端能与阿霉素特异结合, 提示阿霉素有可能破坏 hNopp140在细胞内的功能.
本文首次将10份原发性肝癌患者的血清混合后用噬菌体展示技术筛选肝癌血清特异结合蛋白.
本文研究方法恰当, 采用的技术较先进, 结果结论较可信, 有一定的学术价值.
编辑: 何燕 电编:李军亮
| 1. | Hussain SP, Schwank J, Staib F, Wang XW, Harris CC. TP53 mutations and hepatocellular carcinoma: insights into the etiology and pathogenesis of liver cancer. Oncogene. 2007;26:2166-2176. [PubMed] |
| 2. | McGregor JC, Pollock JG, Anton HC. The value of ultrasonography in the diagnosis of abdominal aortic aneurysm. Scott Med J. 1975;20:133-137. [PubMed] |
| 3. | Crameri R, Walter G. Selective enrichment and high-throughput screening of phage surface-displayed cDNA libraries from complex allergenic systems. Comb Chem High Throughput Screen. 1999;2:63-72. [PubMed] |
| 4. | Crameri R, Kodzius R. The powerful combination of phage surface display of cDNA libraries and high throughput screening. Comb Chem High Throughput Screen. 2001;4:145-155. [PubMed] |
| 5. | Birger Y, Catez F, Furusawa T, Lim JH, Prymakowska-Bosak M, West KL, Postnikov YV, Haines DC, Bustin M. Increased tumorigenicity and sensitivity to ionizing radiation upon loss of chromosomal protein HMGN1. Cancer Res. 2005;65:6711-6718. [PubMed] |
| 6. | Hill DA, Peterson CL, Imbalzano AN. Effects of HMGN1 on chromatin structure and SWI/SNF-mediated chromatin remodeling. J Biol Chem. 2005;280:41777-41783. [PubMed] |
| 7. | Meier UT, Blobel G. Nopp140 shuttles on tracks between nucleolus and cytoplasm. Cell. 1992;70:127-138. [PubMed] |
| 8. | 党 晓燕, 成 军, 刘 妍, 邓 红, 杨 倩, 王 建军, 纪 冬, 王 春花. 丙型肝炎病毒非结构蛋白5A反式激活基因NS5ATP13的克隆化研究. 胃肠病学和肝病学杂志. 2003;12:260-262. |
| 9. | 张 黎颖, 邓 红, 成 军, 党 晓燕, 刘 妍, 蔺 淑梅, 黄 燕萍, 纪 冬, 吴 顺华, 纪 泛扑. 应用表达谱芯片技术研究NS5ATP13的反式调节基因. 世界华人消化杂志. 2004;12:2757-2761. [DOI] |
| 11. | Jin Y, Yu J, Yu YG. Identification of hNopp140 as a binding partner for doxorubicin with a phage display cloning method. Chem Biol. 2002;9:157-162. [PubMed] |
| 12. | Tomkinson AE, Chen L, Dong Z, Leppard JB, Levin DS, Mackey ZB, Motycka TA. Completion of base excision repair by mammalian DNA ligases. Prog Nucleic Acid Res Mol Biol. 2001;68:151-164. [PubMed] |
| 13. | Landi S, Gemignani F, Canzian F, Gaborieau V, Barale R, Landi D, Szeszenia-Dabrowska N, Zaridze D, Lissowska J, Rudnai P. DNA repair and cell cycle control genes and the risk of young-onset lung cancer. Cancer Res. 2006;66:11062-11069. [PubMed] |
| 14. | Narayan G, Bourdon V, Chaganti S, Arias-Pulido H, Nandula SV, Rao PH, Gissmann L, Durst M, Schneider A, Pothuri B. Gene dosage alterations revealed by cDNA microarray analysis in cervical cancer: identification of candidate amplified and overexpressed genes. Genes Chromosomes Cancer. 2007;46:373-384. [PubMed] |