修回日期: 2007-07-21
接受日期: 2007-07-28
在线出版日期: 2007-08-08
目的: 探讨基因表达谱芯片技术在筛查肝细胞癌(HCC)相关基因群表达中的作用.
方法: TRIzol法抽提2例HCC及正常肝脏组织总RNA, 分离纯化两种组织的mRNA; 逆转录合成掺入生物素标记的cDNA合成探针, 与基因芯片(涵盖了18 400个转录本, 代表了14 500个明晰的基因)杂交, 扫描芯片荧光信号图像, 计算机分析, 比较2种组织基因表达谱差异.
结果: 2例HCC组织与正常肝脏组织相比, 有2756条基因(19.01%)共同表达差异, 其中共同上调基因1772条和共同下调基因984条. 对2756条共同差异表达基因作了初步功能分类, 这些基因与HCC的发病机制存在相关性.
结论: 基因表达谱芯片技术可以筛选出HCC表达异常的相关基因群, 有助于认识肝癌的发病机制.
引文著录: 张弘, 黄介飞, 黄晓平, 黄东风, 魏群, 鲍柏军, 华婷琰. 应用基因芯片技术筛选肝细胞癌相关基因. 世界华人消化杂志 2007; 15(22): 2387-2393
Revised: July 21, 2007
Accepted: July 28, 2007
Published online: August 8, 2007
AIM: To ascertain the effectiveness of genechip for screening the same differentially expressed genes of hepatocellular carcinoma (HCC).
METHODS: Total RNAs were isolated from tissues of two cases of HCC and one normal liver by the TRIzol method, and then purified, reverse-transcribed to cDNA incorporating a biotin-labeling probe, and hybridized with genechip (covering 18 400 transcripts, representing 14 500 distinct genes). Picture signals of the fluorescence in the gene array were scanned and compared by computer analysis to the differential expression of the genes in the two tissues.
RESULTS: The same differentially expressed genes were 2756 (19.01%) and the same up regulated and downregulated genes were 1772 and 984, respectively, in two HCC and one normal liver tissues. These genes were initially classified according to function, and further analysis showed that they play an important role in the pathogenesis of HCC.
CONCLUSION: The analysis of the gene expression profile of HCC using genechip can rapidly select associated abnormal genes, while further analysis of the obtained genes can help elucidate the pathogenicity of HCC.
- Citation: Zhang H, Huang JF, Huang XP, Huang DF, Wei Q, Bao BJ, Hua TY. Genechip used for screening differentially expressed genes in hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi 2007; 15(22): 2387-2393
- URL: https://www.wjgnet.com/1009-3079/full/v15/i22/2387.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i22.2387
肝细胞癌(hepatocellular carcinoma, HCC)是我国较为常见的恶性肿瘤, 占肿瘤发病率中的第3位. 该病具有较高的死亡率, 占恶性肿瘤死亡的第2位. HCC未能切除者1 年生存率低于30%, 即使行根治术, 5 年复发率达80%左右, 小HCC的复发率也高达40%-50%, 可见其治疗的关键在于早期发现和早期诊断. 基因芯片技术是近几年发展起来的一项新的技术, 可在一次实验中快速, 准确, 高效的检测成千上万种基因的表达, 是探知基因在肿瘤组织与正常组织中表达差异的重要手段, 现已被广泛地用于肿瘤发生的分子生物学研究领域. 我们通过基因表达谱芯片技术检测HCC和正常肝脏组织中基因群表达的不同, 筛选出相关的基因群, 为进一步深入研究其在肝癌的发生、发展中的作用提供新的线索.
HCC组织标本2例由南通大学附属医院手术切除组织提供, 正常肝组织1例由上海生物芯片有限公司-生物芯片上海国家工程研究中心提供. HCC组织取肿瘤中无坏死、出血及淤胆的组织. 分别于标本切取后立刻(10 min内)将所取组织放入液氮中冻存备用. 所有标本均由病理证实. 所用基因芯片为美国Affymetrix公司的U133 A 2.0基因表达谱芯片, 由上海生物芯片有限公司-生物芯片上海国家工程研究中心提供. Affymetrix U133 A 2.0基因表达谱芯片涵盖了18 400个转录本(RNA), 代表了14 500个明晰的基因.
每例切取肝组织50-100 mg, 再加入TRIzol 1 mL, 然后用匀浆器将组织彻底打碎. 均质化的肝组织在15-30℃放置5 min, 然后加入0.2倍体积的氯仿, 盖子盖紧后剧烈振摇15 s混匀, 15-30℃放置2-3 min, 4℃ 5600 g离心15 min, 离心后形成下层红色的酚氯仿相, 中间相和上层无色水相. 上层水相转移至一个新的离心管(水相的体积大约为均质化时所用TRIzol体积的60%), 加入0.8倍体积的异丙醇, 剧烈振摇使混匀, 4℃ 5600 g离心15 min, 弃上清. 加入700 mL/L酒精, 颠倒离心管数次以洗去沉淀所含盐分. 如沉淀较大可用Tip头先将沉淀打散. 然后4℃ 5600 g离心10 min, 弃上清. 重复以上步骤1次. 15-30℃晾干, 然后加入适量的无RNase的水溶解. 用分光光度计分析RNA浓度, 在260 nm 1A = 40 mg/L的RNA. A260/A280应接近2.0为较纯的RNA(比值在1.9-2.1均可). 10 g/L琼脂糖电泳按Qiagen Oligotex Direct mRNA kit提供的操作手册提取Poly(A)+mRNA.
1.2.1 由RNA合成双链DNA第1链cDNA合成: 在Eppendorf管中加入逆转录试剂, 然后70℃温浴10 min, 稍微离心, 立即放置冰上至少2 min. 在相应Eppendorf管中再加入dNTP及缓冲液, 混合均匀, 42℃温浴1 h, 反应结束放置冰上至少2 min. 第2链DNA合成在第1链合成的管中加入第2链反应试剂, 混匀后稍微离心, 16℃放置2 h. 加2 μL 10 U/μL T4 DNA聚合酶, 16℃放置5 min. 再加0.5 mol/L EDTA 10 μL终止反应. 纯化双链DNA, 在合成的双链cDNA产物中加入cDNA Binding Buffer 600 μL, 然后混匀待用. 把500 μL样品加入cDNA Cleanup Spin Column, ≥8000 g离心1 min, 弃流出液. 把剩余的样品加入离心柱, 以上述同样的方法离心. 弃流出液和收集管. 把离心柱转移至一个新的收集管. 加入cDNA Wash Buffer 750 μL至离心柱内. ≥8000 g离心1 min, 弃流出液. 打开离心柱管盖, 以最大速度离心5 min(≥25 000 g), 弃流出液和收集管. 打开管盖离心可以确保离心柱内膜的干燥. 把离心柱转移至一个新的1.5 mL收集管, 加入cDNA Elution Buffer 14 μL. 室温下静置1 min. 以最大转速(≥25 000 g)离心1 min洗脱DNA. 一般14 μL的洗脱液可以收集到约12 μL流出液. 严格按Qiagen RNeasy Total RNA Isolation kit试剂盒提供的操作手册纯化生物素标记的cRNA. 用分光光度计分析RNA浓度.
1.2.2 杂交和分析: 芯片使用前需平衡至室温, 杂交炉中45℃, 60 r/min预杂交芯片10 min. 配制杂交液, 加等体积处理好的杂交液, 然后在杂交炉中45℃, 60 r/min杂交芯片16 h. 把杂交好的芯片进行洗脱、染色, 最后扫描芯片. 用Affymetrix 扫描仪扫描芯片后, GCOS1.2软件读取、处理数据. 差异基因筛选标准: change为Increase或Marginal Increase, log ratio(实验组和对照组比值取Log2后的值)≥1, 实验组(肝癌组)Detection为Present的为上调基因; change为Decrease或Marginal Decrease, log ratio≤-1, 对照组(正常组)Detection为Present的为下调基因.
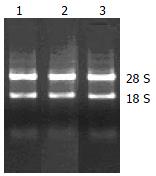
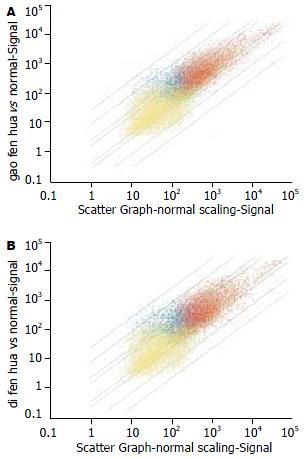
所抽提的总RNA, 经分光光度计检测A260, A280的吸光度, 确定样品的浓度和纯度. 算得A260/A280均>1.9, 表明所提取总RNA纯度较高, 而且没有蛋白质和DNA残留. RNA样品高分化HCC为1.256 g/L; 低分化HCC为1.465 g/L; 正常肝组织为1.806 g/L; cRNA A值分别为1.93, 1.95, 1.94.经10 g/L甲醛变性琼脂糖凝胶电泳后, 18 S和28 S电泳条带清晰, 28 S条带无明显降解(图1), 说明总RNA质量完好, 结果合格, 可以进行芯片实验. 2例基因芯片检测结果显示, 符合质控标准, 信号强度达到要求, 5'/3'比值正常. 正常肝组织cRNA、HCC组织cRNA分别与含18 400条转录本的芯片杂交, 图2A, B以散点图的形式描述了HCC组织/正常肝组织对应的184 00条转录本的信号平均强度. 按差异显著性标准从18 400个转录本(代表了14 500个明晰的基因)中筛选出在HCC组织中共同差异表达基因共有2756条, 占筛选基因总数的19.01%, 其中上调表达的基因有1772条, 下调表达的基因有984条.
两例HCC组织中共同差异表达的基因中有1772条基因上调包括抗氧化活性相关基因、凋亡或肿瘤相关基因、结合蛋白相关基因、代谢相关基因、细胞周期相关基因、酶活性调节相关基因、分子马达相关基因、营养储备相关基因、分子功能退化相关基因、信号传导相关基因、结构分子相关基因、转录调控相关基因、翻译调控相关基因和物质运输相关基因. 在2例组织中未发现共同上调的包括未知分子功能相关基因和分子伴侣调控相关基因(表1). 两例肝癌组织中共同差异表达的基因中有984条基因下调. 包括抗氧化活性相关基因、凋亡或肿瘤相关基因、结合蛋白相关基因、代谢相关基因、细胞周期相关基因、酶活性调节相关基因、分子马达相关基因、分子功能退化相关基因、信号传导相关基因、结构分子相关基因、转录调控相关基因、翻译调控相关基因和物质运输相关基因. 在2例组织中未发现共同下调的分子伴侣调控相关基因、未知分子功能相关基因和营养储备相关基因(表2). 很多基因具有多功能, 参与细胞周期, 信号传导, 结合蛋白, 转录, 翻译, 凋亡. 这些基因均至少参与两种不同的细胞活动过程(表3).
| 功能分类 | GenBank ID Log Ratio | 基因名称 | 基因代码 | 染色体定位 | Signal |
| 抗氧化活性 | NM_002083 | 谷胱甘肽过氧化物酶 | GPX2 | chr14q24.1 | 2.7 |
| NM_003330 | 硫氧还蛋白还原酶 | TXNRD1 | chr12q23 | 3.9 | |
| 凋亡或肿瘤相关 | NM_001467 | v-myc | MYC | chr8q24.12 | 1.4 |
| NM_013250 | r-ras癌基因同系物2 | RRAS2 | chr11p15.2 | 4.7 | |
| NM_003350 | 死亡相关蛋白 | DAXX | chr6p21.3 | 3.1 | |
| AI582238 | 肿瘤排斥抗原 | TRA1 | chr12q24.2 | 2.1 | |
| 结合蛋白相关 | NM_012325 | 微管相关蛋白 | MAPRE1 | chr20q11.1 | 1.8 |
| NM_001903 | 细胞选择蛋白α1 | CTNNA1 | chr5q31 | 3.0 | |
| NM_006396 | 核仁蛋白5A | NOL5A | chr20p13 | 1.6 | |
| 代谢相关 | NM_021299 | 醛酮还原酶 | AKR1B10 | chr7q33 | 1.7 |
| NM_002402 | 葡萄糖-6-磷酸脱氢酶 | G6PD | chrXq28 | 6.4 | |
| NM_003745 | 丝裂原活化蛋白激酶1 | MAPK1 | chr22q11.2 | 1.5 | |
| NM_004396 | RNA解旋酶 | DDX5 | chr17q21 | 2.1 | |
| 细胞周期相关 | NM_002786 | 细胞分裂周期2 | CDC2 | chr10q21.1 | 3.7 |
| NM_003237 | 细胞周期素 A2 | CCNA2 | chr4q25-q31 | 3.3 | |
| NM_001592 | 增殖细胞核抗原 | PCNA | chr20pter-p12 | 1.6 | |
| 酶活性调节 | NM_002734 | cAMP依赖的蛋白激酶 | PRKARIA | chr17q23 | 2.0 |
| NM_004039 | 钙结合蛋白A2 | ANXA2 | chr15q21 | 3.3 | |
| NM_006241 | 蛋白磷酸酶1调节亚单位2 | PPP1R2 | chr2q29 | 1.6 | |
| 分子马达相关 | AF250307 | 细胞质动力蛋白 | DNCI | chr2Q31.1 | 1.0 |
| BF432550 | 肌球蛋白IB | MYOIB | chr2q12 | 1.3 | |
| NM_016141 | 细胞质动力蛋白多肽1 | DNCLI1 | chr3p23 | 2.0 | |
| 营养储备相关 | NM_005637 | 关节肉瘤 | SS18 | chr18q11.2 | 2.8 |
| 退化分子功能相关 | AI761759 | 钙联接蛋白 | CANX | chr5q35 | 1.3 |
| AU146237 | 剪接因子11 | SFRS11 | chr1p31 | 2.2 | |
| U40763 | 亲环蛋白G | PPIG | chr2q31.1 | 2.2 | |
| 信号传导相关 | NM_007354 | 肿瘤相关钙信号转导物1 | TACSTD1 | chr2p21 | 4.8 |
| NM_001315 | 信号传导子及转录激活子1 | STAT1 | chr2q32.2 | 1.9 | |
| NM_002875 | 胰岛素样生长因子1受体 | IGF1R | chr15q26.3 | 3.9 | |
| 结构分子相关 | NM_005582 | 分泌型磷蛋白1 | SPP1 | chr4q21-q25 | 4.2 |
| NM_007227 | 层粘蛋白α3 | LAMA3 | chr18q11.2 | 3.7 | |
| NM_001224 | 角蛋白18 | KRT18 | chr12q13 | 1.6 | |
| 转录调控相关 | NM_006315 | 信号传导子及转录激活子1 | STAT1 | chr2q32.2 | 1.9 |
| NM_014739 | BCL2相关转录因子1 | BCLAF1 | chr6q22 | 2.0 | |
| NM_001518 | 转录因子Ⅱ | GTP2L | chr7q11.2 | 1.4 | |
| 翻译调控相关 | NM_015904 | 真核翻译起始因子5B | EIF5B | chr2p11.1 | 4.0 |
| NM_004730 | 真核翻译起始因子1 | EIF1 | chr5q31.1 | 2.0 | |
| NM_002453 | 线粒体翻译起始因子2 | MTIF2 | chr2p14 | 2.3 | |
| 转运活性相关 | NM_003100 | 分类连接蛋白2 | SNX2 | chr5q23 | 2.4 |
| NM_001444 | 脂肪酸结合蛋白5 | FABP5 | chr8q21.1 | 6.6 | |
| AI816243 | 突触融合蛋白12 | STX12 | chr1p35 | 2.0 |
| 功能分类 | GenBank ID Log Ratio | 基因名称 | 基因代码 | 染色体定位 | Signal |
| 抗氧化活性相关 | NM_000962 | 前列腺素内过氧化物合酶1 | PTGS1 | chr9q32-q33.3 | -1.2 |
| BC001453 | 谷胱甘肽转移酶zeta1 | GSTZ1 | chr14q24.3 | -3.4 | |
| 凋亡或肿瘤相关 | NM_000358 | 转化生长因子β1 | TGFBI | chr5q31 | -1.5 |
| X07868 | 胰岛素样生长因子Ⅱ | IGF-Ⅱ | chr11p15.5 | -4.2 | |
| NM_005538 | 抑制素βC | INHBC | chr12q13.1 | -3.3 | |
| 结合蛋白相关 | X07868 | 胰岛素样生长因子Ⅱ | IGF-Ⅱ | chr11p15.5 | -4.2 |
| NM_005125 | 超氧化物歧化酶铜伴侣蛋白 | CCS | chr11q13 | -1.7 | |
| NM_002162 | 细胞间黏附分子3 | ICAM3 | chr19p13.3-p13.2 | -2.7 | |
| 代谢相关 | M33318 | 细胞色素P450家族2, 亚科A, 多肽6 | CYP2A6 | chr19q13.2 | -5.1 |
| NM_004563 | 线粒体磷酸烯醇丙酮酸羧激酶2 | PCK2 | chr14q11.2 | -3.8 | |
| NM_013267 | 肝线粒体谷氨酰胺酶2 | GLS2 | chr12q13 | -4 | |
| 细胞周期相关 | NM_000592 | 补体成分4A/4B | C4A / C4B | chr6p21.3 | -1.5 |
| N33167 | 周期素依赖性蛋白激酶抑制子1C | CDKN1C | chr11p15.5 | -1.6 | |
| NM_000064 | 补体成分3 | C3 | chr19p13.3-p13.2 | -3.1 | |
| 酶活性调节相关 | NM_000125 | 雌激素受体1 | ESR1 | chr6q25.1 | -3.2 |
| NM_021131 | 蛋白质磷酸酶2A, 调节亚单位B' | PPP2R4 | chr9q34 | -1.9 | |
| NM_006944 | 分泌的磷蛋白2, 24 kDa | SPP2 | chr2q37-qter | -4.7 | |
| M13149 | 富含组氨酸糖蛋白 | HRG | chr3q27 | -1.7 | |
| 分子马达相关 | BF593594 | 驱动蛋白2 | KNS2 | chr14q32.3 | -1.5 |
| X98507 | 肌球蛋白IC | MYO1C | chr17p13 | -1.1 | |
| 分子功能退化相关 | NM_004343 | 钙网织蛋白 | CALR | chr19p13.3-p13.2 | -1.2 |
| NM_003097 | 核内小分子核糖核蛋白多肽N | SNRPN | chr15q11.2 | -2.5 | |
| NM_006275 | 富含精氨酸/丝氨酸胱硫醚γ-裂解酶-6 | SFRS6 | chr20q12-q13.1 | -2.1 | |
| 信号传导相关 | X07868 | 胰岛素样生长因子Ⅱ | IGF-Ⅱ | chr11p15.5 | -4.2 |
| NM_000358 | 转化生长因子β | TGFBI | chr5q31 | -1.5 | |
| NM_005538 | 抑制素βC | INHBC | chr12q13.1 | -3.3 | |
| NM_003227 | 转铁蛋白受体2 | TFR2 | chr7q22 | -2.8 | |
| 结构分子相关 | NM_006097 | 肌球蛋白轻肽9调节子 | MYL9 | chr20q11.23 | -1.8 |
| NM_007234 | 动力蛋白激活蛋白3 | DCTN3 | chr9p13 | -1.6 | |
| AY029208 | VI型胶原α2 | COL6A2 | chr21q22.3 | -1.6 | |
| L27624 | 组织因子通道抑制剂2 | TFPI2 | chr7q22 | -4.8 | |
| 转录调控相关 | NM_003195 | 转录延伸因子A2 | TCEA2 | chr20q13.33 | -2.5 |
| NM_005693 | 细胞核受体亚科1, 组H, 成员3 | NR1H3 | chr11p11.2 | -1.6 | |
| NM_004364 | CCAAT/增强子结合蛋白α | CEBPΑ | chr19q13.1 | -2.7 | |
| AI524138 | 转录因子EB | TFEB | chr6p21 | -1.7 | |
| 翻译调控相关 | NM_003321 | 线粒体Tu·译延伸因子 | TUFM | chr16p11.2 | -2.1 |
| NM_001961 | 真核·译延伸因子2 | EEF2 | chr19pter-q12 | -1.1 | |
| AB014564 | KIAA0664蛋白 | KIAA0664 | chr17p13.3 | -2.2 | |
| 物质运输相关 | NM_006623 | 磷酸甘油酸脱氢酶 | PHGDH | chr1p12 | -4.1 |
| NM_000690 | 线粒体醛脱氢酶2家族 | ALDH2 | chr12q24.2 | -2.5 | |
| NM_002899 | 细胞视黄醇结合蛋白 1 | RBP1 | chr3q23 | -3.1 | |
| NM_000898 | B型单胺氧化酶 | MAOB | chrXp11.23 | -2.5 |
| GenBank ID | 基因名称 | 基因代码 | 染色体定位 | Signal Log Ratio | |
| 上调 | NM_007315 | 信号传导及转录激活因子1 | STAT1 | chr2q32.2 | 3.1 |
| NM_004539 | 天冬酰氨酰-tRNA合成酶 | NARS | Chr18q21.2 | 1.6 | |
| NM_007145 | 锌指蛋白146 | ZNF146 | Chr19q13.1 | 3.0 | |
| NM_002398 | 核仁磷蛋白 | NPM1 | Chr5q35 | 2.5 | |
| NM_004501 | 不均一核糖核蛋白U | HNRPU | Chr1q44 | 1.6 | |
| NM_003750 | 真核翻译起始因子3 | EIF3S10 | Chr10q26 | 1.8 | |
| 亚单位10θ | |||||
| AI582238 | 肿瘤排斥抗原 | TRA1 | Chr12q24.2 | 2.1 | |
| NM_002734 | cAMP依赖的蛋白激酶 | PRKARIA | Chr17q23 | 2.0 | |
| 下调 | NM_002997 | 多配体1 | SDC1 | chr2p24.1 | -1.8 |
| NM_021913 | AXL受体酪氨酸激酶 | AXL | chr19q13.1 | -1.4 | |
| NM_000029 | 血管紧张素原 | AGT | chr1q42-q43 | -1.5 | |
| NM_004364 | CCAAT/增强子结合蛋白, α | CEBPΑ | chr19q13.1 | -2.7 | |
| BC005174 | 转录激活因子5 | ATF5 | chr19q13.3 | -2.3 | |
| NM_000125 | 雌激素受体1 | ESR1 | chr6q25.1 | -3.2 | |
| NM_002567 | 前列腺结合蛋白 | PBP | chr12q24.23 | -2 | |
| NM_006006 | 锌指和BTB 域封闭物16 | ZBTB16 | chr11q23.1 | -1.9 | |
| NM_002387 | 结肠直肠癌突变体 | MCC | chr5q21-q22 | -1.6 | |
| X07868 | 胰岛素样生长因子Ⅱ | IGF-Ⅱ | chr11p15.5 | -4.2 |
众所周知, 肿瘤的发生、发展是一个复杂的多因素、多步骤的过程, 是许多肿瘤相关基因表达失常或多种肿瘤抑制基因失活所致, 因此肿瘤组织癌变过程中相关基因及基因组的变化研究显得更为重要. 随着人类基因组计划的完成, 基因芯片作为后基因时代发展起来的一项DNA分析技术, 该技术具有高通量、高效率、样本少等特点, 满足了对多种样本中基因差异表达研究的要求, 已被广泛地用于肿瘤发生、发展的分子生物学研究中. 本实验中我们采用了美国Affymetrix公司的U133 A 2.0基因表达谱芯片, 在肝癌组织中筛选出2756条差异表达的肝癌相关基因, 占筛选基因总数的19.01%, 其中上调表达的基因有1772条, 下调表达的基因有984条. 这些差异基因初步分为16类, 包括凋亡或肿瘤相关基因、细胞周期、分子马达、信号传导、结构分子、转录调控、翻译调控等. 我们的结果表明肝癌的发生发展是一个多基因参与的复杂的变化过程, 涉及到许多相关基因的变化.
在我们筛选出的差异表达基因中有一些是原癌基因和抑癌基因. 原癌基因是生长因子信号系统的组成成分, 在恶性肿瘤中其改变常为点突变、扩增、染色体易位、基因剪切等. 许多原癌基因的表达产物对细胞膜的稳定、受体的活性、信号传递、酶的活性和基因的表达具有显著的调节作用. 抑癌基因是正常的细胞基因, 有诱导组织分化、细胞凋亡和调节细胞生长等功能, 一旦突变和缺失, 可导致癌发生. MYC基因是一种细胞癌基因, 定位于8号染色体上, 可通过染色体易位及DNA扩增而活化. 常见的是通过8号染色体与14号染色体易位, 使得8号染色体上的MYC基因与14号染色体的免疫球蛋白重链融合而被活化, 活化的MYC基因在细胞的生长调控中发挥作用. 朱波 et al[1], Niu et al[2]认为MYC基因可促使HCC细胞增殖, 使HCC癌细胞具有更强的侵袭力, 与HCC的发生发展密切相关. Ras基因定位于人类第12号染色体上, 正常细胞中Ras基因对细胞生长分化具有重要作用, Ras基因的突变被认为在癌变过程中起启动作用并是一个早期事件[3-4]. 最常见的激活方式是点突变, Ras基因编码的鸟苷酸结合蛋白在细胞增殖分化信号从激活的跨膜受体传递到下游蛋白激酶的过程中起作用, 从而产生具有致癌活性的P21蛋白[5], 导致Ras蛋白与GTP的持续结合继而促进细胞生长. 因此Ras基因的突变以及扩增被认为是肿瘤发生的重要原因. 我们的结果中这两种基因及其相关基因群均呈上调表达, 表明与HCC的发生发展密切相关.
肿瘤的发生是细胞凋亡与细胞增殖失衡的结果, 凋亡减弱/增殖增强是肿瘤的重要发生机制. 我们检测到survivin基因呈上调表达. survivin基因位于17q25, 其表达产物survivin是凋亡抑制蛋白家族的新成员, 近年来研究证实他能抑制细胞凋亡, 促进细胞增殖. 也有研究表明, survivin还参与血管的生成. 肿瘤血管形成是肿瘤生长、转移的基础[6-7]. 有研究报道[8-9]survivin在结肠癌组织中呈高度表达, 而癌旁组织不表达, 并与结肠癌的转移有关联性. 因此survivin在结肠癌的发生发展及转移过程中发挥着重要作用. 同样我们推测其在HCC发生发展及转移过程中也发挥着重要作用. 肿瘤从原位增殖到侵袭转移的过程中, 肿瘤细胞必须具备降解细胞外基质和血管基底膜的能力, 必须借助某些酶. 基质金属蛋白酶家族(MMPs)是具有降解细胞外基质及基底膜的一种酶. MMPs家族具有多个成员, 其表达上调、活性增强是促进多种恶性肿瘤侵袭和转移的重要机制. 有研究表明, HCC组织中MMPs蛋白的高表达促进了肿瘤细胞的运动及对基底膜的侵袭能力, 从而导致HCC细胞发生转移, 影响患者预后[10]. 本研究结果中基质金属蛋白酶家族成员基因表达谱在HCC中呈上调表达, 这一结果表明肝癌具有高转移性、高侵袭性的生物学特征.
我们在对差异表达基因进行分类时发现有许多基因具有多种功能, 参与多种细胞活动过程, 涉及到细胞凋亡或肿瘤相关、结合蛋白相关及信号传导等多个方面. 例如胰岛素样生长因子2(IGF-2)就具有这样的功能, 该基因位于人类染色体11 p15.5, 他的表达产物IGF-2是一种多功能调节因子, 对促进机体正常发育具有重要作用, 在胚胎发育、细胞增殖分化、机体物质代谢等方面均有重要功能[11-13]. 近来研究显示IGF-2还是干细胞分化的重要因子[14-15]. IGF-2基因属于印迹基因, 是第1个被证实的内源性印迹基因, 该基因正常情况下在成人大脑脉络丛、软脑膜及肝组织为双等位基因表达, 其他组织中是父系等位基因表达, 而母系等位基因印迹. 在哺乳动物基因组中只有一小部分基因属于印迹基因, 由于各种原因引起的印迹基因异常表达如过高、过低或缺失均可导致肿瘤的发生[13,16-17]. 在本研究中IGF-2基因表达呈下降, 而IGF-2基因在肝细胞中是呈双等位基因表达, 该基因在肝癌中的低表达是否与该基因的印迹或突变有关有待进一步研究.
目前, 基因芯片技术正在被广泛地应用于对各种肿瘤复杂发生发展过程认识的研究及通过基因表达谱的分析来认识肿瘤的发病机制, 为肿瘤的诊断和治疗提供了科学依据. 本研究结果表明, 共有16类基因群2756条基因在HCC组织中异常表达, 表明HCC中很可能存在着互相协调的共同表达的基因群. 因此利用基因表达谱芯片技术对HCC进行基因群水平研究, 筛选在肿瘤相关的多个环节上起重要作用的分子和起关键调节作用的基因, 以揭示HCC在基因水平上的本质, 为HCC的早期诊断和早期治疗提供理论依据.
肝癌是我国较为常见的恶性肿瘤, 居恶性肿瘤死亡率的第2位. 肝癌与其他肿瘤一样, 其发生发展是一个复杂的多因素、多步骤的过程, 是许多肿瘤相关基因表达失常或多种肿瘤抑制基因失活所致, 因此对肿瘤组织癌变过程中相关基因及基因组的变化研究显得尤为重要.
基因芯片技术在肝癌中的研究不过短短几年, 但已涉及到致癌分子的机制, 肝癌的复发、转移和预后相关基因的表达以及对化疗药物敏感性的评价和中药对肝癌的治疗. 目前的研究趋势是应用基因芯片技术对肿瘤表型进行分类, 提高 早期诊断率, 预测预后, 指导临床治疗.
本文利用美国Affymetrix公司寡核苷酸基因芯片对肝癌进行了基因表达谱的分析, 并对差异表达基因进行了分类, 初步分为16类, 包括细胞周期、信号传导、结构分子、转录调控、翻译调控、代谢相关、分子马 达、营养储备、物质运输、酶活性调节、凋亡或肿瘤相关基因等.具有一定的创新性.
本文应用基因芯片技术从2 例肝癌组织中筛选出 2756条差异表达基因, 为认识肝癌发病的分子生物学机制提供了科学依据. 为肝癌的基因治疗和寻找出肝癌早期诊断的分子生物学标志物具有十分重要的意义.
1 基因芯片: 指将许多特定的寡核苷酸片段或基因片段作为探针, 有规律地排列固定于支持物上, 样品D NA/RNA通过PCR扩增、体外转录等技术掺入标记分子, 然后按碱基配对原理进行杂交, 再通过检测系统等对芯片的杂交信号进行量化, 得出所要的信息.2 基因表达谱: 常被称为是某一生理病理现象的 “分子图像”. 这类复杂的“分子图像”, 可用于同时检测成千上万个基因的表达水平, 再经专门的计算机软件解读出来. 研究人员通过比较源于不同病理条件下的“分子图像”的结果, 可以识别出引发癌症等复杂疾病的标志物.
本文采用了基因表达谱芯片技术对肝癌表达异常的相关基因群进行了筛选, 有助于认识肝癌的发病机制, 方法成熟,设计合理, 对临床有一定的意义.
编辑: 程剑侠 电编:何基才
| 1. | 朱 波, 欧 超, 罗 元, 曹 骥, 班 克臣, 焦 杨, 李 瑗, 苏 建家. CyclinD1 c-myc和p53的表达与肝细胞癌生物学行为关系. 中国肿瘤临床. 2006;33:67-70. |
| 2. | Niu ZS, Li BK, Wang M. Expression of p53 and C-myc genes and its clinical relevance in the hepatocellular carcinomatous and pericarcinomatous tissues. World J Gastroenterol. 2002;8:822-826. [PubMed] |
| 4. | 孙 佰秀, 殷 凡, 朱 伟, 高 玉, 孙 淑红, 罗 兵. EBV相关胃癌ras基因突变及法尼基转移酶β-亚单位基因的表达. 世界华人消化杂志. 2006;14:1294-1299. [DOI] |
| 5. | Sekine I, Takami S, Guang SG, Yokose T, Kodama T, Nishiwaki Y, Kinoshita M, Matsumoto H, Ogura T, Nagai K. Role of epidermal growth factor receptor overexpression, K-ras point mutation and c-myc amplification in the carcinogenesis of non-small cell lung cancer. Oncol Rep. 1998;5:351-354. [PubMed] |
| 6. | O'Connor DS, Schechner JS, Adida C, Mesri M, Rothermel AL, Li F, Nath AK, Pober JS, Altieri DC. Control of apoptosis during angiogenesis by survivin expression in endothelial cells. Am J Pathol. 2000;156:393-398. [PubMed] |
| 7. | Blanc-Brude OP, Mesri M, Wall NR, Plescia J, Dohi T, Altieri DC. Therapeutic targeting of the survivin pathway in cancer: initiation of mitochondrial apoptosis and suppression of tumor-associated angiogenesis. Clin Cancer Res. 2003;9:2683-2692. [PubMed] |
| 10. | 何 萍, 张 雁瑞, 徐 雪冬, 车 丽洪, 吴 惠茜, 薛 玲. 影响肝细胞癌生物学行为及预后的相关因子分析. 临床与实验病理学杂志. 2006;22:61-64. |
| 11. | Yu H, Rohan T. Role of the insulin-like growth factor family in cancer development and progression. J Natl Cancer Inst. 2000;92:1472-1489. [PubMed] |
| 12. | Moschos SJ, Mantzoros CS. The role of the IGF system in cancer: from basic to clinical studies and clinical applications. Oncology. 2002;63:317-332. [PubMed] |
| 13. | Jerome L, Shiry L, Leyland-Jones B. Deregulation of the IGF axis in cancer: epidemiological evidence and potential therapeutic interventions. Endocr Relat Cancer. 2003;10:561-578. [PubMed] |
| 14. | Paunescu V, Deak E, Herman D, Siska IR, T anasie G, Bunu C, Anghel S, Tatu CA, Oprea TI, Henschler R. In vitro differentiation of human mesenchymal stem cells to epithelial lineage. J Cell Mol Med. 2007;11:502-508. [PubMed] |
| 15. | Lee KD, Kuo TK, Whang-Peng J, Chung YF, Lin CT, Chou SH, Chen JR, Chen YP, Lee OK. In vitro hepatic differentiation of human mesenchymal stem cells. Hepatology. 2004;40:1275-1284. [PubMed] |
| 16. | Jirtle RL. Genomic imprinting and cancer. Exp Cell Res. 1999;248:18-24. [PubMed] |