修回日期: 2007-02-01
接受日期: 2007-02-13
在线出版日期: 2007-05-28
目的: 通过一种能够在细胞内表达短发卡RNA的逆转录病毒载体来研究RNA干扰对乙型肝炎病毒复制的影响.
方法: 首先针对HBV基因组Pol基因RT区寻找RNAi的靶位, 并设计相对应的寡核苷酸链. 然后再将一对碱基配对寡核苷酸链退火后连接入载体形成重组的质粒, 并进行鉴定. 在Huh-7细胞中, 将通过鉴定的干扰质粒与HBV复制型质粒pHBV3.8共转染, 分别应用ELISA方法来检测HBV相关的抗原, 应用Northern印迹检测HBV RNA, 以及应用实时荧光定量PCR和Southern印迹检测HBV核心颗粒DNA.
结果: 研究通过计算机方法协助寻找了3条RNAi靶位, 并且构建了相应的基于逆转录病毒载体的RNA干扰质粒154i、312i和734i. 结果发现312i对pHBV3.8表达有明显的抑制, HBsAg和HBeAg分别为阴性对照组的39%和41%, 差异均有显著性(P = 0.001, P = 0.000). 定量荧光PCR结果显示312i组核心颗粒DNA水平显著低于阴性对照组(21.3%±1.1% vs 100.0%±10.6%, P = 0.0046). Southern blot和Northern blot结果均显示, 312i组病毒复制及mRNA转录水平最低(10.5%, 12.0%).
结论: 在HBV基因组RT区找到了一个可用于RNAi的靶位, 并且构建了相对应的干扰质粒, 此质粒可成功地抑制HBV复制型质粒在细胞中的复制和表达.
引文著录: 王新宇, 张继明, 尹有宽, 谢怡, 黄玉仙, 邬祥惠, 翁心华. RNA干扰对乙型肝炎病毒复制的体外影响. 世界华人消化杂志 2007; 15(15): 1688-1694
Revised: February 1, 2007
Accepted: February 13, 2007
Published online: May 28, 2007
AIM: To study the RNA interference on hepatitis B virus (HBV) replication by a reverse transcription virus vector which can express short hairpin RNA inside cells.
METHODS: pSIREN vectors with inserted oligonucleotides targeting on reverse transcriptase (RT) regions of HBV genome were constructed. These plasmids were co-transfected with pHBV3.8 into Huh-7 cells. Viral antigens were measured by enzyme-linked immunosorbent assay (ELISA). HBV core particle DNA was measured and quantified by real-time fluorescence quantitative polymerase chain reaction (RFQ-PCR) and Southern blot. Viral RNA was analyzed by Northern blot.
RESULTS: Three RNA interfering targets were identified, and three corresponding retrovirus vectors, named 154i, 312i and 734i, were obtained. It was found that 312i markedly inhibited the expression of pHBV3.8, and the levels of HBsAg and HBeAg were 39% and 41% of those in the negative control group (P = 0.001, P = 0.000). RFQ-PCR showed that the level of HBV core particle DNA was significantly lower in 312i group than that in the negative control group (21.3% ± 1.1% vs 100.0% ± 10.6%, P = 0.0046). Southern and Northern blot demonstrated a lowest replication and transcription level of HBV in 312i group (10.5%, 12.0%).
CONCLUSION: A new RNAi system is identified in the RT regions of HBV genome, and the corresponding retrovirus vectors, which can remarkably inhibit the replication and expression of HBV, are also constructed.
- Citation: Wang XY, Zhang JM, Yin YK, Xie Y, Huang YX, Wu XH, Weng XH. Inhibition of hepatitis B virus expression and replication by RNA interference. Shijie Huaren Xiaohua Zazhi 2007; 15(15): 1688-1694
- URL: https://www.wjgnet.com/1009-3079/full/v15/i15/1688.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i15.1688
乙型肝炎病毒(HBV)感染是一个严重的世界公共卫生难题[1]. 目前, 公认有效的抗病毒药物中a-干扰素由于其副作用较多并且治疗的应答率较低从而限制了其在临床上的应用[2]. 核苷类似物虽然能有效的使患者血液中的病毒载量降到低于检测水平的程度, 但是持久的病毒学应答却也不能令人满意[3]. 因此, 人们需要新的治疗方法来控制HBV感染, 以RNA干扰(RNAi)为手段的抗病毒基因治疗可能是一种具有潜力的治疗策略. RNAi是指由双链RNA(dsRNA)启动的序列特异性的转录后基因沉默现象(PTGS)[4-9]. 最近, 科学家们已经在数种不同的哺乳动物细胞中成功的利用RNAi技术抑制了包括HCV、HBV和HDV在内的数种不同病毒的复制[10-15]. 与其他基因治疗的方法类似, 给药途径是RNAi目前遇到的最大障碍[16-17]. 通过化学合成的siRNA只能显示出短暂的反应, 而基于载体体内表达shRNA的方法虽然能够获得一个较为持续的效果, 但在体内表达的效率相对较低. 逆转录病毒载体可以避免以上的两个问题. 我们尝试利用逆转录病毒载体来研究siRNA对HBV复制的影响, 而目前国际上尚无类似的文献报道.
NheⅠ内切酶(NEB公司), RNAi-Ready pSIREN-RetroQ Vector(CloneTech公司), T4 DNA 连接酶(NEB公司), 质粒小量抽提试剂盒(Sigma公司), Premix Taq酶(TaKaRa公司), 感受态细菌(CaCl2方法自制), HBV复制型质粒pHBV3.8(中国科学院生化所汪垣教授惠赠), 氨苄青霉素(上海生工生物技术公司), LB培养液(Sigma公司), 长链寡核苷酸DNA合成(上海博亚生物技术有限公司), PCR引物合成(上海博亚生物技术有限公司), Huh-7细胞株(本室保存), 培养瓶(Nunc公司), 48孔板-6孔板(Corning公司), 100 mm培养皿(Corning公司), DMEM培养液(Gibco公司), 胎牛血清(JRH公司), CalPhos转染试剂盒(BD公司), Fugene 6脂质体转染试剂盒(Roche公司), 地高辛标记, 检测系统(Roche公司), TRIzol(Sigma公司), 质粒大量抽提试剂盒(Qiagen公司), pGL2萤火虫荧光素酶表达质粒和pRL海肾荧光素酶表达质粒(Promega公司), 荧光素酶检测试剂盒(Promega公司), HBV表面抗原和E抗原ELISA检测试剂盒(上海科华生物), 尼龙膜(Roche公司), FastStart DNA Master SYBR Green I mixture (Roche公司), 热循环仪(Eppendorf公司), 紫外成像仪(上海复日生物技术有限公司), 摇床(上海精密仪器有限公司), 振荡器(上海精密仪器有限公司), 离心机(Eppendorf公司), 微量移液枪(Eppendorf公司), 电泳槽仪(上海复日生物技术有限公司), 序列测定(上海博亚生物技术有限公司), 恒温培养箱(上海精密仪器有限公司), 荧光发光计(TurnerBiosystems公司), 显微镜(Nikon公司), 酶标仪(Biorad公司).
1.2.1 寡核苷酸的设计: 参考现有文献[18-19], 在HBV复制型质粒pHBV3.8序列中HBV基因组的Pol基因RT区中搜索潜在的干扰靶位. 使用RNA structure软件(http://rna.urmc.rochester.edu/rnastructure.html)预测连同AA二聚体在内的21个核苷酸序列的二级结构, 排除那些可能形成复杂二级结构的候选序列. 通过DNA数据库相似性比较分析软件BLAST比较候选序列与人类基因库基因的相似性, 以确认候选序列的特异性, 即与人类基因没有高度同源性. 通过上述条件筛选的序列成为本研究候选的干扰靶位.
1.2.2 RNA干扰质粒的构建: 我们选用RNAi-Ready pSIREN-RetroQ载体做为RNAi的研究工具, 该载体带有y+信号, 可以在包装细胞(HEK293)内包装出高滴度的复制缺陷的逆转录病毒, 进而通过感染宿主, 在宿主体内稳定表达shRNA. 由上海博亚生物技术有限公司合成长链寡核苷酸序列, 使用TE缓冲液溶解PAGE纯化的寡核苷酸至浓度100 mmol/L. 1∶1比例混合上下游寡核苷酸溶液, 使寡核苷酸终浓度为50 mmol/L. 在热循环仪上设置95℃ 30 s以去除二级结构, 随后72℃ 2 min, 37℃ 2 min, 最后25℃ 2 min. 使用TE缓冲液将退火的寡核苷酸稀释到0.5 mmol/L, 将每对退火的寡核苷酸配制一个连接反应. 同时也将试剂盒提供预先已退火的特异性针对萤火虫荧光素酶的寡核苷酸和阴性对照寡核苷酸与载体进行连接. 将连接后的质粒转化至DH5a感受态细菌中, 挑取阳性克隆, 培养过夜后, 使用Sigma公司的质粒小量抽提试剂盒进行质粒抽提. 用Qiagen Plasmid Maxi kit质粒纯化试剂盒抽提细胞转染用的高纯度质粒DNA. 实验使用的阴性对照Neg均为针对荧光素酶pGL2的特异性干扰质粒, 经过BLAST搜索与HBV无同源性.
1.2.3 RNA干扰质粒的鉴定: 测序鉴定是为了确认插入的序列的准确性. 使用的测序引物为U6FSP(5'>GGGCAGGAAGAGGGCCTAT<3'). 每组挑选两个酶切阳性的克隆送上海博亚生物技术有限公司进行DNA测序, 测序采用的是ABI 377测序仪及配套的BigDye Terminator试剂盒.
1.2.4 细胞及细胞培养: Huh-7细胞来源于人肝癌细胞系, HBV复制型质粒pHBV3.8能在Huh-7细胞中转录出HBV前基因组RNA及相关病毒mRNA, 然后翻译出病毒相关蛋白, 如核心蛋白、表面抗原(HBsAg)、e抗原(HBeAg)等, 这些病毒抗原在细胞上清中易于检出. 细胞均放于含有100 mL/L胎牛血清、10 000 U/L青霉素、0.1 g/L链霉素的DMEM培养液中. 细胞均培养于50 mL/L CO2, 37℃的细胞培养箱中培养, 每48 h通过胰酶/EDTA进行消化后传代培养.
1.2.5 真核细胞体外转染及检测: 转染前消化细胞后, 按照1×105细胞数/cm2铺板, 按照质粒: 脂质体(w/v)为3∶1配制转染液: 先在进口离心管中加入无血清培养液, 再将脂质体直接加入培养液中, 再加入转染的质粒, 轻轻混匀后, 室温静置20 min, 用移液枪滴加入培养板后, 置入37℃, 50 mL/L CO2的培养箱中孵育至分析. 转染效率的监测及校正采用Promega双荧光检测系统, 操作规范按照相关说明. HBsAg和HBeAg的检测采用上海科华生物技术有限公司试剂盒.
1.2.6 地高辛探针的制备: 使用地高辛标记的dNTP作为PCR的反应原料, 使用pHBV3.8作为模板, 进行常规PCR, 引物分别为: 5'-CCTCTTCATCCTGCTGCT-3', 5'-AACTGAAAGCCAAACAGTG-3'. 通过扩增可以得到带有地高辛标记的334 bp的HBV S基因探针. 合成得到的杂交探针用于Southern和Northern印迹实验.
1.2.7 Southern印迹: 细胞内HBV核心颗粒DNA抽提按照Lin et al[20]的方法进行操作. 提取的核心颗粒DNA经10 g/L琼脂糖凝胶电泳分离(50 V, 2 h). 胶经30 min碱变性(0.5 mol/L NaOH, 0.15 mol/L NaCl), 15 min中和(1.5 mol/L NaCl, 0.5 mol/L Tris-HCl pH7.2)两次后, 按常规方法经36 h虹吸转移至阳离子尼龙膜, 转移后的尼龙膜以2×SSC液漂洗5 min, 自然晾干后置120℃烘烤30 min. 42℃预杂交30 min后. 将地高辛标记的DNA探针通过在沸水中水浴5 min后迅速冰浴的方法变性后, 加入预杂交液充分混匀, 42℃杂交过夜. 次日先用高盐洗液(2×SSC, 1 g/L SDS)室温洗涤5 min 2次, 再用低盐洗液(0.5×SSC, 10 g/L SDS) 65℃洗涤15 min 2次. 在洗涤缓冲液中浸泡5 min后, 用100 mL阻断液孵育30 min, 再在20 mL抗体溶液中孵育30 min, 洗涤缓冲液100 mL 15 min洗涤2次后, 置入20 mL检测缓冲液平衡5 min. 使用CSPD(3-4-甲氧基螺甾[1,2-二氧六环-3 2'(5-氯)三环[3.3.1.13,7]癸基]-4-苯基磷酸钠)化学发光液孵育尼龙膜30 min后, 将膜转移至干净的袋中并放在底片盒中. 取医用底片曝光2 h后显影, 定影.
1.2.8 Real-Time PCR分析: 定量PCR检测HBV核心颗粒中DNA含量所用样品按上述的转染细胞胞质内HBV病毒核心颗粒中DNA抽提的方法制备. 定量PCR样品反应体系采用FastStart DNA Master SYBR Green I mixture(罗氏公司产品).
1.2.9 RNA抽提与Northern印迹: 细胞转染72 h后, 用TRIzol抽提转染细胞总RNA. 抽提方法为将细胞培养皿中的上清吸去后, 加入TRIzol(1 mL/直径35 mm培养皿), 在室温放置5 min后, 将细胞裂解液移入离心管中, 加1/5体积的氯仿, 振荡15 s混匀后, 静置3 min, 12 000 g 4℃离心15 min后, 吸取上层水相, 加入0.5 mL异丙醇, 室温静置10 min, 12 000 g 4℃离心10 min, 弃上清, 将沉淀用750 mL/L乙醇洗过后, 溶于DEPC处理过的H2O中. 取溶解得到的细胞总RNA与2×RNA上样缓冲液混合后加入经甲醛变性的琼脂糖凝胶的加样孔中, 80 V电泳2 h后, 紫外成像仪摄影, 再将凝胶按常规方法经36 h毛细转移法转移至阳离子尼龙膜, 转移后的膜经6×SSC漂洗后自然晾干, 置烤箱内120℃烘烤30 min. 以后杂交, 洗涤, 发光, 显影的方法与Southern印迹基本相同.
统计学处理 两样本均数差别采用t检验, 若未达到方差齐性, 则采用t'检验, 以P值<0.05定为具有统计学显著性差异, 统计软件为Stata.
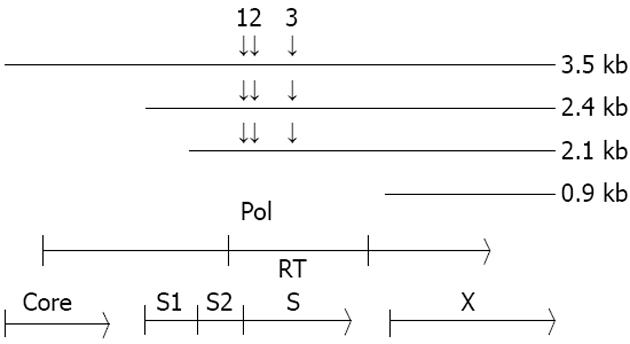
根据既定的研究方法, 我们在HBV复制型质粒pHBV3.8序列的Pol基因RT区选择了3条候选序列(图1). 分别根据序列相对的位置命名为154i, 312i和734i. 这3条寡核苷酸的序列分别为154i: 5'CATGGAGAACATCACATCA3', 312i: 5'TTCGCAGTCCCCAACCTCC3', 734i: 5'GTTATATGGATGATGTGGT3'. 在计算了他们的GC含量后认为他们基本符合成为RNA干扰靶位的条件. 然后我们通过RNA Structrue软件对这3条靶位序列相对应的RNA进行了RNA二级结构的计算机模拟. 结果发现154i和734i均出现在对应RNA序列形成的二级环状结构的顶部, 而312i对应的RNA序列则不能自行折叠形成二级结构. 再将这3条序列通过BLAST程序(http://www.ncbi.nlm.nih.gov/BLAST)与人类基因组进行了同源性分析, 结果均没有发现与该3条序列高度同源的序列. 其中154i和734i与人类基因组最高发生16个碱基的同源, 而312i与人类基因组最高发生17个碱基的同源, 且同源性较高的基因多为癌基因. 该3条序列均符合进一步研究的要求.
实验结果发现312i对pHBV3.8表达有明显的抑制, HBsAg和HBeAg分别为Neg的39%和41%. 而734i和154i也有不同程度抑制, 但抑制的效率较312i弱, 734i为Neg的56%和62%, 154i为Neg的80%和83%. 进行统计检验发现, 无论是HBsAg还是HBeAg, Neg组和312i组的差异均有显著性(P = 0.001和P = 0.000). 在312i组中可以发现对HBsAg的抑制效率略高于HBeAg(表1).
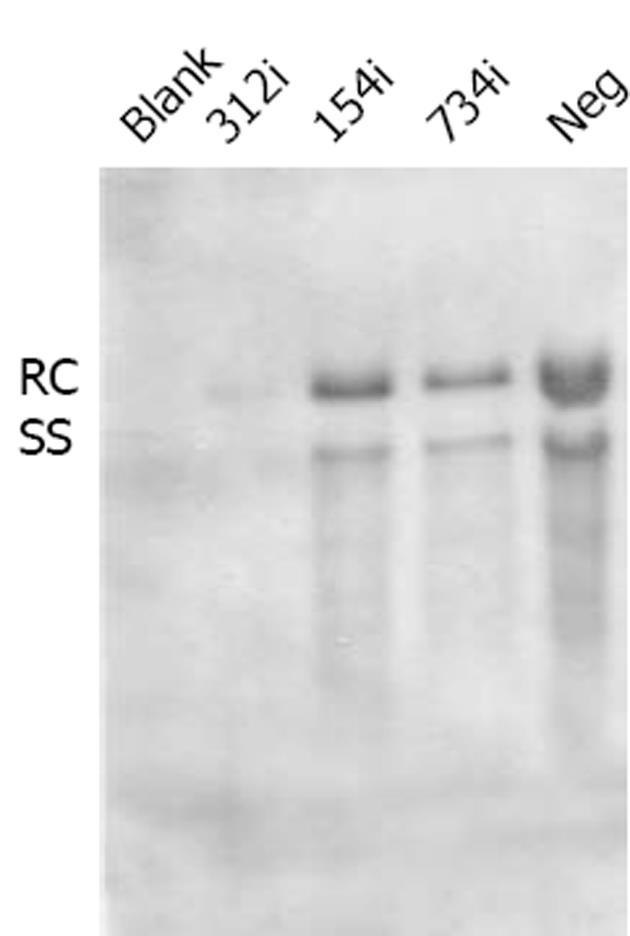
本实验结果(表1), 可见核心颗粒DNA测定结果中312i组对pHBV3.8质粒表达的抑制最为明显. 而734i和154i与Neg相比差异不明显. 进行统计检验发现Neg组和312i组的差异有显著性(P = 0.0046).
实验结果发现Neg组泳道条带强度要明显强于其他泳道条带, 而312i组条带强度则很弱, 而154i与734i组条带强度类似(图2). 我们对Southern印迹的图像进行定量分析. 分析结果我们看到, 312i, 154i和734i相对密度分别为Neg组的10.5%, 48.3%和34.7%. 312i对于HBV3.8复制型质粒的复制影响最明显(表2).
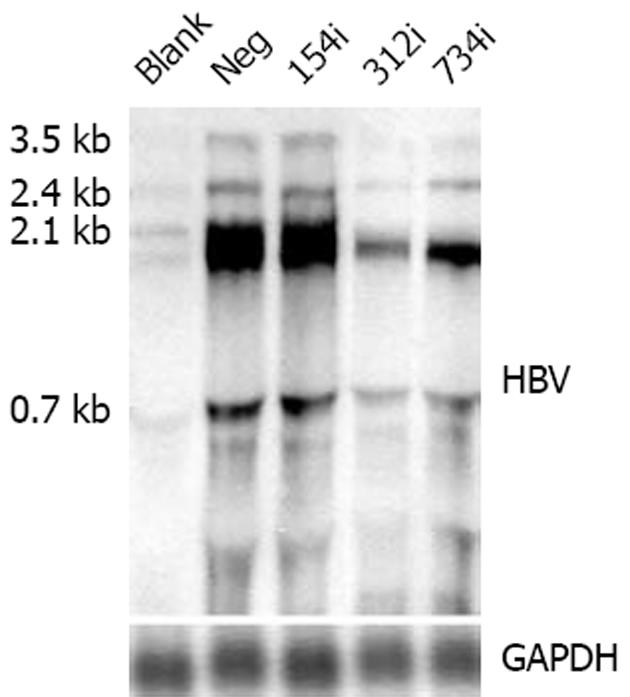
实验结果发现Neg组和154i组泳道条带强度要明显强于其他泳道条带, 而312i组条带强度则很弱, 而734i组条带强度略强于312i组(图3). 我们使用Gel-Pro Analyzer软件对Nouthern印迹的图像进行定量分析. 分析结果我们看到, 154i, 312i, 和734i相对密度分别为Neg组的95.3%, 12.0%和42.9%. 312i对于HBV3.8复制型质粒的转录mRNA影响最明显(表2).
成功地研究开发出一个具有潜力的抗HBV治疗策略依旧是一项难题. 目前抗HBV药物的研发主要都是针对抑制HBV的多聚酶. 然而, 由于HBV的逆转录酶缺乏校读的机制[21], HBV基因组经历一系列突变后在HBV复制过程中发生了耐药. 即使是HBV基因组的一个点突变也可能导致耐药的发生[22]. RNAi可以特异性地沉默基因表达, 他提供了一种与目前药物不同的抗HBV机制, 可能可以克服或者弥补当前药物的不足.
从整个基因治疗的现状来看, 最有效的给药途径无疑是病毒载体. Cavazzana Calvo et al[23]报道, 他们已经通过逆转录病毒载体导入患者原有体内缺陷的基因的方法, 有效地治疗了18例患有严重联合免疫缺陷病(SCID)的儿童, 这是近年来在基因治疗研究中最成功的临床研究案例. 逆转录病毒能够感染正常的人体体细胞, 此后逆转录病毒RNA在体内便可以被正常逆转录为DNA, 随后病毒DNA可以正常整合到宿主染色体上并转录为RNA. 通过这一方法, 理论上宿主可以持久稳定地表达逆转录病毒载体上的基因序列. 在自然感染HBV的病例中, HBV以共价闭合环状DNA(ccc DNA)作为转录的模板. 因此, 要治疗慢性HBV感染, siRNA需要或者持续地在感染的肝细胞中保持工作浓度以持续的降解HBV RNA, 或者是使ccc DNA停止转录. 然而, 在哺乳动物细胞中用普通方法转染的siRNA介导的基因沉默效应都只是瞬间的. 并且, 缺少在蠕虫和植物中RNA沉默的放大效应[24]. 最近发展起来的慢病毒、逆转录病毒和腺病毒相关病毒的基因于shRNA表达的载体提供了1种在哺乳动物细胞中长期表达的有效途径[25-27]. 本研究成功利用逆转录病毒载体构建了能够表达针对HBV基因组的干扰质粒, 为进一步的研究奠定了基础.
不同的研究小组已经通过一定的计算机软件来预测了HBV前基因组RNA的二级结构[28-30]. 文献表明, RNAi靶位不能具有复杂的二级结构, 复杂的二级结构会影响siRNA与靶RNA的结合, 大大降低RNAi的效果. 在本研究中, 我们利用RNA Structure软件来预测HBV前基因组RNA的二级结构. 研究发现不能够形成二级结构的312i靶位对HBV复制和表达的抑制效果最佳, 而针对环型RNA结构的734i和154i都没有312i的抑制效率高. 研究证实RNA的二级结构可以影响靶位RNA的沉默效率. 由于所有的HBV转录子在他们的C末端都有重叠, 因此设计1条shRNA同时针对多条关键的转录子可能可以最大化抑制的效能. 正是出于这个推论, 我们的3个靶位都能够针对除了X mRNA之外的所有HBV转录子. 因此他对于HBsAg和HBeAg都有基本相同的抑制效应. 因为他们所针对的靶位同时针对两者的mRNA. 在先前的报道中, Klein et al[12]仅针对S靶位的siRNA发现抑制HBeAg的效率却优于HBsAg. Song et al[31]也发现针对人类免疫缺陷病毒的同一个siRNA在不同细胞抑制病毒的效率并不同. 我们发现, 干扰质粒抑制HBsAg的效率要略高于HBeAg. 目前, 并不清楚为何在不同的细胞中针对同一siRNA的抑制效率并不一致. 有一种可能的解释是在不同的细胞中, 靶位的RNA形成了不同的RNA二级结构, 或者是RNA结合了不同的蛋白. 这种差异造成了同一siRNA的不同效率.
总之, 目前看来RNAi作为1种抗HBV的工具具有以下一些重要的优点: 首先, RNAi特异性地针对病毒转录, 进而影响其复制和翻译过程, RNAi并不会激活非特异性的细胞反应, 因此可以使意料外的副作用最小化. 其次, 病毒基因组上众多潜在的靶位可以使研究者尽量选择保守区域以避免产生逃逸突变. 更值得重视的是由于核苷类似物只是在HBV复制过程中起作用, 而siRNA甚至可以在病毒复制停止的细胞中降低转录子和蛋白的水平, 这使其可能成为新1类的抗病毒药物. 本研究结果表明RNAi可能是治疗乙型肝炎病毒感染的一种潜在的有效治疗工具, 虽然有些技术方面的障碍尚待解决. 如果我们能够解决如何优化选择靶序列、发现更有效的给药途径、设计出更安全的载体这些问题的话, siRNA将很可能在不久后的将来成为一种新兴的抗病毒, 特别是治疗HBV感染的药物.
感谢复旦大学上海医学院教育部卫生部医学分子病毒学实验室袁正宏教授及其研究小组对本研究的指导和帮助.
基因治疗是把正常的基因整合入细胞, 以校正和置换致病基因的一种治疗方法. 从广义上来讲, 将某种遗传物质转移到患者细胞内, 使其在体内发挥作用, 以达到治疗疾病的方法, 也谓之基因治疗. RNAi便属于后者, 他是指由双链RNA(dsRNA)启动的序列特异性的转录后基因沉默现象(PTGS).
科学家们已经在数种不同的哺乳动物细胞中成功地利用RNAi技术抑制了病毒复制. Bitko et al利用siRNA沉默了由呼吸道合胞病毒(RSV)产生的mRNA, RSV是一种引起呼吸道疾病的负链病毒. 最近, 又有多个研究将RNAi的目标瞄准了HIV. 在体外模型中, siRNA或者直接针对HIV病毒, 或者针对编码HIV-1重要的辅助受体CXCR4/CCR5的RNA. 研究者在对于包括HCV、HBV和HDV进行的RNAi研究中也获得了成功. 他们以病毒复制子细胞或整合细胞以及动物模型为对象, 观察到RNAi能有效干扰这些病毒的复制和表达. RNAi已经成功抑制的其他病毒还包括登革热病毒, 兽棚病毒(FHV), 恒河猴病毒(RRV), É林脑炎病毒(SFV), 流感病毒和脊髓灰质炎病毒.
Konishi et al通过化学合成siRNA的方法在一种能够持续产生HBV感染颗粒的肝癌细胞系中成功地抑制了病毒复制. Shlomai和Shaul利用一种以质粒为载体的RNAi来抑制HBV. 这种方法是通过位于该质粒H1RNA启动子后的短发卡状RNA(shRNA)的表达, 来起到与siRNA相同的作用. 这些shRNA同样也抑制了在持续表达病毒颗粒的细胞中HBV病毒的复制. 这项研究的靶位选择了核心基因和X基因. Klein et al的研究又进了一步, 他们成功地在小鼠模型中抑制了HBV. MaCaffrey et al以质粒为载体, 利用RNAi方法成功在小鼠体内抑制了HBV.
与其他基因治疗的方法类似, 有效的给药途径是RNAi目前遇到的最大障碍. 通过化学合成的siRNA只能显示出短暂的反应, 而基于载体体内表达shRNA的方法虽然能够获得一个较为持续的效果, 但在体内表达的效率相对较低. 逆转录病毒载体可以避免以上的两个问题. 本研究尝试利用逆转录病毒载体来研究siRNA对HBV复制的影响, 而目前国际上尚无类似的文献报道.
本研究为进一步利用该干扰质粒在包装细胞(HEK293)中包装产生逆转录病毒, 并且尝试利用这种复制缺陷的逆转录病毒在细胞和实验动物体内稳定表达shRNA以持久地抑制HBV的表达与复制奠定了基础. 本研究结果表明RNAi可能是治疗乙型肝炎病毒感染的一种潜在的有效治疗工具, 虽然有些技术方面的障碍尚待解决. 如果我们能够解决如何优化选择靶序列、发现更有效的给药途径、设计出更安全的载体这些问题的话, siRNA将很可能在不久后的未来成为一种新兴的抗病毒, 特别是治疗HBV感染的药物.
siRNA: 小干扰RNA, 是dsRNA被一种称为Dicer的RNaseⅢ家族的酶所分解后产生的21-25 bp大小的短链RNA. siRNA在与酶复合体RISC(RNA介导的沉默复合体)结合展开后, 又在RISC的协助下, 根据Watson-Crick碱基配对的规则来选择目标RNA, 与siRNA同源的靶RNA将被降解.
本文作者利用逆转录病毒载体找到了一个有效的抑制HBV复制的SiRNA. 文章技术路线明确, 书写规范, 有较好的科学价值.
电编: 张敏 编辑:王晓瑜
| 1. | Maddrey WC. Hepatitis B: an important public health issue. J Med Virol. 2000;61:362-366. [PubMed] [DOI] |
| 2. | Lee WM. Hepatitis B virus infection. N Engl J Med. 1997;337:1733-1745. [PubMed] [DOI] |
| 3. | Liaw YF. Chronic hepatitis B guidelines: east versus west. Hepatology. 2002;35:979-981. [PubMed] [DOI] |
| 4. | Elbashir SM, Harborth J, Lendeckel W, Yalcin A, Weber K, Tuschl T. Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells. Nature. 2001;411:494-498. [PubMed] [DOI] |
| 5. | Hammond SM, Caudy AA, Hannon GJ. Post-transcriptional gene silencing by double-stranded RNA. Nat Rev Genet. 2001;2:110-119. [PubMed] [DOI] |
| 6. | Hutvagner G, Zamore PD. RNAi: nature abhors a double-strand. Curr Opin Genet Dev. 2002;12:225-232. [PubMed] [DOI] |
| 7. | Nykanen A, Haley B, Zamore PD. ATP requirements and small interfering RNA structure in the RNA interference pathway. Cell. 2001;107:309-321. [PubMed] [DOI] |
| 8. | Schwarz DS, Hutvagner G, Haley B, Zamore PD. Evidence that siRNAs function as guides, not primers, in the Drosophila and human RNAi pathways. Mol Cell. 2002;10:537-548. [PubMed] [DOI] |
| 10. | Bitko V, Barik S. Phenotypic silencing of cytoplasmic genes using sequence-specific double-stranded short interfering RNA and its application in the reverse genetics of wild type negative-strand RNA viruses. BMC Microbiol. 2001;1:34. [PubMed] [DOI] |
| 11. | Gitlin L, Karelsky S, Andino R. Short interfering RNA confers intracellular antiviral immunity in human cells. Nature. 2002;418:430-434. [PubMed] [DOI] |
| 12. | Klein C, Bock CT, Wedemeyer H, Wustefeld T, Locarnini S, Dienes HP, Kubicka S, Manns MP, Trautwein C. Inhibition of hepatitis B virus replication in vivo by nucleoside analogues and siRNA. Gastroenterology. 2003;125:9-18. [PubMed] [DOI] |
| 13. | Konishi M, Wu CH, Wu GY. Inhibition of HBV replication by siRNA in a stable HBV-producing cell line. Hepatology. 2003;38:842-850. [PubMed] [DOI] |
| 14. | McCaffrey AP, Nakai H, Pandey K, Huang Z, Salazar FH, Xu H, Wieland SF, Marion PL, Kay MA. Inhibition of hepatitis B virus in mice by RNA interference. Nat Biotechnol. 2003;21:639-644. [PubMed] [DOI] |
| 15. | Shlomai A, Shaul Y. Inhibition of hepatitis B virus expression and replication by RNA interference. Hepatology. 2003;37:764-770. [PubMed] [DOI] |
| 16. | Locarnini S. Therapies for hepatitis B: where to from here? Gastroenterology. 2005;128:789-792. [PubMed] [DOI] |
| 17. | Radhakrishnan SK, Layden TJ, Gartel AL. RNA interference as a new strategy against viral hepatitis. Virology. 2004;323:173-181. [PubMed] [DOI] |
| 18. | Chen Y, Du D, Wu J, Chan CP, Tan Y, Kung HF, He ML. Inhibition of hepatitis B virus replication by stably expressed shRNA. Biochem Biophys Res Commun. 2003;311:398-404. [PubMed] [DOI] |
| 19. | Zhang XN, Xiong W, Wang JD, Hu YW, Xiang L, Yuan ZH. siRNA-mediated inhibition of HBV replication and expression. World J Gastroenterol. 2004;10:2967-2971. [PubMed] [DOI] |
| 20. | Lin X, Yuan ZH, Wu L, Ding JP, Wen YM. A single amino acid in the reverse transcriptase domain of hepatitis B virus affects virus replication efficiency. J Virol. 2001;75:11827-11833. [PubMed] [DOI] |
| 21. | Seeger C, Mason WS. Hepatitis B virus biology. Microbiol Mol Biol Rev. 2000;64:51-68. [PubMed] [DOI] |
| 22. | Fu L, Cheng YC. Role of additional mutations outside the YMDD motif of hepatitis B virus polymerase in L(-)SddC (3TC) resistance. Biochem Pharmacol. 1998;55:1567-1572. [PubMed] [DOI] |
| 23. | Cavazzana-Calvo M, Fischer A. Efficacy of gene therapy for SCID is being confirmed. Lancet. 2004;364:2155-2156. [PubMed] [DOI] |
| 25. | Chen M, Granger AJ, Vanbrocklin MW, Payne WS, Hunt H, Zhang H, Dodgson JB, Holmen SL. Inhibition of avian leukosis virus replication by vector-based RNA interference. Virology. 2007;. [PubMed] |
| 26. | Harper SQ, Staber PD, Beck CR, Fineberg SK, Stein C, Ochoa D, Davidson BL. Optimization of feline immunodeficiency virus vectors for RNA interference. J Virol. 2006;80:9371-9380. [PubMed] [DOI] |
| 27. | Moore MD, McGarvey MJ, Russell RA, Cullen BR, McClure MO. Stable inhibition of hepatitis B virus proteins by small interfering RNA expressed from viral vectors. J Gene Med. 2005;7:918-925. [PubMed] [DOI] |
| 28. | Kidd AH, Kidd-Ljunggren K. A revised secondary structure model for the 3'-end of hepatitis B virus pregenomic RNA. Nucleic Acids Res. 1996;24:3295-3301. [PubMed] [DOI] |
| 29. | Kidd-Ljunggren K, Zuker M, Hofacker IL, Kidd AH. The hepatitis B virus pregenome: prediction of RNA structure and implications for the emergence of deletions. Intervirology. 2000;43:154-164. [PubMed] [DOI] |
| 30. | Patzel V, Sczakiel G. The hepatitis B virus posttranscriptional regulatory element contains a highly stable RNA secondary structure. Biochem Biophys Res Commun. 1997;231:864-867. [PubMed] [DOI] |