修回日期: 2006-01-05
接受日期: 2006-01-25
在线出版日期: 2006-03-08
目的: 利用基因芯片技术筛选胃腺癌组织和癌旁正常组织间的差异表达基因.
方法: 分别抽取胃腺癌组织和癌旁正常组织的总RNA. 采用逆转录的方法, 制成cDNA链, 并以两种荧光Cy5和Cy3标记后作为探针, 与含有14 784条人类14KcDNA基因表达谱芯片进行杂交. 以Agilent荧光扫描仪扫描芯片上两种荧光信号, 并用计算机处理和分析.
结果: 在14 784条基因中, 4例胃腺癌组织和癌旁正常组织共同差异表达基因29条, 其中上调基因10条, 下调基因19条, 上调的基因中有2条功能信息不明.
结论: 胃腺癌发生过程中有多基因的参与, 胃腺癌与癌旁正常组织共同差异表达的29条基因可能与胃癌的发生、发展有关.
引文著录: 李曼, 赵作伟, 辛彦. 应用基因表达谱芯片筛选胃腺癌相关基因. 世界华人消化杂志 2006; 14(7): 666-670
Revised: January 5, 2006
Accepted: January 25, 2006
Published online: March 8, 2006
AIM: To investigate the genes differently expressed between gastric adenocarcinoma and normal mucosal tissues by cDNA microarray technique.
METHODS: The total RNA was extracted from gastric adenocarcinoma and normal mucosal tissues and then purified. The cDNA was obtained by reverse transcription polymerase chain reaction (RT-PCR), and then labeled with Cy5 and Cy3 fluorescence as probes, which were hybridized with gene chip containing 14 784 human 14KcDNA. Subsequently, the signal images were scanned by Agilent fluorescence scanner and analyzed by Pix QuantArray software.
RESULTS: Of the 14 784 target genes, 29 genes were screened out for differences in gene expression level between gastric adenocarcinoma and normal gastric mucosa. Ten of the 29 genes were up-regulated, of which 2 were with unknown function, and 19 were down-regulated.
CONCLUSION: Multiple genes take part in the carcinogenesis of gastric adenocarcinoma. The 29 gene differentially expressed may be involved in the occurrence and development of gastric cancer.
- Citation: Li M, Zhao ZW, Xin Y. Screening of genes differentially expressed in gastric adenocarcinoma by gene expression profiling technique. Shijie Huaren Xiaohua Zazhi 2006; 14(7): 666-670
- URL: https://www.wjgnet.com/1009-3079/full/v14/i7/666.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i7.666
胃癌是严重威胁人类健康的主要恶性肿瘤, 其中胃腺癌占绝大多数, 约为40%-60%, 成为胃癌最常见的组织学类型. 目前对胃癌病因、发病机制的研究已经进入了分子水平, 认为胃癌的发生和发展是多种肿瘤相关基因表达失常. 基因表达谱芯片为研究胃癌相关基因表达提供了理想的技术支持, 通过对来源不同个体、不同组织、不同刺激条件下的组织细胞内表达情况的对比分析, 筛选出具有个体特异性、组织特异性、刺激特异性差异表达基因群, 并对基因群的变化特征和规律进行描述[1-2]. 我们利用基因芯片技术的同步高通量、高灵敏度、平行性的特性, 应用含有14 784条人类14KcDNA基因表达谱芯片, 以临床切除的胃腺癌组织和癌旁正常组织标本为研究对象, 对其差异基因进行研究, 探讨相关基因与胃腺癌生物学特性可能的内在联系.
手术病理检查证实的胃腺癌和癌旁正常组织标本4例, 术前均未经放疗和化疗. 男女各2例. 平均年龄49(42-71)岁, 高分化腺癌1例, 中低分腺癌2例, 低分化腺癌1例.无转移2例, 淋巴结转移1例, 肝转移1例, 癌旁组织取自距肿瘤基底部至少5 cm外黏膜. 芯片采用上海生物芯片有限公司提供的人14KcDNA表达谱芯片, cDNA克隆用通用引物进行PCR扩增, 扩增片段长度为800-3 000 bp, PCR产物用异丙醇纯化, 通过琼脂糖电泳监控PCR质量, 将样品cDNA溶解于3×SSC盐溶液中, 用Gene machine公司Omigrid Sloa点样仪进行自动点样, 将点样后的玻片进行扫描, 然后经水合1-2 h, 室温下干燥, 置于紫外交联仪中交联, 以2 g/LSDS、水及2 g/L硼氢化钠溶液处理, 晾干备用, 人14KcDNA表达谱芯片基因总数14 784个, 矩阵点数为18点×18点×48(亚矩阵), 点间距230 μm.
按TRIzol一步法抽提胃癌组织和癌旁正常组织总RNA, 使用QIAGEN Rneasy Kit进步一纯化总RNA, 应用琼脂糖凝胶电泳判断28S和18S的亮度比例评价总RNA的质量, 分离mRNA. 在20 μL逆转录体系中加入10 μg mRNA, 参照Schena et al[3]的方法逆转录cDNA探针并标记mRNA, 用Cy3-dUTP标记胃癌组织mRNA, 用Cy5-dUTP标记癌旁正常组织mRNA. 用无水乙醇将上述两种标记的探针沉淀, 再混合溶解在杂交试剂中(杂交试剂由上海生物芯片公司提供). 将含混合探针的杂交液与芯片分别经95℃变性后, 将杂交液滴于芯片点样区, 用盖玻片覆盖, 置于杂交舱中, 用Parafilm密封, 42℃温水浴杂交16 h. 芯片杂交完成后分别用洗液Ⅰ(2×SSC+2 g/L SDS)、洗液Ⅱ(0.1×SSC+2 g/L SDS)、洗液Ⅲ(0.1×SSC)洗涤10 min, 室温晾干. 采用激光共聚焦荧光扫描仪Agilent扫描芯片, 用QuantArrayR分析软件读取数据, 分辨率Scan resolution为10 μm, PMT为100%, 采用Genespring进行标准化处理分析, 得出Cy3和Cy5标记的强度值, 计算Ratio值为Cy3/Cy5. Cy3和Cy5信号荧光强度必须有一个>800; Ratio(Cy3/Cy5)比值的自然对数绝对值>2或<0.5, 判断为差异表达的基因.
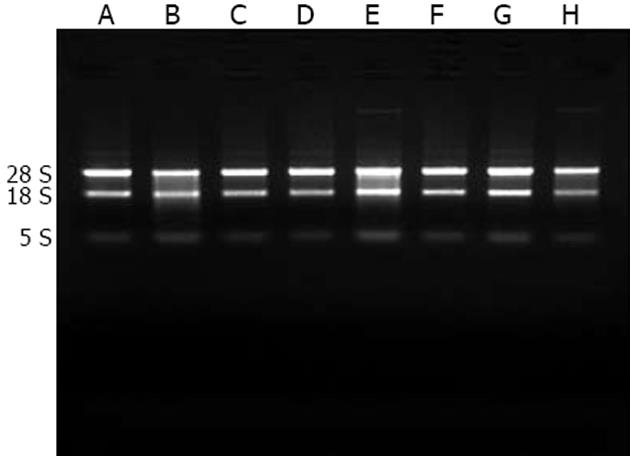
总RNA提取结果良好, 提取胃癌组织和癌旁正常组织中总RNA的含量在50-100 μg之间, 琼脂糖凝胶电泳结果分析RNA, 28S RNA和18S RNA条带清晰, 5S条带模糊, 说明RNA纯度和完整性较好(图1).
为了监控芯片和杂交过程, 设立5个阴性对照, 结果这些点的杂交信号极低, 说明杂交时无外源污染. 空白对照分散于48个亚矩阵中, 杂交信号低, 证明实验中不存在交叉污染. 在每个亚矩阵中设立的11个管家基因均为阴性, 在数据处理时能校对2种荧光标记之间的信号强度, 保证检测体系正常, 杂交结果的可靠性.
胃腺癌组织和癌旁正常组织(图2,3)中共同差异表达基因29条, 其中表达上调基因10条(表1), 表达下调基因19条(表2), 发现功能信息不明新基因2条, 均为上调基因.
| GenBank ID | ratio | gene_description |
| BG540617 | 6.24 | |
| NM_014297 | 2.76 | Homo sapiens ethylmalonic encephalopathy 1 (ETHE1) |
| NM_006412 | 3.302 | Homo sapiens 1-acylglycerol-3-phosphate O-acyltransferase 2(AGPAT2) |
| NM_004444 | 2.581 | Homo sapiens EphB4 (EPHB4) |
| NM_000308 | 2.231 | Homo sapiens protective protein for beta-galactosidase(galactosialidosis)(PPGB) |
| AW962980 | 5.451 | 'EST375053 MAGE resequences, MAGH Homo sapiens cDNA, mRNA sequence |
| AW054747 | 2.019 | Homo sapiens cDNA clone IMAGE:2501448 3' |
| NM_001091 | 9.894 | Homo sapiens amiloride binding protein 1 (ABP1) |
| NM_007329 | 22.75 | Homo sapiens deleted in malignant brain tumors 1 (DMBT1) |
| AW966777 | 3.018 | 'EST378851 MAGE resequences, MAGI Homo sapiens cDNA, mRNA sequence |
| GenBank ID | ratio | gene_description |
| NM_002065 | 0.138 | Homo sapiens glutamate-ammonia ligase (GLUL) |
| NM_198057 | 0.265 | Homo sapiens delta sleep inducing peptide, immunoreactor (DSIPI) |
| AL831930 | 0.113 | 'Homo sapiens mRNA; cDNA DKFZp761C2021 |
| NM_006472 | 0.243 | Homo sapiens thioredoxin interacting protein (TXNIP) |
| NM_198159 | 0.350 | Homo sapiens microphthalmia-associated transcription factor (MITF) |
| BC011762 | 0.211 | |
| NM_021069 | 0.485 | Homo sapiens Arg/Abl-interacting protein ArgBP2 (ARGBP2) |
| NM_018192 | 0.398 | Homo sapiens myxoid liposarcoma associated protein 4 (MLAT4) |
| NM_001647 | 0.436 | Homo sapiens apolipoprotein D (APOD) |
| NM_001823 | 0.094 | |
| NM_001442 | 0.364 | Homo sapiens fatty acid binding protein 4, adipocyte (FABP4) |
| NM_002084 | 0.258 | Homo sapiens glutathione peroxidase 3 (plasma) (GPX3) Homo sapiens protein phosphatase 1, regulatory (inhibitor) |
| NM_005398 | 0.427 | subunit 3C (PPP1R3C) |
| NM_018185 | 0.366 | Homo sapiens chromosome 13 open reading frame 17 (C13orf17) Homo sapiens vascular endothelial growth factor and type I |
| AF480883 | 0.472 | collagen inducible protein (VCIP) Homo sapiens creatine kinase, brain (CKB) |
| NM_001823 | 0.084 | Homo sapiens protein phosphatase 2 (formerly 2A), regulatory subunit B, |
| NM_002718 | 0.453 | alpha (PPP2R3A) |
| AF318382 | 0.278 | Homo sapiens pp9974 mRNA, complete cds |
| NM_003819 | 0.271 |
自从1972年Huebner和Todar提出细胞癌变的癌基因学说后, 对肿瘤机制的研究在近几年已进入分子水平. 目前认为肿瘤是一个多因素、多步骤、多基因参与的复杂过程, 是由一组基因的变化及其相互作用决定的, 不是一两个或少数几个基因所能决定的. 基因表达谱芯片的应用提供了相关基因的基因组学的证据, 突破了以往单个基因孤立研究的局限, 从整体上全面了解整个基因组多个基因的变化, 为在全基因组范围内寻找与肿瘤发生、发展相关的基因变化提供了一个强有力的手段.
我们应用含14 784个基因的基因芯片技术比较胃癌原发灶与癌旁正常组织之间基因表达谱的差异, 发现在两组间共同差异表达基因29条, 涉及癌基因、抑癌基因、免疫相关基因、细胞信号和传递蛋白相关基因、细胞周期蛋白相关基因和细胞受体相关基因, 与肿瘤的发生和发展过程有着密切的关系. 这一结果进一步证实胃腺癌的发生是一个多基因参与的过程, 多基因变异的结果. 在胃腺癌组织中呈上调表达的基因中, EphB4是受体酪氨酸蛋白激酶癌基因, 编码具有酪氨酸蛋白激酶活性的受体蛋白, 酪氨酸蛋白激酶在细胞生长、增殖、分化、转化过程中起着重要的作用, 与肿瘤的发生密切相关. Xia et al[4]报道抑癌基因p53, PTEN的缺失和酪氨酸激酶及其受体、胰岛素样生长因子及其受体的改变能够诱导EphB4基因表达, 增强酪氨酸蛋白激酶活性, 激活细胞内信号传递系统, 刺激细胞生长与增殖、转化恶变. 研究表明EphB4癌基因在前列腺癌、乳腺癌、头颈鳞癌等恶性肿瘤细胞中高表达, 促进肿瘤形成[4-6]. Xia et al[4]报道利用RNA干扰或反义寡核苷酸技术敲除EphB4编码的癌蛋白, 使肿瘤细胞生长、迁移和侵袭受到明显抑制并诱导凋亡, 这表明EphB4基因可以直接或间接参与肿瘤细胞的增殖调控过程.DMBT1基因是从染色体10q25.3-26.1分离出来的, 近来被认为是食管癌、胃癌、大肠癌和肺癌新的候选抑癌基因[7].Kang et al[8]报道在胃黏膜分化过程中, 通过激活细胞内信号分子蛋白激酶C(PKC)和细胞外调节蛋白激酶(ERK), 催化核内转录因子磷酸化反应, 调节基因的转录, 使DMBT1基因表达降低, 从而启动细胞分裂和增殖, 引起肿瘤的发生. 但是在本实验中原发胃腺癌组织DMBT1基因高表达的原因还不清楚.
与癌旁正常组织相比, 在胃腺癌组织表达下调的基因中, TXNIP基因是细胞氧化还原过程中的调控子, 也是转录因子的抑制剂, 通过抑制特异性基因表达所需的某些转录因子活性而抑制细胞分化. Takahashi et al[9]报道利用原位杂交技术检测出TXNIP基因转录多发生在细胞分化末期, 其mRNA水平在胃肠道上皮细胞中低表达, 而在淋巴滤泡里的淋巴细胞中高表达, 这说明TXNIP基因可能在胃肠道上皮细胞分化和黏膜免疫反应中起作用. 作为血浆脂质转运系统中的ApoD基因是乳腺癌和前列腺癌的雄激素调节蛋白[10], 对正常细胞有负向调节作用, 通过选择性地抑制血小板衍生的生长因子BB诱导细胞外信号调节激酶的核转运, 从而抑制血管平滑肌细胞增殖, 对一些肿瘤细胞的侵袭具有抑制作用, 被看作是肿瘤细胞分化和生长停滞的标记物[11], 在乳腺癌、前列腺癌、卵巢癌、子宫内膜癌和视网膜母细胞癌中低表达[10,12]. 1960年代人们第一次发现鼠MITF基因转录因子突变, 之后在基因座中发现了24个自发的和诱发的MITF基因突变体[13]. MITF基因编码具有碱性区螺旋环-螺旋-拉链亚型Myc基因家族的转录因子, 与结构相似的基因形成同质二聚体或异质二聚体, 此二聚体与DNA特异性结合, 起转录调节作用, 引起细胞增殖, 产生促癌作用. 本研究中MITF基因表达下调与以上作用机制不同, 很可能存在其它的致癌途径. Hershey et al[14]报道MITF基因是破骨细胞、黑色素细胞、视网膜上皮细胞、肥大细胞和自然杀伤细胞发育过程中所必需的转录因子, 本研究中MITF基因表达下调是否与抑制自然杀伤细胞转录调节功能导致癌变的发生有关, 有待进一步证实. CKB基因在卵巢癌细胞和神经胶质瘤细胞等恶性肿瘤细胞中高表达, 并能促进癌细胞转移[15-16]. 而本研究中胃腺癌组织与癌旁正常组织相比, CKB基因表达下调, 其机制有待进一步探讨.
通过上述研究, 我们已经筛选出一些胃腺癌相关表达基因, 有助于人们从分子水平理解胃腺癌发生的病因, 为寻找理想的胃腺癌肿瘤标志物和探索基因治疗提供可靠的线索. 但由于本研究为初步试验结果, 许多基因在胃腺癌发生过程中存在什么样的时空关系、彼此有何联系, 以及哪些基因是真正的胃腺癌易感基因, 目前还了解甚少, 有待进一步研究.
本研究通过基因芯片技术获得胃癌组织和癌旁正常组织在不同状态下的基因表达谱, 通过生物信息学处理, 对其进行比较, 确定正常组织和胃癌组织的基因表达上的差异以及不同基因在表达上的相关性, 进而帮助人们在基因水平上深入了解胃癌的发生、发展.
基因芯片是目前筛选差异表达基因较为成熟的方法, 寻找并克隆与肿瘤相关差异表达基因是肿瘤基因诊断和基因治疗的关键.
本研究利用基因芯片技术筛选胃腺癌组织和癌旁正常组织间的差异表达基因, 对于两种组织之间已知基因表达的差异提供了初步的研究结果, 表明胃癌发生发展过程中多基因参与的特性. 具有一定的探索性.
电编: 李琪 编辑:潘伯荣
| 1. | Yeatman TJ. The future of cancer management: translating the genome, transcriptome, and proteome. Ann Surg Oncol. 2003;10:7-14. [PubMed] [DOI] |
| 2. | Golub TR, Slonim DK, Tamayo P, Huard C, Gaasenbeek M, Mesirov JP, Coller H, Loh ML, Downing JR, Caligiuri MA. Molecular classification of cancer: class discovery and class prediction by gene expression monitoring. Science. 1999;286:531-537. [PubMed] [DOI] |
| 3. | Schena M, Shalon D, Davis RW, Brown PO. Quantitative monitoring of gene expression patterns with a complementary DNA microarray. Science. 1995;270:467-470. [PubMed] [DOI] |
| 4. | Xia G, Kumar SR, Masood R, Zhu S, Reddy R, Krasnoperov V, Quinn DI, Henshall SM, Sutherland RL, Pinski JK. EphB4 expression and biological significance in prostate cancer. Cancer Res. 2005;65:4623-4632. [PubMed] [DOI] |
| 5. | Cromer A, Carles A, Millon R, Ganguli G, Chalmel F, Lemaire F, Young J, Dembele D, Thibault C, Muller D. Identification of genes associated with tumorigenesis and metastatic potential of hypopharyngeal cancer by microarray analysis. Oncogene. 2004;23:2484-2498. [PubMed] [DOI] |
| 6. | Wu Q, Suo Z, Risberg B, Karlsson MG, Villman K, Nesland JM. Expression of Ephb2 and Ephb4 in breast carcinoma. Pathol Oncol Res. 2004;10:26-33. [PubMed] [DOI] |
| 7. | Imai MA, Moriya T, Imai FL, Shiiba M, Bukawa H, Yokoe H, Uzawa K, Tanzawa H. Down-regulation of DMBT1 gene expression in human oral squamous cell carcinoma. Int J Mol Med. 2005;15:585-589. [PubMed] [DOI] |
| 8. | Kang W, Nielsen O, Fenger C, Leslie G, Holmskov U, Reid KB. Induction of DMBT1 expression by reduced ERK activity during a gastric mucosa differentiation-like process and its association with human gastric cancer. Carcinogenesis. 2005;26:1129-1137. [PubMed] [DOI] |
| 9. | Takahashi Y, Ishii Y, Murata A, Nagata T, Asai S. Localization of thioredoxin-interacting protein (TXNIP) mRNA in epithelium of human gastrointestinal tract. J Histochem Cytochem. 2003;51:973-976. [PubMed] [DOI] |
| 10. | Rodriguez JC, Diaz M, Gonzalez LO, Sanchez J, Sanchez MT, Merino AM, Vizoso F. Apolipoprotein D expression in benign and malignant prostate tissues. Int J Surg Inv. estig. 2000;2:319-326. [PubMed] |
| 11. | Sarjeant JM, Lawrie A, Kinnear C, Yablonsky S, Leung W, Massaeli H, Prichett W, Veinot JP, Rassart E, Rabinovitch M. Apolipoprotein D inhibits platelet-derived growth factor-BB-induced vascular smooth muscle cell proliferated by preventing translocation of phosphorylated extracellular signal regulated kinase 1/2 to the nucleus. Arterioscler Thromb Vasc Biol. 2003;23:2172-2177. [PubMed] [DOI] |
| 12. | Alvarez ML, Barbon JJ, Gonzalez LO, Abelairas J, Boto A, Vizoso FJ. Apolipoprotein D expression in retinoblastoma. Ophthalmic Res. 2003;35:111-116. [PubMed] [DOI] |
| 13. | Steingrimsson E, Copeland NG, Jenkins NA. Melanocytes and the microphthalmia transcription factor network. Annu Rev Genet. 2004;38:365-411. [PubMed] [DOI] |
| 14. | Hershey CL, Fisher DE. Mitf and Tfe3: members of a b-HLH-ZIP transcription factor family essential for osteoclast development and function. Bone. 2004;34:689-696. [PubMed] [DOI] |
| 15. | Huddleston HG, Wong KK, Welch WR, Berkowitz RS, Mok SC. Clinical applications of microarray technology: creatine kinase B is an up-regulated gene in epithelial ovarian cancer and shows promise as a serum marker. Gynecol Oncol. 2005;96:77-83. [PubMed] [DOI] |
| 16. | Willis D, Zhang Y, Molloy GR. Transcription of brain creatine kinase in U87-MG glioblastoma is modulated by factor AP2. Biochim Biophys Acta. 2005;1728:18-33. [PubMed] |