修回日期: 2005-01-28
接受日期: 2005-02-02
在线出版日期: 2005-04-01
目的: 克隆幽门螺杆菌(Helicobacter pylori, H. pylori)前炎症外膜蛋白基因oipA, 并对其进行生物信息学分析.
方法: 利用GenBank已公布序列设计引物, 用PCR进行扩增后, 酶切, 连入载体pGEX-5X-1, 并进行序列测定. 用数据库Blast, NCBI CDD, Swiss-model, Propred以及生物学软件Omiga v2.0和Antheprot v5.0分析其生物学特性.
结果: 克隆片段与GenBank公布序列完全一致. 经Blast比对未发现与oipA同源的其他种属基因, 通过NCBI CDD未找到已知保守区域和oipA 编码氨基酸序列相关. 应用Omiga v2.0, Antheprot v5.0软件预测显示该蛋白具有良好的疏水性和抗原性, 将该分析结果和Propred结果比对得到预测的抗原表位.
结论: 应用生物信息学技术, 对幽门螺杆菌基因oipA进行分析, 将为H. pylori与相关致病发生机制研究, H. pylori疫苗的开发等工作奠定一定基础.
引文著录: 陈阳, 李健峰, 姬秋彦, 闫玲梅, 龙海亭, 徐维明. 幽门螺杆菌oipA基因的克隆及其生物信息学分析. 世界华人消化杂志 2005; 13(7): 867-870
Revised: January 28, 2005
Accepted: February 2, 2005
Published online: April 1, 2005
AIM: To clone and characterize the oipA gene of Helicobacter pylori (H. pylori).
METHODS: The primers were designed based on the sequence in GenBank. PCR was used to amplify the oipA gene. The amplified fragment was inserted into plasmid pGEX-5X-1 for nucleotide sequencing. The bioinformatics analysis was carried out by using databases of Blast, NCBI CDD, Swiss-model, Propred and software of Omiga v2.0 and Antheprot v5.0.
RESULTS: The oipA gene was successfully cloned with the same sequence as that published in GenBank. No homeotic gene was found in other species through the Blast, and no conserved domain was found through NCBI CDD. Potential hydrophobicity and antigenicity were predicted by software Omiga v2.0 and Antheprot v5.0. The epitopes were predicted by comparing the results from Omiga v2.0, Antheprot v5.0 and Propred.
CONCLUSION: Bioinformatics analysis of gene oipA may help to elucidate the pathogenesis of H. pylori related diseases and to develop H. pylori vaccines.
- Citation: Chen Y, Li JF, Ji QY, Yan LM, Long HT, Xu WM. Cloning and bioinformatics analysis of gene oipA of Helicobacter pylori. Shijie Huaren Xiaohua Zazhi 2005; 13(7): 867-870
- URL: https://www.wjgnet.com/1009-3079/full/v13/i7/867.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v13.i7.867
幽门螺杆菌(Helicobacter pylori, H. pylori)感染是慢性胃炎和消化性溃疡的主要病因, 与胃腺癌、胃黏膜相关性淋巴样组织恶性淋巴瘤的发生亦密切相关[1]. 在H. pylori定植和致病的过程中各种外膜蛋白起着重要作用. 近年来的临床统计研究中发现幽门螺杆菌oipA基因所编码的外膜蛋白和宿主高水平的IL-8分泌, 严重地中性粒细胞聚集以及消化性溃疡密切相关[2]. 在体外实验中亦发现oipA能有效提高胃表皮细胞IL-8的分泌[3]. 还有报道认为oipA和Cag PAI能够分别独立的影响宿主IL-8的分泌[4]. 对oipA基因的克隆和生物信息学的分析将有助于对其理化特性、同源性以及结构和功能的进一步认识, 为研究H. pylori致病机制和H. pylori疫苗的开发等工作打下一定基础.
幽门螺杆菌Hp 26695购自中国预防医学科学院传染病预防控制所. 菌株E. coli DH5α, 质粒pGEX-5x-1为本所保存. 限制性内切酶BamHⅠ, EcoRⅠ, Taq酶, T4 DNA聚合酶、DL2000购自Takara 公司, 脑心浸液培养基购自BD Difco公司, 幽门螺杆菌选择性添加剂购自Oxoid公司, 细菌DNA抽提试剂盒购自上海华舜生物工程有限公司, DNA片段快速提取试剂盒、质粒快速提取试剂盒购自北京鼎国生物技术有限责任公司, 其他试剂为国产分析纯.
H. pylori染色体DNA的提取, 通过在37 ℃下, 50 mL/L O2, 850 mL/L N2, 100 mL/L CO2气体环境下已培养72 h 的H. pylori固体培养基上刮取生长良好的H. pylori菌落, 用细菌DNA抽提试剂盒制备. 具体操作按说明书指导进行. 质粒的提取及纯化使用质粒快速提取试剂盒制备. 具体操作按说明书指导进行. 目的基因的PCR扩增根据GenBank公布序列NC_000915 REGION: 684774. 685691用Prime Primer 5.0设计引物, 由上海生工公司合成. 序列如下: oipA sense: 5'-CG GGATCC CCA TGA AAA AAA GCT CTC TTA CT-3'; oipA anti-sense: 5'-CG GAATTC TTA ATG TTT GTT TTT AAA GTT-3'EcoRⅠ. 热启动法进行PCR, 94℃变性60 s, 52℃退火50 s, 72℃ 延伸70 s, 30个循环后再72℃延伸10 min.8 g/L琼脂糖凝胶电泳观察扩增结果. DNA片段的酶切、连接、转化和阳性克隆的鉴定详见参考文献[5]. 用质粒快速抽提试剂盒抽提经双酶切鉴定的重组克隆质粒, 送交上海生工公司测序. 基因片段同源性分析登陆NCBI使用Blastn 2.2.10在nr数据库中对oipA基因的同源性进行比对分析; 使用Blastp在nr数据库中对oipA编码氨基酸序列的同源性进行比对, 使用保守序列数据库(conserved domain database CDD)寻找oipA编码氨基酸序列是否和已知保守序列相关. 编码蛋白理化特性应用序列分析软件包Omiga 2.0分析oipA编码氨基酸序列. 编码蛋白结构预测应用蛋白质序列分析软件包Antheprot v5.0采用GORI, Gibrat, Homologue, DPM, Predator共5种算法对oipA的二级结构进行预测分析. 登陆Swiss-Model自动蛋白同源建模数据库进行3D结构预测. 编码蛋白免疫表位分析应用序列分析软件包Omiga v2.0和Antheprot v5.0采用Parker算法对其潜在的免疫表位进行预测分析. 登陆Propred(http://www.imtech.res.in/raghava/propred)分析可能被MHCⅡ所的呈递的oipA肽段[6].
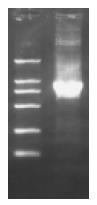
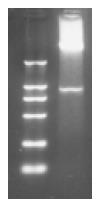
PCR产物电泳分析后发现在750-1 000 bp间有一明显条带(图1), 大小与预计oipA基因相符. 重组质粒酶切后电泳鉴定结果(图2), 酶切片断大小和预期一致. oipA基因片段序列分析结果与GenBank公布序列一致.
2.2.1 nr数据库序列分析: 在目前nr数据库2793175条非冗余序列中分析, 得到153个期望值小于10的oipA基因同源序列, 但均为H. pylori不同菌种间的相关序列. 使用Blastp 2.2.10在目前nr数据库2, 244, 936条蛋白序列中分析, 得到172个期望值小于10的oipA同源蛋白, 但均为H. pylori不同菌种间的所属蛋白, 且主要为外膜蛋白和黏附蛋白. 使用NCBI保守序列搜索服务器(CD-Search service v2.02)在数据库CDD v2.02-10897 PSSMs和Smart v4.0-663 PSSMs中分析, 得到oipA编码氨基酸序列和目前已知的氨基酸保守序列无关联.
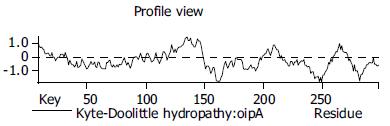
2.0分析得到oipA的分子质量为34.187 ku, 等电点为10.118, 280 nm紫外摩尔消光系数为1.340(1 mg/L), 在pH7.0时所带电荷为18.358. 应用Antheprot v5.0对其疏水性进行分析(图3), oipA N'末端3-14残基, 蛋白中部117-144残基以及C'末端258-272为3个较明显的疏水片段. oipA中128-140残基为Antheprot预测的跨膜区.
v5.0采用GORI, Gibrat, Homologue, DPM, Predator共5种算法对oipA的二级结构进行预测分析. 得到oipA可能存在4个较明显的α螺旋片段, 他们大约位于1-20位残基, 40-90位残基, 100-115位残基和157-196位残基间. oipA中没有明显的β折叠区, 4个由较小β折叠串联构成的区域位于20-40位残基, 116-144位残基, 197-218位残基和236-269位残基间. 转角和无规则卷曲主要散布于4个β折叠区中. 总体上看oipA在靠近N末端一侧结构较为简单, 除在20-40位残基间存在部分β折叠和无规则卷曲外, 其他部分主要以α螺旋为主, 而oipA的C末端一侧则有各种二级结构间或出现. 在使用Swiss-Model自动蛋白同源建模数据库进行3D结构预测时, 由于没有找到和oipA有较高同源性的已知3D结构的蛋白序列, 预测没能得以进行.
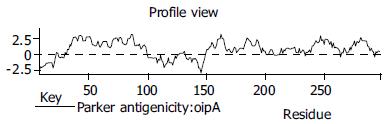
v5.0和Omiga v2.0预测后得到7个预测分值较高的区域(图4). 登陆Propred后将oipA序列和51个HLA等位基因序列进行分析后得到107个可能的MHCⅡ呈递片段. 将107个表位与Antheprot v5.0, Omiga v2.0所分析的高免疫原性区域比对得到结果(表1).
| Parker算法 | 同51个等位HLA比对分析所得表位 |
| 13-56 aa | WLHAERNGF(2/51) LNFLEGSYI(2/51) |
| 75-95 aa | No Peptides were predicted |
| 150-166 aa | LLINWTNDK(14/51) WTNDKRASQ(7/51) YVERRVKGLS(5/51) |
| 174-200 aa | FYKDMTGRT(4/51) |
| 215-238 aa | FNQVKSRTI(4/51) VKSRTIFQL(1/51) FQLQGKFGV(12/51) |
| 240-260 aa | YLGGSSVEL(2/51) |
| 272-301 aa | YKRVVSVYL(4/51) VYLNYTYNF(4/51) |
自1999年Yamaoka et al[7]推测在分离的H. pylori外膜蛋白中分子质量约为3.4 ku的条带很可能是和严重的嗜中性粒细胞浸润和高浓度IL-8密切相关的oipA以来, 又有进一步的临床统计和实验结果表明oipA可能引起宿主T细胞免疫偏移引发消化性溃疡. 但目前在国内外对oipA的研究还多集中于临床统计研究和体外实验, 关于oipA结构与功能以及免疫性评价的具体实验性结果还未见报道, 这可能和外膜蛋白oipA本身疏水性强, 等电点高, 不易表达和分离纯化有关. 使用数据库对oipA基因的同源性和保守性进行分析, 将有助于对其未知功能做进一步推测. 在使用Blastn对oipA基因进行分析后并未得到与其密切相关的其他种属基因, 这可能是因为Blastn以寻找较高分值匹配的策略, 对较远关系不太适用. 但是在使用Blastp应用取代矩阵策略寻找oipA较远关系时, 也未得到与其密切相关其他种属的蛋白. 应用NCBI CDD分析也没能得到oipA基因编码氨基酸序列和目前数据库中保守序列匹配. 这可能是因为以上数据库的信息相对物种的多样性与变化性还显得比较有限, 这也可能意味着oipA通过某种特殊的尚未知的信号传导方式引起了宿主的病变. 而这有待实验的进一步证明.
对蛋白基本理化特性, 2级结构, 3D结构以及抗原表位的分析与预测, 将有助于蛋白克隆表达, 分离纯化和功能区域的分析, 有效的辅助研究者对实验的设计和实验结果的分析. 由于oipA疏水性强, 等电点高等原因本实验室在调整各相关条件后亦未能实现oipA的表达. 目前在国内外报道中也还未见关于oipA全蛋白的表达. 但oipA是临床统计上和各种胃肠道疾病密切相关的因子, 很可能是抗H. pylori的重要保护性抗原, 应用Antheprot v5.0和Omiga v2.0 Parker算法根据oipA基因编码氨基酸序列得到的分析结果和Propred HLA数据库通过oipA基因序列和HLA等位基因序列比对分析预测结果进行比较, 是对oipA免疫表位预测的一种较为有效的方法. 然而H. pylori 致病过程并非为单一因子所致, 有效地应用当前不断丰富的序列同源性分析, 结构功能预测, 表位分析, 信号通路分析等生物信息学技术将会在H. pylori的诊断与治疗, H. pylori与相关致病发生机制研究, H. pylori疫苗的开发等领域发挥积极作用.
编辑: 张海宁 电编: 潘伯荣
| 1. | Lamarque D, Peek Jr RM. Pathogenesis of Helicobacter pylori infection. Helicobacter. 2003;8:21-30. [PubMed] |
| 2. | Yamaoka Y, Kikuchi S, el-Zimaity HM, Gutierrez O, Osato MS, Graham DY. Importance of Helicobacter pylori oipA in clinical presentation, gastric inflammation, and mucosal interleukin 8 production. Gastroenterology. 2002;123:414-424. [PubMed] |
| 3. | Yamaoka Y, Kudo T, Lu H, Casola A, Brasier AR, Graham DY. Role of interferon-stimulated responsive element-like element in interleukin-8 promoter in Helicobacter pylori infection. Gastroenterology. 2004;6:1030-1043. [PubMed] |
| 4. | Lamarque D, Tran Van Nhieu J, Breban M. What are the gastric modifications induced by acute and chronic Helicobacter pylori infection? Gastroenterol Clin Biol. 2003;27:391-400. [PubMed] |
| 6. | Singh H, Raghava GP. ProPred: prediction of HLA-DR binding sites. Bioinformatics. 2001;17:1236-1237. [PubMed] |
| 7. | Yamaoka Y, Kodama T, Graham DY, Kashima K. Search for putative factions of Helicobacter pylori: the low-molecular weight (33-35kDA) antigen. Dig Dis Sci. 1998;43:1482-1487. [PubMed] |