Copyright
©The Author(s) 2005. Published by Baishideng Publishing Group Inc. All rights reserved.
丙型肝炎病毒非结构蛋白2反式调节基因NS2TP的克隆化
张黎颖, 成军, 邓红, 刘妍, 王琳
张黎颖, 成军, 北京地坛医院传染病研究所 北京市 100011
邓红, 西安交通大学第二医院感染科 陕西省西安市 710004
刘妍, 王琳, 中国人民解放军第302医院传染病研究所基因治疗研究中心 北京市 100039
张黎颖, 女, 1978-10-26生, 陕西省西安市人, 汉族, 2002年毕业于西安交通大学临床医学系, 获医学学士学位, 2005年毕业于西安交通大学医学系, 获医学硕士学位, 主要研究方向是肝炎病毒与肝细胞相互作用的分子生物学机制.
ORCID number: $[AuthorORCIDs]
基金项目: 国家自然科学基金攻关项目, No. C03011402, No. C30070689; 军队"九、五"科技攻关项目, No. 98D063; 军队回国留学人员启动基金项目, No. 98H038; 军队"十、五"科技攻关青年基金项目, No. 01Q138; 军队"十、五"科技攻关项目, No. 01MB135.
电话: 010-64481639 传真: 010-64281540
收稿日期: 2005-01-11
修回日期: 2005-03-22
接受日期: 2005-05-25
在线出版日期: 2005-07-28
目的: 克隆化HCV非结构蛋白2(NS2)蛋白反式调节靶基因(NS2TP).
方法: 依据我们构建的HCV NS2反式调节基因差异表达的cDNA消减文库筛选结果, 利用分子生物学与生物信息学技术获得新基因NS2TP的编码序列, 设计特异性引物, 并对其进行克隆化研究.
结果: NS2TP基因编码区为456核苷酸(nt), 编码产物为151氨基酸残基(aa), 经核苷酸序列数据库(GenBank)和蛋白质一级结构序列数据库(SwissProt)同源序列的搜寻, 与已知基因序列和蛋白序列之间没有显著同源性, 属于未知功能新基因.
结论: 发现了HCV NS2反式调节新的靶基因.
关键词: 丙型肝炎病毒; 非结构蛋白2; 反式调节; 克隆
引文著录: 张黎颖, 成军, 邓红, 刘妍, 王琳. 丙型肝炎病毒非结构蛋白2反式调节基因NS2TP的克隆化. 世界华人消化杂志 2005; 13(14): 1700-1704
Cloning and identification of gene NS2TP transregulated by non-structural protein 2 of hepatitis C virus
Li-Ying Zhang, Jun Cheng, Hong Deng, Yan Liu, Lin Wang
Li-Ying Zhang, Jun Cheng, Institute of Infectious Diseases, Beijing Ditan Hospital, Beijing 100011, China
Hong Deng, Department of Infectious Diseases, the Second Hospital of Xi'an Jiaotong University Xi'an 710004, Shaanxi Province, China
Yan Liu, Lin Wang, Research Center of Gene Therapy, the 302 Hospital of Chinese PLA, Beijing 100039, China
Supported by National Science Foundation of China, No. C03011402, No.C30070689; Returned Scholar Fundation of General Logistics Department of Chinese PLA, No. 98H038; the Science and Technique Foundation of Chinese PLA during the 9th and 10th Five-Year Period, No. 98D063, No.01MB135; and the Science and Technique Foundation of Chinese PLA for Young Scholars during the 10th Five-Year Plan Period, No. 01Q138
Correspondence to: Dr. Jun Cheng, Institute of Infectious Diseases, Beijing Ditan Hospital, Beijing Dongcheng District, 13 Anwai Avenue, Beijing 100011, China. cj@genetherapy.com.cn
Received: January 11, 2005
Revised: March 22, 2005
Accepted: May 25, 2005
Published online: July 28, 2005
0 引言
丙型肝炎病毒(HCV)的非结构蛋白2(NS2)除了作为非结构蛋白3(NS3)丝氨酸蛋白酶的辅助因子, 在HCV多蛋白的酶解过程中具有重要作用. 关于NS2蛋白的生物学功能目前还知之甚少[1]. 为了研究NS2蛋白在肝细胞中新的生物学功能, 发现并鉴定一系列NS2蛋白反式调节的靶基因, 其中包括一些未知功能的新基因[2-3]. 我们在成功构建转染HCV NS2表达载体的HepG2细胞差异表达基因cDNA消减文库的基础上, 克隆未知功能基因NS2TP, 为今后更加广泛深入地研究新基因的生物学功能打下基础, 并为研究HCV NS2的反式调节作用、丙型肝炎发病机制提供了新的思路.
1 材料和方法
1.1 材料
肝母细胞瘤细胞系HepG2及感受态大肠杆菌DH5a为本室保存, Taq酶、T4 DNA连接酶、pGEM-T(Promega), 玻璃奶回收试剂盒购自博大公司, DNA序列测定由上海联合基因公司完成. 利用抑制性消减杂交技术构建NS2反式调节基因差异表达的cDNA消减文库, 挑选30个克隆测序, 与GenBank数据库进行初步比较. 3个克隆未检索到任何已知同源基因序列, 可能代表了某些新基因, 将其一未知功能基因命名为NS2TP, 发现其开放读码框长456 bp, 编码151个氨基酸残基.
1.2 方法
应用Vector 8.0版软件将推断的ORF翻译为氨基酸序列, 并将氨基酸序列与储存在GenBank中已知的氨基酸的进行比较, 并进行功能分析. 根据NS2TP的全长编码基因, 设计上下游引物. 上游引物5'-GAA TTC ATG CCG CGT GGA AGC CGA-3', 下游引物5'-GGA TCC TTA GGC CAA TCC GTT TGC-3'.提取HepG2细胞的总RNA, 进行反转录, 以反转录产物为模板进行PCR, PCR参数如下:94 ℃ 5 min预变性, 94 ℃ 30 s变性, 62 ℃ 30 s退火, 72 ℃ 30 s延伸, 共35个循环, 72 ℃延伸10 min. 将PCR产物在9 g/L琼脂糖凝胶中电泳, 切取目的片段, 玻璃奶法回收PCR产物, 与pGEM-T载体连接, 转化DH5a感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养18 h. 挑取阳性菌落, 增菌. 提取质粒进行限制性酶切分析鉴定, 选择经鉴定的菌落送测序.
2 结果
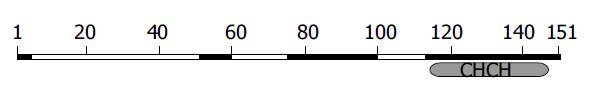
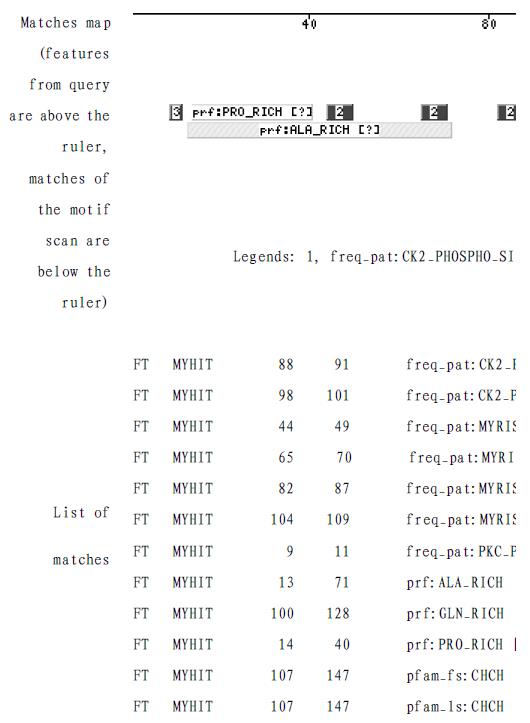
新基因的开放读码框架(ORF)长度为456个核苷酸(nt), 编码产物由151个氨基酸残基(aa)组成(图1). 新基因的核苷酸序列在GenBank中登录, GenBank收录号为AY605046. 在NIH网站应用rpsBLAST软件以NS2TP基因编码的推断氨基酸序列进行搜索, 发现氨基酸序列114-147 aa是一CHCH 结构域([coiled coil 1]-[helix 1]-[coiled coil 2]-[helix 2] domain, CHCH domain)(图2), Marchler-Bauer et al[4]发现了这一保守结构域, 并与相关成员比较发现三种蛋白亚群具有该结构域, 分别是S(Small, 小蛋白), N(N-terminal extended, N末端扩展蛋白)和 C(C-terminal extended, C末端扩展蛋白), 结构域中的每一个螺旋的C-X9-C基序含有两个半胱氨酸残基. N末端扩展蛋白包括一些特殊蛋白, 例如Cox19蛋白、Mrp10、NDUFA8.Cox19是酿酒酵母和基因, 编码11 ku的蛋白, 存在于胞质和线粒体中, 为细胞色素氧化酶表达所需[5-6]. 应用internet上motif scan软件, 以NS2TP基因编码的氨基酸序列进行搜索, 发现该蛋白具有以下基序:2个酪蛋白激酶2(CK2)磷酸化位点(位于氨基酸残基88-91 aa, 98-101 aa), 4个N-端酰基化位点(44-49 aa, 65-70 aa, 82-87 aa, 104-109 aa), 1个蛋白激酶C(PKC)磷酸化位点(位于氨基酸残基9-11 aa), 丙氨酸富集区(位于氨基酸残基13-71 aa), 谷氨酰胺富集区(位于氨基酸残基100-128 aa), 脯氨酸富集区(位于氨基酸残基14-40 aa), 1个CHCH结构域(位于氨基酸残基107-147 aa)(图3).
图2 NS2TP蛋白保守结构域的生物信息学分析结果.
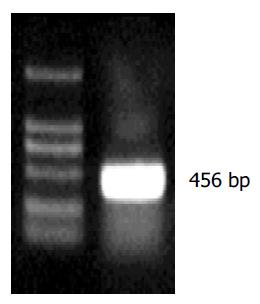
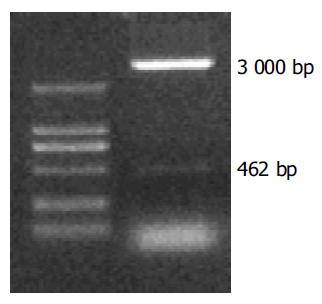
以HepG2细胞系cDNA文库为模板, PCR反应后经0.9%琼脂糖凝胶电泳, 可见长度为456 bp的电泳条带, 见图4.PCR产物与T-载体连接, 转化大肠杆菌, 提取质粒进行酶切鉴定, 结果见图5. 测序结果完全符合NS2TP的推断序列, 见图6, 表明我们已顺利得到NS2TP基因编码序列.
图5 pGEM-T-NS2TP质粒EcoRI/BamHI酶切图.
3 讨论
丙性肝炎病毒非结构蛋白2(HCV NS2)位于HCV多蛋白前体810-1 026 aa之间, 编码216个氨基酸残基. 已有研究发现NS2是一种金属/半胱氨酸蛋白酶, NS2-3酶活性的存在成为目前治疗丙型肝炎的一个药物作用靶点[7].Yamaga et al[8]研究NS2的膜定位机制发现, NS2本身具有至少2个与其膜定位相关的内部信号序列, 分别位于氨基酸839-883 aa和928-960 aa区. 他们还预测NS2具有4个跨膜结构, 亲水性图谱和NS2的拓扑学分析显示四个跨膜区分别位于氨基酸810-835 aa, 843-866 aa, 872-919 aa和928-956 aa区, 其氨基末端和羧基末端均定位在内质网腔内, 但NS2的功能还有待于进一步研究[9]. 抑制性消减杂交技术(suppression subtractive hybridization, SSH)是20世纪90年代后期建立的一种基因克隆的新技术, 可以快速有效地检测到差异表达的基因, 而且其可能检测到的结果具有无限可能性, 与高通量筛选的技术-基因芯片技术相比, 发现新基因是其独特的优势[10-12]. 本实验室采用SSH技术成功筛选了多条新基因[13-15]. 确定一个新基因的编码序列, 研究该基因的表达与调控, 编码蛋白的结构与功能, 确定新基因的生物学和医学意义, 是医学分子生物学研究领域中一项具有挑战性的工作[16]. 本项研究发现了NS2TP基因, 这是一种未知功能的新基因, 对于这一新基因结构与功能的研究虽然还是空白, 但是从生物信息学分析结果来看, NS2TP基因编码产物具有CHCH结构域, 根据其同源结构蛋白的亚细胞定位和功能研究, 推测NS2TP基因编码产物存在于胞质和线粒体中, 与细胞色素氧化酶功能相关.
NS2TP基因开放读码框架(ORF)长度为456个核苷酸(nt), 编码产物由151个氨基酸残基(aa)组成, GenBank 收录号为AY605046. 应用motif scan分析发现, NS2TP可能具有以下基序:2个酪蛋白激酶2(CK2)磷酸化位点(位于氨基酸残基88-91, 98-101), 4个N端酰基化位点(44-49, 65-70, 82-87, 104-109), 1个蛋白激酶C(PKC)磷酸化位点(位于氨基酸残基9-11), 丙氨酸富集区(位于氨基酸残基13-71), 谷氨酰胺富集区(位于氨基酸残基100-128), 脯氨酸富集区(位于氨基酸残基14-40), 1个CHCH结构域(位于氨基酸残基107-147).CK2是1969年Pinna et al[17-24]发现的酪蛋白激酶, 到目前CK2的内源性底物已发现307个, 这些蛋白质底物具有非常重要的生物学作用, 其中60余个是转录因子, 40余个与基因转录和表达有关的核蛋白, 10多个翻译因子和80多个在细胞信号转导中发挥不同功能的蛋白质(这些蛋白质大多数组成了细胞的信号转导网络).NS2TP作为CK2的潜在底物, 可能具有类似的生物学功能, 需要进一步研究. 酰基化和磷酸化是蛋白质合成后的修饰方式, 不同氨基酸富集区可能会决定蛋白质结构和功能的不同. 这些结果均可对研究NS2TP新基因的结构的功能有一定的指导意义.
生物信息学技术伴随计算机网络的发展而产生, 其强大的功能使其已经成为生命科学研究的重要手段[25-30]. 我们成功克隆HCV NS2下调靶基因NS2TP, 并利用生物信息学分析技术对其进行了初步分析, 为新基因的研究奠定基础, 同时也为研究HCV NS2的生物学功能, 阐明HCV的致病机制提供了新的方向.