修回日期: 2005-03-05
接受日期: 2005-03-16
在线出版日期: 2005-05-15
良好的基因芯片实验设计是充分发挥基因芯片技术优势的前提, 而研究目的是实验设计的基础.本文介绍了参照、平衡和环形三种常用的单因子、时间序贯和复因子等基本的基因芯片实验设计方案;探讨了重复实验、混合样本和样本规模等影响基因芯片实验设计的因素, 为基因芯片实验设计提供参考.
引文著录: 吴斌, 林乔, 王米渠, 王建国. 试论影响基因芯片实验设计的因素. 世界华人消化杂志 2005; 13(10): 1206-1209
Revised: March 5, 2005
Accepted: March 16, 2005
Published online: May 15, 2005
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2005; 13(10): 1206-1209
- URL: https://www.wjgnet.com/1009-3079/full/v13/i10/1206.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v13.i10.1206
基因芯片对于同时研究成千上万的基因表达是一个强有力的技术, 这种新技术在生物学、农学、医学等都有重要的应用, 但严谨的实验设计是充分发挥基因芯片技术优势的基础[1].基因芯片实验同其他实验设计一样需要考虑因素与水平, 但基因芯片实验又有他的特殊性, 因此为了减少基因芯片实验和数据分析的误差, 仔细地进行实验设计显得尤为重要.我们以自己研究的经验为基础结合国外研究动态对基因芯片实验设计探讨如下.
基因表达谱的差异包括三层[2], 一是生物差异(上层):生物差异是所有生物的内在本质, 除遗传和环境因素影响外与样本有密切的关系.如不同人群中的个体差异、同一个体不同标本之间的差异.二是技术差异(中层):技术差异是由于样本的提取、标记和杂交等引起的差异, 如同样的mRNA样本不同标记反应之间的差异等.三是测量误差(下层):测量误差是与阅读荧光信号相关, 因为荧光信号可能被芯片上的灰尘等所影响.基因表达谱的研究目的就是要寻找生物差异, 故实验设计的目的是尽量减少技术差异和测量差异对实验的影响, 从而使数据的分析和结果的解释尽可能简单有力.基因芯片实验设计的问题包括决定样本标记什么样的染料?那些样品在同一张芯片上杂交?另外如果RNA样本有限, 或者芯片数目有限制(如研究经费不足), 我们又应当如何设计实验等一系列问题.但基因芯片设计最重要取决于研究的目的, 只有当研究的设计与目的一致时我们才可能达到我们的研究目的[3-4], 基因芯片实验的研究目的包括如下三方面.
类别比较是指对一些类别已经明确的实验样本之间进行基因表达谱的比较.比如Hedenfalk et al[5]比较Brca1基因突变乳腺癌、Brca2基因突变乳腺癌以及没有上述基因突变的乳腺癌之间的差异基因表达谱.Golub et al[6]对急性淋巴细胞白血病和急性粒细胞白血病之间的基因表达差异.Ross et al[7]比较了来源于不同组织的癌细胞的差异表达等.人们通过这些实验主要想达到三个目的:一是这些不同种类样本之间是否存在差异基因表达谱, 二是哪些基因在不同种类样本之间存在差异表达;三是通过筛选基因的表达水平对不同样本进行判断, 从而降低误判率.
一些芯片研究是为了探测在基因表达谱和临床结果之间是否存在关系, 以便进一步研制基于基因表达谱基础上的预兆预报系统[8].例如一些药物遗传学研究企图知道那些患者在有效剂量内可能中毒等.
基因芯片研究的另一个目的就是类别找寻, 这是基于样本之间存在重要的生物学差异, 比如临床和形态上的相似可能在分子上获得区别[9].又如肿瘤通常以原发的器官而命名, 亚型是以细胞的类型进行分类.通常以形态学和组织学不能探测起源细胞.很多有关癌症的基因芯片研究目的就在于肿瘤的分类, 这些研究可能揭示疾病的生物特点, 通过鉴定治疗的分子靶标为改进疾病的治疗铺平道路.
单因子实验是指整个试验中只比较一个试验因子不同水平的试验.单因子试验方案由该试验因子的所有水平构成.基因芯片的单因子实验设计包括直接与间接比较, 所有的双色基因芯片检测都是成对比较, 比如治疗和非治疗之间、突变和野生型生物或者来源于不同组织的细胞之间的比较等.如图1, 假如我们想比较样本T和C的基因表达水平, 就可以在同一张基因芯片上进行比较.差异基因表达可以通过Log2(T/C)来计算, Log2T和Log2C的值来自样本T和C.由于他们来自于同一杂交, 我们称之为直接比较.另外Log2T和Log2C可以在2个杂交中获得, T和C的检测都通过与另一样本R的比较获得, Log2T/C值为Log2(T/R)-Log2(C/R)所代替.由于Log2T和Log2C值来自于2个杂交, 故称为间接比较. 具体可分为如下3类[10].
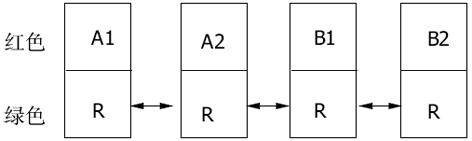
2.1.1 参照设计(common reference resign): 由于每一检测样本与参照样本配对杂交, 故样本量等于芯片数, 参照样品作为内参标准.检测样本标记为一种颜色, 参照样品标记为另一种颜色.如图2所示A组样本A1、A2和B组样本B1、B2都标记为红色, 对照品R标记为绿色.因为通常没有生物学意义的参照样品都在每张芯片测量, 故增加了实验的干扰降低了实验的灵敏性;但他的优点是利于任何分组样本的差异基因表达分析, 另外如果没有大的实验技术上的差异, 使用相同参照的不同实验理论上可以相互比较.如果将欲比较的一方样本混合后再与另一方各样本比较则称为混合样本的参照设计(pooled reference sample), 混合样本参照对于小量RNA样本的比较是有利的, 不同基因的表达量在样本混合后将起到平均的作用, 缺点是混合样本掩盖了生物的多样性.
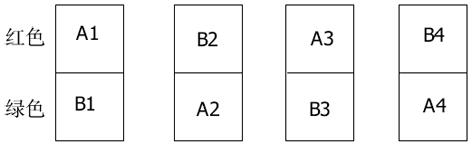
2.1.2 平衡区组设计(balanced block design): 平衡设计多用于二组样本之间的比较, 首先对两组样本进行任意配对, 用红色、绿色染料交替标记二组检测样本, 如图3, A1、A2、A3、A4分别标记为红色、绿色、红色、绿色.B1、B2、B3、B4则分别标记为绿色、红色、绿色、红色.芯片数目是参照设计的一半.其缺点是如果二组之间样本不相等或者比较的样本超过二组, 则必须进行复杂的修改.如果研究者想进行诸如聚类等分类, 芯片引入了人为的相关性因素可能会影响聚类分析的结果.以肿瘤和正常组织比较为例, 如果我们不考虑正常组织之间的差异, 则平衡设计是研究正常组织与肿瘤组织之间差异的好方法.
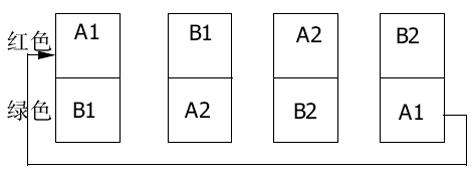
2.1.3 环形设计(loop design): 环形设计要求每一个样本都标记二种颜色(红色、绿色), 并分别与另外二个样本杂交(图4), 他要求和参照设计同样的芯片数目.如果芯片数目固定, 则环形设计就不如平衡设计效率高;但是如果只有二组样本时, 则比参照设计效率高.环形设计不适合于聚类分析, 而且由于实验技术的原因导致某些芯片数据的不可靠就会打断环形, 寻找合适的统计方法就变得非常困难, 所以一般尽量不选用环形设计.
参照设计、平衡设计和环形设计都能够提供客观的差异基因表达, 但是他们的效率不是一样的, 实验设计的有效率是与统计的要求的精度是相关的[11].实验设计的选择依赖于样本的数目和芯片的数量, 比如只能负担20张芯片, 应当如何设计芯片实验;如果只有12个样本, 又应当怎样设计芯片实验? Dobbin和Simon[12]对于三种实验设计进行了比较, 认为当基因芯片实验次数是固定时, 平衡设计比参照设计和环形设计更有效;当样本是有限时参照设计优于环形设计和平衡设计.
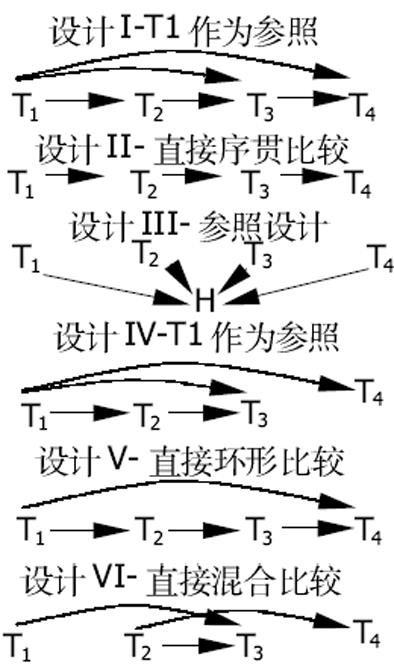
时间进程设计是以时间点的比较为基础, Yang和Speed[13]对单因素时间进程设计如图5所示:设计I使用T1作为共同参照, 设计II引入了连续时间点的杂交, 当研究目的是观察T1、T2、T3和T4之间的差异时, 设计I是较好的. 假如要更细微地观察一个时间点与另一个时间点的差异, 则设计II将更好;设计III是一个共同参照方法;设计IV相似设计I使用T1作为共同参照, 并额外增加了T2和T3之间的比较; 设计V是环形设计;设计VI是直接与间接的混合应用, 这比其他设计更精密.设计V与设计VI的选择取决于比较兴趣.如果对连续时间点的比较比2个时间点的比较更有兴趣, 则设计V更好.时间序贯的多因素的时间进程则应该参照多因素实验进行设计.
复因子实验设计是指在同一试验中同时研究两个或两个以上试验因素的试验.多因素试验方案由该试验的所有试验因素的水平组合构成.多因素试验方案分为完全方案和不完全方案两类.完全方案是在列出因素水平组合时, 要求每一个因素的每个水平都要碰见一次, 这时, 水平组合数等于各个因素水平数的乘积.由于基因芯片成本昂贵, 故完全方案是不现实的.不完全方案是将试验因素的某些水平组合在一起形成少数几个水平组合.这种试验方案的目的在于探讨试验因素中某些水平组合的综合作用, 而不在于考察试验因素对试验指标的影响和交互作用.在基因芯片实验中如何进行水平组合取决于研究目的, Glonek[14]和Townsend et al[15]鉴于芯片数量及mRNA量因素等原因, 探讨了以2×2析因设计为基础设计多因子实验.
为了消除实验技术等因素带来的误差, 一个通常的问题是芯片的重复实验是否是必要, 重复实验包括3层含义[16]:一是同一基因在同一芯片上多次布点重复;二是同一样品多次重复实验;三是多个样品多次实验.前二者是技术重复, 后者是生物重复.回答基因芯片实验是否重复、怎样重复这个问题, 首先必须了解导致基因芯片实验误差的因素.基因芯片实验的目的是解释生物问题, 因此选择来源于同一生物群体的不同样本进行实验是最好的方法, 即生物重复.技术重复主要是对同一样本进行多次杂交实验, 技术重复比生物重复包含更少的差异范围, 他在基因芯片实验的质量控制是必要的.技术重复是提供基因芯片重复性能的估计, 即通过同样的mRNA样本对基因芯片的标记、杂交和定量分析过程的重复性能的探索.技术重复可通过平均样本的表达量达到提高表达精度的测量要求.此外还有染料交叉重复, 染料交叉重复是指同样的二份RNA样本进行二次杂交, 即是二次杂交采取相反的染料标记.染料交叉重复对于纠正红色、绿色染料偏差是有意义的, 如想做单次芯片实验是可行的方法.技术重复可以通过计算平均值而提高测量的准确性, 多次的重复还可进行聚类分析.但是技术重复是不能代替生物重复的[17], 比如对1个群体中1个样本进行了100次技术重复实验与另一群体的1个样本的100次技术重复实验结果相比较.结论只能是2个生物样本而不是2个生物群体的异同.
混合样本是在标记杂交前, 将几个不同来源的RNA样本混合, 混合样本有二个目的, 一是单个样本没有足够的RNA量进行芯片实验, 二是为了降低研究成本而减少芯片实验的数目.研究者通过混合样本而减少杂交次数, 理由是通过混合可以达到平均每类样本的基因表达的目的.但是每类样本混合的实验设计是不适合于统计处理的, 因为混合样本没有考虑生物和技术的差异.即使进行多次重复实验能够减少实验差异, 但是也不能反应出生物学的差异.混合样本有如下一些缺点, 他没有考虑单个样本对基因表达的贡献;混合样本的平均基因表达可能与单个样本间的平均基因表达水平不同, 因为不同样本RNA的混合量不等, 或者混合后的样本改变了基因表达谱;用混合的资料是不易理解基因表达谱在群体中的分布规律的[18].当然, 当单个样本没有足够量的RNA进行基因芯片时混合样本还是有用的.
由于样本量的大小直接影响差异基因和非差异基因的可信度, 究竟需要多少样本量是基因芯片实验经常面临的问题[19].一般情况下, 样本量的多少取决于基因表达变化的幅度、统计精确度的要求、I型误差率以及不同的统计方法等.一般说来实验误差越小, 则I型误差率p(α)、II型误差率p(β)也越小, 所以通过不同的试验设计提高实验的准确度是首要的措施[20].当然样本越大时, 则I型误差率、II型误差率也越小, 但基因芯片实验是昂贵的, 因此根据研究目的不同, 以适合做变量分析(统计要求)确定最小样本量就显得尤为重要.Hwang et al[21]人以公式i个基因的总平方和公式一, (1是若干单个基因的矩阵);组内变量为, 公式二, Wi, 为单个基因在组内的平方和;组间Bi(某确定基因i在两组间平均表达的总平方和)Bi = Ti-Wi;向量Xi(N×1)为某i基因在N个样本中的表达水平, xi是i基因在N个样本中的平均表达值, 上标j代表总的C个类中的第j类;用下列公式进行F检验公式三.他们选择72个白血病样本的基因表达谱资料, 其中47个样本为急性淋巴细胞白血病(ALL)样本(38个为B细胞淋巴细胞白血病, 9个为T细胞淋巴细胞白血病), 25个为急性骨髓白血病(AML)样本.经上式显著性检验得出:当I型误差率p(α)和II型误差率p(β)取0.05时, 每一组样本至少需要7个样本, 但为了获得更为准确的结果, 实验设计通常选择50个以上的样本.
总之, 基因芯片的实验设计以研究目的为基础, 以统计学的因素水平为原则, 同时应注意生物学特点.
编辑: 张海宁
| 1. | Foster WR, Huber RM. Current themes in microarray experimental design and analysis. Drug Discov Today. 2002;7:290-292. [PubMed] [DOI] |
| 2. | Churchill GA. Fundamentals of experimental design for cDNA microarrays. Nat Genet. 2002;32:490-495. [PubMed] [DOI] |
| 3. | Pichler FB, Black MA, Williams LC, Love DR. Design, normalization, and analysis of spotted microarray data. Methods Cell Biol. 2004;77:521-543. [PubMed] [DOI] |
| 4. | Murphy D. Gene expression studies using microarrays: principles, problems, and prospects. Adv Physiol Educ. 2002;26:256-270. [PubMed] [DOI] |
| 5. | Hedenfalk I, Duggan D, Chen Y, Radmacher M, Bittner M, Simon R, Meltzer P, Gusterson B, Esteller M, Kallioniemi OP. Gene-expression profiles in hereditary breast cancer. N Engl J Med. 2001;344:539-548. [PubMed] [DOI] |
| 6. | Golub TR, Slonim DK, Tamayo P, Huard C, Gaasenbeek M, Mesirov JP, Coller H, Loh ML, Downing JR, Caligiuri MA. Molecular classification of cancer: class discovery and class prediction by gene expression monitoring. Science. 1999;286:531-537. [PubMed] [DOI] |
| 7. | Ross DT, Scherf U, Eisen MB, Perou CM, Rees C, Spellman P, Iyer V, Jeffrey SS, Van de Rijn M, Waltham M. Systematic variation in gene expression patterns in human cancer cell lines. Nat Genet. 2000;24:227-235. [PubMed] [DOI] |
| 8. | Lorenz MG, Cortes LM, Lorenz JJ, Liu ET. Strategy for the design of custom cDNA microarrays. Biotechniques. 2003;34:1264-1270. [PubMed] |
| 9. | Simon R, Radmacher MD, Dobbin K. Design of studies using DNA microarrays. Genet Epidemiol. 2002;23:21-36. [PubMed] [DOI] |
| 10. | Draghici S, Kuklin A, Hoff B, Shams S. Experimental design, analysis of variance and slide quality assessment in gene expression arrays. Curr Opin Drug Discov Devel. 2001;4:332-337. [PubMed] |
| 11. | Kerr MK, Churchill GA. Experimental design for gene expression microarrays. Biostatistics. 2001;2:183-201. [PubMed] [DOI] |
| 12. | Dobbin K, Simon R. Comparison of microarray designs for class comparison and class discovery. Bioinformatics. 2002;18:1438-1445. [PubMed] [DOI] |
| 13. | Yang YH, Speed T. Design issues for cDNA microarray experiments. Nat Rev Genet. 2002;3:579-588. [PubMed] |
| 14. | Glonek GF, Solomon PJ. Factorial and time course designs for cDNA microarray experiments. Biostatistics. 2004;5:89-111. [PubMed] [DOI] |
| 15. | Townsend JP. Multifactorial experimental design and the transitivity of ratios with spotted DNA microarrays. BMC Genomics. 2003;4:41. [PubMed] [DOI] |
| 16. | Kerr MK. Design considerations for efficient and effective microarray studies. Biometrics. 2003;59:822-828. [PubMed] [DOI] |
| 17. | McShane LM, Shih JH, Michalowska AM. Statistical issues in the design and analysis of gene expression microarray studies of animal models. J Mammary Gland Biol Neoplasia. 2003;8:359-374. [PubMed] [DOI] |
| 18. | Kendziorski CM, Zhang Y, Lan H, Attie AD. The efficiency of pooling mRNA in microarray experiments. Biostatistics. 2003;4:465-477. [PubMed] [DOI] |
| 19. | Yang MC, Yang JJ, McIndoe RA, She JX. Microarray experimental design: power and sample size considerations. Physiol Genomics. 2003;16:24-28. [PubMed] [DOI] |
| 20. | Mace J, Sybil Biermann J, Sondak V, McGinn C, Hayes C, Thomas D, Baker L. Response of extraabdominal desmoid tumors to therapy with imatinib mesylate. Cancer. 2002;95:2373-2379. [PubMed] [DOI] |
| 21. | Hwang D, Schmitt WA, Stephanopoulos G, Stephanopoulos G. Determination of minimum sample size and discriminatory expression patterns in microarray data. Bioinformatics. 2002;18:1184-1193. [PubMed] [DOI] |