修回日期: 2004-09-17
接受日期: 2004-10-18
在线出版日期: 2005-01-01
目的: 应用抑制性消减杂交技术构建人类新基因NS5ATP2(615)反式激活基因差异表达的cDNA消减文库, 克隆NS5ATP2反式激活相关基因, 了解该基因的可能生物学功能.
方法: 构建NS5ATP2表达质粒pcDNA3.1(-)NS5ATP2-TP2转染HepG2细胞, 以空载体pcDNA3.1(-)转染的HepG2细胞为对照; 提取转染后细胞的mRNA, 反转录为cDNA. 半定量RT-PCR显示实验组NS5ATP2的转录水平明显高于对照组. cDNA经RsaI酶切后, 将实验组cDNA分成两组, 分别与两种不同的接头衔接, 再与对照组cDNA进行两次消减杂交及两次抑制多聚酶链反应(PCR), 将产物与pEGM-Teasy载体连接, 构建cDNA消减文库, 并转染大肠杆菌进行文库扩增, 随机挑选克隆PCR扩增后进行测序及同源性分析.
结果: 成功构建人类新基因NS5ATP2反式激活基因差异表达的cDNA消减文库. 文库扩增后得到76个白色克隆, 进行菌落PCR分析, 均得到200-1000 bp插入片段. 挑取含有插入片段的32个克隆进行测序, 并通过生物信息学分析获得17种已知功能基因序列, 和2个未知功能基因.
结论: 应用SSH技术成功构建了NS5ATP2反式激活基因差异表达的cDNA消减文库. 该文库的建立为阐明NS5ATP2生物学功能提供理论依据.
引文著录: 杨倩, 成军, 刘妍, 洪源, 王琳, 董菁, 张树林. 应用抑制性消减杂交技术克隆NS5A-TP2(615)反式激活基因. 世界华人消化杂志 2005; 13(1): 82-85
Revised: September 17, 2004
Accepted: October 18, 2004
Published online: January 1, 2005
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2005; 13(1): 82-85
- URL: https://www.wjgnet.com/1009-3079/full/v13/i1/82.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v13.i1.82
HCV NS5A对细胞周期及细胞生长具有调节作用, 可能与慢性肝炎、肝细胞癌的发生发展有着密切的关系[1-3]. 我室对NS5A反式激活基因进行克隆化研究, 发现了NS5A蛋白的上调一些基因的表达. 其中包括未知功能基因, 命名为NS5ATP2[4]. 利用生物信息学技术确定其开放读码框架(ORF), 并对其进行了克隆化研究, 在克隆化研究过程中我们不仅顺利得到了NS5ATP2基因编码序列, 同时意外得到了NS5ATP2的不同剪接体, 将其命名为NS5ATP2(512). 为探究新基因的功能提供新的依据[5]. 为了解其细胞内功能, 我们利用抑制性消减杂交技术构建NS5ATP2作用于肝母细胞瘤细胞系HepG2细胞后反式调节的cDNA消减文库, 筛选差异表达的基因片段, 并应用生物信息学进行分析, 为了解NS5ATP2的功能、进一步探讨HCV发病及致癌机制提供新的研究方向.
肝母细胞瘤细胞系HepG2细胞及大肠杆菌DH5α(本室保存), pcDNA3.1(-)真核表达载体(Invitrogen), FuGENE6转染试剂(Roche), QuikPreP mico mRNA Purification试剂盒, PCR-Select cDNA Subtraction试剂盒(Clontech), 50×PCR Enzyme Mix、Advantage PCR Cloning试剂盒(Clontech), High Pure PCR Product Purification试剂盒(Promega), pGEM-Teasy载体(Promega)T7、SP6通用引物(上海博亚公司合成). 真核表达质粒pcDNA3.1(-)-NS5ATP2(615)由本室构建. DNA序列测定由上海申友公司完成.
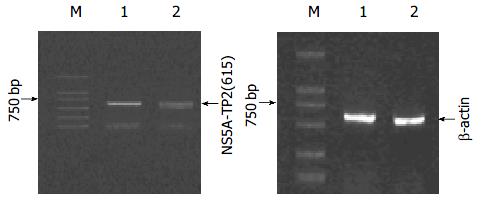
(1)半定量RT-PCR检测pcDNA3.1(-)-NS5A TP2(615)转录水平: 根据NS5A TP2(615)的ORF设计上下游引物, 上游引物5'-GGA TTC ATG GCT TCG GTC TCC TCT GC-3'下游引物5'-GGT ACC TCA GGA GTG TGG CTC ACT GG-3'以看家基因β-actin为内参. 将经分光光度计定量分析的mRNA分别稀释为0.5 g/L, 各取0.5 μL进行反转录, 以反转录产物为模板, 进行PCR.
(2)真核表达载体的细胞转染及mRNA提取: 用FuGENE6转染试剂将2 μg pcDNA3.1(-)-NS5A TP2(615)及pcDNA3.1(-)空载体分别转染35 mm平皿HepG2细胞, 48 h后收获细胞. 使用QuikPreP mico mRNA Purification试剂盒从HepG2细胞中直接提取重组表达质粒及空载体的HepG2细胞的mRNA, 经分光光度计分别进行定量分析.
(3)杂交文库的建立: 采用PCR-Select cDNA Subtraction Kit, 常规SSH方法按说明书进行: 以转染了重组表达质粒及空载体的HepG2细胞mRNA为模板逆转录合成双链cDNA(dscDNA), 并分别标记为Tester和Driver, dscDNA经RsaⅠ(一种识别4碱基序列的内切酶)消化, 产生相对较短的平端片段, 纯化酶切产物. 将Tester的dscDNA分为两份, 分别连接试剂盒提供的特殊设计的寡核苷酸接头Adapter 1和Adapter 2, 然后与过量的Driver dscDNA进行杂交; 合并两种杂交产物后再与Driver dscDNA作第2次杂交; 然后将杂交产物做选择性PCR扩增, 使Tester dscDNA中特异性表达或高表达的片段得到特异性扩增.
(4)消减文库扩增及克隆分析: 扩增产物与pGEM-Teasy载体连接, 转化DH5α感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养18 h. 挑取白色菌落, 增菌, 以pGEM-Teasy载体多克隆位点两端T7/SP6引物进行菌落PCR扩增, 证明含有插入片段后(200-1000 bp), 测序. 应用生物信息学将测得序列与GenBank数据库进行在线同源性分析(http://www.ncbi.nlm.gov/blast/blast.cgi).
紫外分光检测显示, 转染了真核表达质粒及空载体的HepG2细胞提取mRNA分别为2.1 μg和3.3 μg, A260/A280 = 1.79. 2.0 g/L琼脂糖凝胶电泳mRNA为大于0.5 kb清晰慧尾片状条带, 证实mRNA质量完全满足进行消减杂交的要求.
半定量RT-PCR产物9 g/L琼脂糖凝胶电泳显示: 转染了质粒pcDNA3.1(-)-NS5A TP2(615)的细胞中NS5A TP2(615)转录水平明显高于转染空载体的x细胞, 以空载体mRNA为模板的RT-PCR产物凝胶电泳图中可以看见NS5A TP2(615)的可变见解体NS5A TP2(512)的表达(图1).
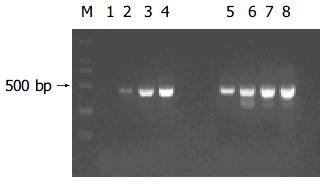
以消减及未消减PCR产物为模板, 用G3PDH引物进行PCR扩增, 分别在18、23、28、33次循环结束时从体系中吸取5 μL进行电泳鉴定. 结果显示: 与未消减组PCR产物相比, 消减组PCR产物中G3PDH基因产物大大减少, 说明所构建的消减文库具有很高的消减效率(图2).
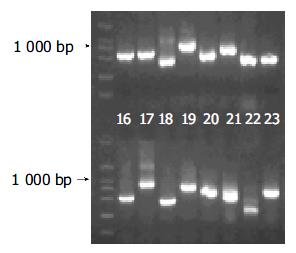
杂交产物经两轮PCR扩增后, 菌落PCR扩增结果显示为200-1000 bp大小不等的插入片段, 所获得的76个克隆中几乎均含有插入片段, 这些条带可能代表差异表达的基因片段(图3).
挑选30个克隆测序, 与GenBank数据库进行初步比较. 其中17个克隆均与已知基因的部分序列高度同源(96%-100%), 2个克隆未发现与已知功能基因高度同源, 部分结果(表1).
| 已知功能基因 | 克隆数 | 同源性 |
| 纤连蛋白(fibronectin) | 2 | 99% |
| 脂肪酸辅酶A连接酶(fatty-acid-Coenzyme A ligase) | 1 | 98% |
| 过量表达的胸壁肿瘤蛋白(over-expressed breast tumor protein, OBTP) | 3 | 98% |
| NADH脱氢酶(NADH dehydrogenase) | 2 | 98% |
| 脱氢酶dihydrodiol dehydrogenase | 1 | 99% |
| 肺癌癌基因(lung cancer oncogene 1) | 1 | 97% |
| 真核翻译延伸因子1(eukaryotic translation elongation factor 1) | 2 | 99% |
| 核糖体蛋白L27(ribosomal protein L27) | 2 | 100% |
| 核糖体蛋白S6(ribosomal protein S6) | 1 | 99% |
丙型肝炎病毒的NS5A蛋白是一种具有广泛反式激活作用的蛋白质分子, 其羧基末端富含酸性氨基酸及脯氨酸, 这是真核细胞转录因子特有的结构特征. 一般来说, 转录的反式激活因子是在细胞核中起作用的, 而NS5A主要定位于细胞内质网(ER), 因此推测NS5A可能参与了细胞信号传导途径[6]. NS5A的生物学作用还包括对细胞周期及细胞生长的调节[7-10]. 我室利用抑制性消减杂交技术[11], 对于NS5A表达载体转染的肝母细胞瘤细胞系HepG2的基因表达谱变化进行比较研究, 发现了NS5A蛋白可以反式激活一些基因的表达. 其中包括未知功能基因, 命名为NS5ATP2. 我们对其进行了克隆化研究, 成功获得了NS5ATP2的编码序列, 其开放读码框架ORF区为615个核苷酸, 编码205个氨基酸, 其氨基酸序列"masvssatfsghga rsllqflrlvgqlkrvprtgwvyrnvqrpesvsdhmyrm avmamvik ddrlnkdrcvrlalvhdmaecivgdiapareeeamkqitdnipkeekhr qllpedlrkelyelweeyetqssaeakfvkqldqcemilqaseyedle hkpgrlqdfy dstagkfnhpeivqlvseleaerstniaaaasephs". 在此过程中我们意外发现了NS5ATP2基因的不同剪切体, 对NS5ATP2基因组进行分析, 确定了该基因剪切体的编码序列, 并进行了克隆化研究. 并将其命名为NS5ATP2(512), 其ORF为216个核苷酸, 编码72个氨基酸. 为进一步对新基因功能进行研究我们建立了NS5ATP2抑制性消减杂交文库, 对反式激活哪些基因进行了克隆化研究. 挑选文库中30个克隆送测序进行鉴定, 结果发现NS5ATP2可上调一些与细胞代谢、肿瘤发生、肝纤维化有关的基因. 例如: 过量表达的胸壁肿瘤蛋白, 虽然该基因的功能并不明确, 但研究表明在胸壁肿瘤组织中该基因表达明显升高[12]; 肺癌癌基因1亦与肿瘤的发生密切相关[13]. 纤连蛋白在肝纤维化、肝硬化的发生发展中起者重要的作用[14-17], NS5ATP2上调该基因的表达是否与丙型肝炎肝硬化有关有待进一步研究. 在筛选的文库基因中还发现与肝细胞胆汁代谢有关的脂肪酸辅酶A, 研究表明脂肪酸辅酶A与类固醇激素代谢有关[18-19].
在对NS5ATP2研究的同时我们运用同样的方法对其可变剪接体NS5ATP2(512)的功能进行了研究. 与NS5ATP2(512)抑制性消减文库结果相比, 我们发现二者均可上调一些与肿瘤发生相关的基因. 人类基因组可能有约35000个基因, 生物的复杂性与其基因组基因数量似乎存在明显差异. 原因在蛋白质组. 基因重排, RNA编辑:, 和可变剪接等机制可以从一个基因产生多种蛋白, 从而使蛋白质组中蛋白质的数量超过基因组中基因的数量[20-22]. 其中, 从影响的基因数量和生物种类范围来看, 可变剪接是扩大蛋白质多样性的最重要的机制. 单独一个基因通过可变剪接产生的十几种剪接异构体的现象很常见[23-25]. 有些基因甚至能够产生成千上万种剪接异构体一个基因通过可变剪接产生多个转录异构体, 各个不同的转录异构体编码结构和功能不同的蛋白质, 他们分别在细胞/个体分化发育不同阶段, 在不同的组织, 有各自特异的表达和功能[26-27]. 因此, 可变剪接是一种在转录后RNA水平调控基因表达的重要机制. 我们利用抑制性消减杂交技术对NS5ATP2和其可变剪接体NS5ATP2(512)功能学进行了初步研究, 两者功能上到底是相互依赖还是相互抑制有待进一步探索.
编辑: 潘伯荣
| 2. | Girard S, Vossman E, Misek DE, Podevin P, Hanash S, Bréchot C, Beretta L. Hepatitis C virus NS5A-regulated gene expression and signaling revealed via microarray and comparative promoter analyses. Hepatology. 2004;40:708-718. [PubMed] |
| 3. | Franco S, Giménez-Barcons M, Puig-Basagoiti F, Furcic I, Sánchez-Tapias JM, Rodés J, Sáiz JC. Characterization and evolution of NS5A quasispecies of hepatitis C virus genotype 1b in patients with different stages of liver disease. J Med Virol. 2003;71:195-204. [PubMed] |
| 5. | 杨 倩, 刘 妍, 成 军, 王 建军, 王 春花, 纪 冬, 党 晓燕. 丙型肝炎病毒非结构蛋白NS5A反式激活基因2基因组DNA结构分析及其不同剪切体的克隆化研究. 世界华人消化杂志. 2004;12:801-804. [DOI] |
| 6. | Street A, Macdonald A, Crowder K, Harris M. The Hepatitis C virus NS5A protein activates a phosphoinositide 3-kinase-dependent survival signaling cascade. J Biol Chem. 2004;279:12232-12241. [PubMed] |
| 7. | Sarcar B, Ghosh AK, Steele R, Ray R, Ray RB. Hepatitis C virus NS5A mediated STAT3 activation requires co-operation of Jak1 kinase. Virology. 2004;322:51-60. [PubMed] |
| 8. | Macdonald A, Crowder K, Street A, McCormick C, Harris M. The hepatitis C virus NS5A protein binds to members of the Src family of tyrosine kinases and regulates kinase activity. J Gen Virol. 2004;85:721-729. [PubMed] |
| 9. | Qadri I, Iwahashi M, Simon F. Hepatitis C virus NS5A protein binds TBP and p53, inhibiting their DNA binding and p53 interactions with TBP and ERCC3. Biochim Biophys Acta. 2002;1592:193-204. [PubMed] |
| 10. | Goh PY, Tan YJ, Lim SP, Lim SG, Tan YH, Hong WJ. The hepatitis C virus core protein interacts with NS5A and activates its caspase-mediated proteolytic cleavage. Virology. 2001;290:224-236. [PubMed] |
| 11. | Ji W, Wright MB, Cai L, Flament A, Lindpaintner K. Efficacy of SSH PCR in isolating differentially expressed genes. BMC Genomics. 2002;3:12. [PubMed] |
| 12. | Wu I, Moses MA. Cloning of a cDNA encoding an isoform of human protein phosphatase inhibitor 2 from vascularized breast tumor. DNA Seq. 2001;11:515-518. [PubMed] |
| 13. | Weber HC, Walters J, Leyton J, Casibang M, Purdom S, Jensen RT, Coy DH, Ellis C, Clark G, Moody TW. A bombesin receptor subtype-3 peptide increases nuclear oncogene expression in a MEK-1 dependent manner in human lung cancer cells. Eur J Pharmacol. 2001;412:13-20. [PubMed] |
| 14. | Schlaf G, Schmitz M, Heine I, Demberg T, Schieferdecker HL, Götze O. Upregulation of fibronectin but not of entactin, collagen IV and smooth muscle actin by anaphylatoxin C5a in rat hepatic stellate cells. Histol Histopathol. 2004;19:1165-1174. [PubMed] |
| 15. | Astrof S, Crowley D, George EL, Fukuda T, Sekiguchi K, Hanahan D, Hynes RO. Direct test of potential roles of EIIIA and EIIIB alternatively spliced segments of fibronectin in physiological and tumor angiogenesis. Mol Cell Biol. 2004;24:8662-8670. [PubMed] |
| 16. | Svegliati-Baroni G, Ridolfi F, Di Sario A, Saccomanno S, Bendia E, Benedetti A, Greenwel P. Intracellular signaling pathways involved in acetaldehyde-induced collagen and fibronectin gene expression in human hepatic stellate cells. Hepatology. 2001;33:1130-1140. [PubMed] |
| 17. | Kato R, Kamiya S, Ueki M, Yajima H, Ishii T, Nakamura H, Katayama T, Fukai F. The fibronectin-derived antiadhesive peptides suppress the myofibroblastic conversion of rat hepatic stellate cells. Exp Cell Res. 2001;265:54-63. [PubMed] |
| 18. | Sung YK, Hwang SY, Park MK, Farooq M, Han IS, Bae HI, Kim JC, Kim M. Glypican-3 is overexpressed in human hepatocellular carcinoma. Cancer Sci. 2003;94:259-262. [PubMed] |
| 19. | Richardson AJ, Easton T, Puri BK. Red cell and plasma fatty acid changes accompanying symptom remission in a patient with schizophrenia treated with eicosapentaenoic acid. Eur Neuropsychopharmacol. 2000;10:189-193. [PubMed] |
| 20. | Zolfaghari R, Ross AC. Cloning, gene organization and identification of an alternative splicing process in lecithin: retinol acyltransferase cDNA from human liver. Gene. 2004;341:181-188. [PubMed] |
| 21. | Ghosh A, Stewart D, Matlashewski G. Regulation of human p53 activity and cell localization by alternative splicing. Mol Cell Biol. 2004;24:7987-7997. [PubMed] |
| 22. | Akgul C, Moulding DA, Edwards SW. Alternative splicing of Bcl-2-related genes: functional consequences and potential therapeutic applications. Cell Mol Life Sci. 2004;61:2189-2199. [PubMed] |
| 23. | Ellington WR, Yamashita D, Suzuki T. Alternative splicing produces transcripts coding for alpha and beta chains of a hetero-dimeric phosphagen kinase. Gene. 2004;334:167-174. [PubMed] |
| 24. | Resch A, Xing Y, Modrek B, Gorlick M, Riley R, Lee C. Assessing the impact of alternative splicing on domain interactions in the human proteome. J Proteome Res. 2004;3:76-83. [PubMed] |
| 25. | Wagner EJ, Baines A, Albrecht T, Brazas RM, Garcia-Blanco MA. Imaging alternative splicing in living cells. Methods Mol Biol. 2004;257:29-46. [PubMed] |
| 26. | Sorek R, Shamir R, Ast G. How prevalent is functional alternative splicing in the human genome? Trends Genet. 2004;20:68-71. [PubMed] |