修回日期: 2004-06-09
接受日期: 2004-06-17
在线出版日期: 2004-09-15
目的: 探讨乙型肝炎病毒(HBV)基因型的分型方式, 并根据病毒蛋白结构提出新的病毒蛋白分型方式.
方法: 自GenBank中按基因型搜索符合要求的HBV基因组序列, 并应用Vector NTI suite 8.0版软件进行基因组核苷酸及各基因编码蛋白质序列比较, 并利用软件分析前前-S基因、前-X基因和前-C基因的存在状态.
结果: 在GenBank中根据HBV基因型分型搜索出119个病毒株全基因组, 比较后发现选择病毒株基因组核苷酸序列总阳性率和总一致率分别为95.7%和47.7%; 选择病毒株编码的全C蛋白、全S蛋白、全X蛋白和多聚酶的总阳性率分别为98.6%、87.3%、57.2%和95.2%, 总一致率分别为37.4%、24.1%、27.7%和43.5%. 在病毒群中, 33.61%的病毒株编码前前-S多肽, 14.3%的病毒株编码前-X多肽, 26.1%的病毒株不编码前-C多肽, 94.1%编码前-X多肽的病毒株同时编码前前-S多肽. 基因组1-700 nt一致率30.6%, 1 103-1 653 nt一致率20.8%, 为高变区; 基因组1 654-1 950 nt的一致率为74.2%, 为高保守区.4种病毒蛋白各有其相应的高变区和高保守区. 根据病毒蛋白前导性序列的变异情况提出新的分型方法, 命名为蛋白表型.蛋白表型分7型, IV型为主要流行表型, 占39.5%, V型和VII型各占19. 3%. 亚洲HBV蛋白分型分布分散, I、IV、V和VII型所占比例均大于20%; 欧洲IV型占58.3%, VII型占25.0%, V型占13.9%.
结论: 在综合分析HBV基因组的基础上, 初步划分出HBV基因组和病毒蛋白内部存在的高变区和高保守区. 提出蛋白表型的新概念, 并综合展示基因核苷酸突变所导致的病毒蛋白的结构差异.
引文著录: 董菁, 任建林, 卢雅丕. 乙型肝炎病毒蛋白表型定义初探. 世界华人消化杂志 2004; 12(9): 2074-2085
Revised: June 9, 2004
Accepted: June 17, 2004
Published online: September 15, 2004
AIM: To create a new typing method showing the difference among HBV strains after reviewing the HBV genome sequences labeled with different genotypes in the GenBank.
METHODS: HBV genome sequences were collected from the GenBank and then classified into 8 groups based on their genotypes labeled by authors. The Vector NTI suite 8.0 software was used to compare the identity and difference among the strains of HBV genomes. Possible regions encoding pre-pre-S, pre-X and pre-C peptides were also analyzed with this software.
RESULTS: One hundred and nineteen full-length HBV genomes from GenBank were collected, and then sorted into 8 groups according to their genotypes. The total positive rate and total identical rate of 119 sequences were 95.7% and 47.7%, respectively. The total positive rates of whole C protein, whole S protein, whole X protein and polymerase amino acids sequences were 98.6%, 87.3%, 57.2% and 95.2%, respectively; and total identical rates were 37.4%,24.1%, 27.7% and 43.5%. In the study group, 33.61% strains encoded pre-pre-S peptide, 14.3% strains encoded pre-X peptide, 26.05% strains had no function of encoding pre-C peptide, whereas, 94.1% of pre-X coding strains also encoded pre-pre-S peptide. The identical rates of region 1-700 nt and 1 103-1 653 nt of HBV genome were 30.6% and 20.8%, respectively, and therefore they were considered as hypervariable region; the identical rate of region 1 654-1 950 nt of HBV genome was 74.2% and defined as hyperconversable region. Hypervariable and hypterconversable regions could be found in all of the four viral proteins. Based on mutations of leading peptides of the three HBV viral proteins, a novel typing method named protype was therefore generated. In this new category, 7 protypes were listed, and there were 39.5% strains belonging to the major one, type IV, type V and type both covering 19.3%. All 7 protypes were found in Asia, with the percentages of I, IV, V and VII types above 20%. There was no stain isolated from Europe belonging to protypes I, II, or III, and the percentages of IV was 58.3%, V 13.9% and VII 25.0%, respectively.
CONCLUSION: Hypervariable and hypterconversable regions are noticed while analyzing HBV genome sequences. Furthermore, protype, a novel term is raised to elucidate encoding of the 3 leading peptides and structural variation of viral proteins due to gene mutation.
- Citation: Dong J, Ren JL, Lu YP. Definition of protype of hepatitis B virus: A preliminary study. Shijie Huaren Xiaohua Zazhi 2004; 12(9): 2074-2085
- URL: https://www.wjgnet.com/1009-3079/full/v12/i9/2074.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i9.2074
1968年发现了乙型肝炎病毒(HBV)的抗原, 1972年Le Bouvier et al[1]提出HBV表面抗原(HBsAg)根据血清反应的不同而分为不同亚型, 即提出血清型分型概念.Galibert et al[2]于1979年第1次解读了HBV ayw血清型基因组的核苷酸序列, 长度为3 182 nt. 1988年Okamoto et al[3]首次提出HBV基因型的概念, 即根据各基因组之间差异大于8%而人为的将病毒群划分为不同的基因型, 1990年Norder et al[4]基于多聚酶链反应(PCR)方法建立了简便的基因型别分析方法, 之后学者在研究中不断提出存在新的基因型.目前将HBV分为8种基因型, 分别为A、B、C、D、E、F、G、H型[5], 基因型分布具有一定的地理特征, A型主要分布在北欧、西欧和北美, B和C型流行于东亚和远东, D型分布广泛, 在地中海、印度、近东和中东地区多见, E型流行于西撒哈拉地区, F型主要在美洲大陆流行, G型主要在美国[6], H型在中美洲流行[7]. 国内主要的基因型为B、C两型[8-9], 台湾学者报告提示除E型外, 其他基因型均可在华人HBV感染者中被检出, B和C型占患者人群的85%[10]. 我们早期的研究认为不同的HBV病毒株的4个开放读码框架(ORF)分区中存在差异[11], 部分病毒分离株的S基因在原有的前S1区之前, 存在有前前-S区编码前前-S多肽[12]; 而X基因之前可能存在前-X区, 编码前-X多肽[13]. 在本组早期的研究中认为前前-S区和前-X区的存在可能具有基因型特异性, 提出应当对中国HBV流行株的结构与功能复杂性进行重新认识[14-15], 本研究分析了目前存储在GenBank中的不同基因型HBV病毒株基因组, 探讨了一种新的HBV分型方法.
应用生物信息学技术进行研究, 利用的材料为GenBank中存储的HBV全基因组序列.
1.2.1 病毒株的选择: 进入美国国立卫生院(NIH)网站, 在GenBank中搜寻HBV基因组序列, 之后进一步限定基因型分型分别为A, B, C, D, E, F, G, H型, 将搜寻结果下载以进一步分析, 其中也包括本研究组以往报告[16-17]的全基因组序列. 所筛选出的序列首先检查其病毒蛋白序列的完整性, 部分病毒株虽然标明基因型别, 但其序列不能编码一种或一种以上HBV病毒蛋白, 或序列中含有少量测定不准确的核苷酸位点(r或n等), 这些病毒株序列被排除在本研究之外.
1.2.2 核苷酸序列分析: 应用Vector 8. 0版软件对下载的存储于GenBank中不同基因型的HBV基因组序列进行比较. 比较前将原存储于GenBank中的HBV基因组序列进行了起始点的统一计数处理, 即与Gunther et al[18]和董菁 et al[19]文献中HBV基因组序列的排列方式一致, 各序列均以5- TTT TTC ACC TCT GC-3为开始, 保证了各序列之间的可比较性.
1.2.3 编码病毒蛋白氨基酸序列分析: 将各病毒株基因组报告者所公布的P基因和前-C/C基因编码产物, 即多聚酶和HBeAg氨基酸序列收集后, 应用Vector 8.0版软件进行比较序列之间的一致率. 利用Vector 8.0版软件具有的ORF判读功能, 重新判读X、S基因, 判断各病毒株是否编码前-X区[13,20]和前前-S区[12,21]. 将表达前-C多肽和核心蛋白的病毒蛋白命名为全C蛋白; 如病毒株存在前-X基因和前前-S基因序列, 将核酸序列进行翻译后获得的前-X多肽和前前-S多肽与原X蛋白和原S蛋白命名为全X蛋白和全S蛋白, 将来自不同基因型的HBV全X蛋白(原X蛋白)和全S蛋白(原S蛋白)进行比较. Vector 8.0版软件将所有选择出的HBV序列进行比较后, 提供以下重要数据: 一致性序列, 是软件自动比较所有序列, 参考每个对应的核苷酸/蛋白质位点上不同克隆的编码/表达方式, 由软件形成最具代表性的一致性序列; 如果某位点出现3种以上的编码/表达方式, 一致性序列中提示为空缺, 表明了该位点的核苷酸/氨基酸多样性. 阳性率是选定区域一致性序列的核苷酸/氨基酸序列数目与区域核苷酸/蛋白质序列总长度之比, 提示差异位点的比例, 用于展示区域内部的插入突变/缺失突变, 以及单一位点多种核苷酸/氨基酸替换突变所占比例. 一致率是选定区域全部克隆均为一致的核苷酸/氨基酸数目与最长的单一克隆核苷酸/氨基酸序列数目之比, 表示该段区域核苷酸/氨基酸序列一致性, 用于展示区域内部的替换突变或/和缺失突变.如插入突变的克隆不能占据简单多数, 则阳性率和一致率均为0%. Vector 8.0版软件比较核苷酸/氨基酸可推导出系统发生树, 以分支树形式表示分子之间的进化关系, 以及遗传关系的远近.
经过在GenBank中按不同基因型进行搜寻, 分别获得的基因型A型病毒株12株, B型16株, C型48株, D型13株, E型4株, F型26株, G型12株, H型3株, 共134株HBV病毒株全基因组序列. 按照方法中的排除条件, 剔除部分不适合本研究的病毒株序列, 共有119株HBV病毒株全基因组核苷酸序列本引入到本研究中, 选取率为88.8%. 选用的序列中基因型A型12株, B型11株, C型44株, D型13株, E型3株, F型21株, G型12株, H型3株. 其中C型的44株病毒株包括本组以往的研究[15-16]获得的5个HBV全基因组克隆, 分别命名为China Dong 1 C, 2 C, 3 C, 6 C和7 C.在本文中选择的病毒株的编号为地点+顺序号+基因型别, 如Japan 12 C, Japan是该序列来源国家, 12为本研究组在GenBank搜寻过程中所定义的顺序号, C为基因型别, 按照此方法, 所有参加比较的序列各有其独立的标记. 本研究组既往研究中获得的序列在国家后加Dong以示区别.基因型别按照报告单位的地理分布如下: A型: 加拿大1株, 法国4株, 南非7株; B型: 中国3株, 日本2株, 荷兰1株, 南非1株, 瑞典4株; C型: 澳大利亚5株, 中国17株, 日本13株, 瑞典9株; D型: 西班牙1株, 法国2株, 日本5株, 瑞典5株; E型: 加纳1株, 日本2株; F型: 阿根廷4株, 瑞典6株, 委内瑞拉11株; G型: 德国1株, 日本11株; H型: 瑞典3株.
HBV基因组多态性表现为长度的明显差异, 所研究的119株病毒株中, 长度最长为3 254 nt, 最短为2 996 nt; 62株病毒株的基因组全长为3 215 nt, 占研究总数的52.1%(表1). 16株病毒株的基因组全长为3 182 nt, 占13.5%. HBV基因组长度具有一定的基因型特异性, 在GenBank中搜寻获得的12株G型HBV基因组序列中, 有7株病毒株的长度为3 248 nt, 该长度是G基因型的一个重要特征; 长度为3 221 nt的6个病毒株中, A型占5株; 长度为3 212 nt的3个病毒株均为E型. 所研究的119株病毒株中有22种长度形式, 其中12种长度形式仅有1株病毒株(10.1%, 12/119).
| HBV不同基因组 | 3215 nt | 3182 nt | 长度范围(nt) | 阳性率(%) | 一致率(%) |
| A型(13株) | 0 | 0 | 3 149-3 254 | 98.8 | 85.3 |
| B型(11株) | 9 | 0 | 3 194-3 221 | 99.7 | 88.5 |
| C型(41株) | 31 | 4 | 2 996-3 215 | 99.7 | 70.4 |
| D型(16株) | 0 | 12 | 3 182-3 194 | 99.5 | 86.4 |
| E型(3株) | 0 | 0 | 3 212 | 99.9 | 96.8 |
| F型(22株) | 18 | 0 | 3 129-3 215 | 100.0 | 84.0 |
| G型(9株) | 1 | 0 | 3 089-3 248 | 100.0 | 93.2 |
| H型(4株) | 3 | 0 | 3 206-3 215 | 100.0 | 95.9 |
所有119株HBV基因组序列比较后, 推导出的系统发生树. 经过比较后, 可以得出以下几点结论: (1)除C基因型病毒株之外, 各基因型的HBV均表现为相对独立的分支, C型在各基因型中显得较为古老; (2)虽然在存储HBV基因组序列时, 各作者进行了基因型的判断, 但经过全基因组序列分析, 发现部分分型方法并不正确, 如Australian 3 C, 4 C和5 C应当属于D基因型, Japan 1 G属于F基因型, Japan 11 G属于H基因型, Japan 2 G属于A基因型. 因此将上述型别重新划分到各基因型组中, 进行比较分析.
在119个基因型序列的HBV基因组比较后, 基因组的跨度总长为3 303 nt, 这是由于不同型别之间在不同区域存在插入突变, 导致基因组序列长于最长的3 254 nt. 比较后总阳性率为95.7%, 说明有140 nt(4.3%, 140/3 303)为插入或缺失突变; 总一致性仅为47.7%(表2), 说明一半以上的位点存在多态性表现.
| 阳性率(%) | 一致率(%) | |
| 1-700 | 88.1 | 30.6 |
| 701-1 102 | 96.3 | 53.1 |
| 1 103-1 653 | 97.1 | 20.8 |
| 1 654-1 950 | 99.3 | 74.2 |
| 1 951-3 303 | 98.1 | 59.0 |
| 总计 | 95.7 | 47.7 |
应用Vector 8.0版软件ORF判定功能, 对HBV基因组蛋白编码区域进行分析, 发现40株序列编码前前-S多肽, 占33.6%, 分别来自C, F和H基因型(Japan11G划归H基因型, 下同). 17株序列编码前-X多肽, 占14.3%, 均来自C基因型, 4株来自日本病毒株, 13株来自中国病毒株; 其中16株病毒序列编码前前-S多肽, 占94.1%(16/17). 31株病毒序列不编码前-C多肽, 占26.1%, 除了E, F基因型外, 各基因型均有病毒株不编码前-C多肽.根据HBV基因组编码病毒蛋白结构的不同, 将HBV分为7种蛋白表型, 分型方式见表3. 根据这种分型方式, 我们分析的119株病毒基因组中, I型: 共10株, 均来自C基因型, 占8.40%; II型: China Dong 6C, 7C, China 14C, 共3株, 占2.5%;III型: China 1C, 仅1株, 占0.8%;IV型: 共47株, 占39.5%, 为主要的流行蛋白表型, 来自除G, H基因型以外的所有基因型;V型, 23株, 占19.3%, 来自C, D, F和H基因型;VI型: Australian 2C, Japan 4C, Sweden 1C, Japan 11G(H型), 共4株, 占3.4%;VII型: 23株, 占19.3%, 来自A, B, C, D和G基因型, 其中G基因型前-C多肽的表达具有其独特的特异性, 长度小于传统定义的前-C区29 aa长度的多肽, 仅长12 aa; 有8个病毒株不能完整表达一种或一种以上HBV病毒蛋白, 表达序列长度小于预计长度的50%, 占6.7%, 无法进行蛋白表型分型, 属于缺陷型病毒.我们提出的HBV蛋白表型分型方法, 以IV型为主要流行表型, 占39.5%, 为Galibertet al[2]最早解读的HBV基因组序列编码方式;V型和VII型各占19.3%, 是重要的流行型别. 前-X多肽多与前前-S多肽联动表达, 仅1例例外. I, II, III型均来自C基因型, I型以China Dong 1 C为代表, 占8.4%.
| 全S蛋白 | 全X蛋白 | 全前C-C蛋白 | 多聚酶 | |||||
| 阳性率(%) | 一致率(%) | 阳性率(%) | 一致率(%) | 阳性率(%) | 一致率(%) | 阳性率(%) | 一致率(%) | |
| A型 (13株) | 99.8 | 75.6 | 74.8 | 61.7 | 99.5 | 44.7 | 99.8 | 58.4 |
| B型 (11株) | 100.0 | 81.5 | 96.8 | 82.7 | 99.5 | 77.4 | 99.8 | 85.5 |
| C型 (41株) | 99.6 | 49.9 | 65.8 | 47.0 | 100.0 | 67.5 | 99.8 | 71.6 |
| D型 (16株) | 98.7 | 85.3 | 100.0 | 83.8 | 99.5 | 69.8 | 99.4 | 85.5 |
| E型 (3株) | 100.0 | 96.0 | 100.0 | 94.2 | 100.0 | 98.6 | 100.0 | 95.2 |
| F型 (22株) | 89.9 | 80.0 | 100.0 | 82.5 | 100.0 | 76.9 | 100.0 | 84.9 |
| G型 (9株) | 100.0 | 87.7 | 100.0 | 96.8 | 100.0 | 85.1 | 100.0 | 91.9 |
| H型 (4株) | 100.0 | 96.9 | 100.0 | 89.6 | 100.0 | 84.0 | 100.0 | 95.1 |
| 总计 | 87.3 | 24.1 | 57.2 | 27.7 | 98.6 | 37.4 | 95.2 | 43.5 |
病毒蛋白的表达除部分位点上表现出型特异性特征外, 也表现出区域性的多态性, 113株不同基因型HBV编码的HBeAg总阳性率为98.6%, 总一致率仅为37.4%, 其大部分区域存在明显的氨基酸位点多态性(见表4).分区的比较发现: 前-C区编码的29 aa, 31个序列不编码前-C多肽, 故其一致率为0%; 30-61aa和130-173aa为HbeAg的2个高保守区, 其阳性率均为100%, 无明显的插入/缺失突变; 这2段区域的一致率明显高于总一致率. 相应的, 62-129 aa和174-214 aa为HBeAg的2段高变区, 一致率较总一致率分别低10.9%和10.3%. 118株不同基因型HBV编码的全S基因编码产物包含前前-S区, 前-S1, 前-S2和主蛋白(下同), 总阳性率仅为87.3%, 总一致率仅为24.1%, 说明存在较多的缺失/插入突变(表4). 进一步分析发现存在2个高度变异区和1个高度保守区, 以往定义的前-S1和前-S2区为高变区, 46-187 aa区域的阳性率为95.8%, 一致率仅为7.7%; 410-448 aa一致率为0, 说明2个区域的缺失/插入突变较多.
| HbeAg | 全S蛋白 | ||||
| 阳性率 | 一致率 | 阳性率 | 一致率 | ||
| 1-29 | 100.0 | 0.0 | 1-45 | 0.0 | 0.0 |
| 30-61 | 100.0 | 65.6 | 46-187 | 95.8 | 7.7 |
| 62-129 | 98.5 | 26.5 | 188-409 | 97.7 | 43.7 |
| 130-173 | 100.0 | 70.5 | 410-448 | 97.4 | 0.0 |
| 174-214 | 95.1 | 24.4 | |||
| 总计 | 98.6 | 37.4 | 总计 | 87.3 | 24.1 |
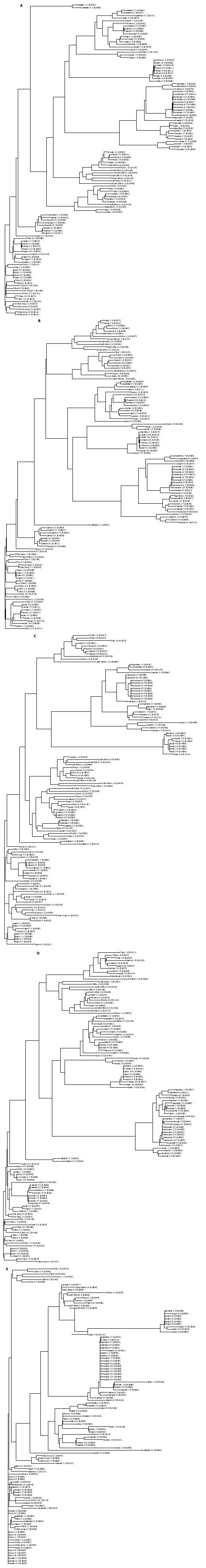
118株不同基因型HBV编码的全X蛋白包括: 前-X和原X蛋白, 总阳性率仅为57.2%, 总一致率仅为27.7%, 说明存在较多的缺失/插入突变. 进一步分析发现存在1个高度变异区和1个高度保守区. 17株C基因型序列编码前-X多肽, 该段的阳性率和一致率为0%; 57-133 aa区域的阳性率为97.4%, 一致率低为54.5%, 高于总一致率1倍; 134-264 aa一致率为23.7%, 说明区域内存在较多的缺失/插入突变. 117株不同基因型HBV编码的多聚酶总阳性率仅为95.2%, 总一致率为43.5%, 其一致率为4个病毒蛋白中最高的, 与其编码区域内不包括表达前导区有关. 进一步分析发现存在2个高度变异区和1个高度保守区, 180-369 aa区域的阳性率为87.4%, 一致率低为12.6%; 465-514 aa一致率为20.0%, 说明2个区域的缺失/插入突变较多.而370-464 aa区域的阳性率为99.0%, 一致率为76.8%, 远高于平均的43.5%. 上述4种病毒蛋白按基因型进行比较后, 获得的阳性率和一致率见表3. 将不同基因型病毒株编码的病毒蛋白进行比较, 所获得的系统发生树见图1A-E.
HBV的基因组是最早被解读的病原体基因完整信息, 我们探讨过HBV准种现象[22-24], 观察了HBsAg基因多态性与蛋白质多态性之间的关系[25], HBV血清型仅反映表面抗原的变化, 不足以反映高度变异的病毒准种群核酸序列差异与病毒蛋白氨基酸序列差异之间的关系, 现试图探讨一种以病毒蛋白差异为主要分型标准的HBV分型方式, 暂命名为HBV蛋白表型(protype).在GenBank中, 我们初步搜寻出200多株HBV全基因组序列, 之后再次限定作者标定的HBV基因型, 自A至H进行再次筛选, 选择出的病毒株按照不同的基因型分组以备进一步分析. 将筛选出的HBV基因组序列进行初步分析, 凡是序列内部包含有测序结果不精确的病毒株均被排除在本研究之外. 经过上述选择过程, 共筛选出119株HBV全基因组序列, 长度最长为3 254 nt, 最短为2 996 nt. HBV多态性表现在长度的不一致性, 共有22种长度形式, 52.1%的病毒株基因组长度为3 215 nt, 为主要流行长度; 13.5%的病毒株基因组全长为3 182 nt; 其中12种长度形式仅有1株病毒株.基因组长度具有一定的基因型特异性, 经过调整后, G基因型9例病毒株中, 7例长度为3 248 nt, 经比较发现在91 nt之后有一段长26 nt的插入序列, 为5'TAGAACAACTTTGCCATATGGCCTTTTTGGC TTAGA-3'G基因型特异性序列.该段序列与上游5 nt共同编码前C区MDRTTLPYGLFGL, 成为缩水的前-C区, 替代了其他基因型中的前-C区编码的29 aa和HBcAg的第一位M. 该编码现象是G基因型的一个重要蛋白分子特征.
基因型的分型原则是根据全基因核苷酸序列之间比较后所获得的一致性进行分类的, 至此, 目前报告了8种基因型[3, 4, 6, 7, 26]. 我们自GenBank中搜索出的119个作者进行基因型分型的基因组病毒株中, 调整后A型13株, B型11株, C型41株, D型16株, E型3株, F型22株, G型9株, H型4株, 其中以C型最多, 占34.5%;其次为F型, 占18.5%. 曾有学者[7,26]认为基因型的分布具有明显的地理特性, 但本研究119株病毒株的地理分布广泛, 欧洲36株, 亚洲53株, 非洲9株, 美洲16株, 大洋洲5株.除报告较少的E型和H型, HBV基因型的地理分布没有一定的规律, 可能与目前世界范围交通发达, 人员流动大有关, 但在与外界交流很少的民族中可能存在少见的HBV基因型. 我们针对119例不同基因型的HBV基因组进行了全序列比较, 同时对其编码的不同的病毒蛋白进行了比较分析, 结果发现: (1)除全C蛋白氨基酸系统发生树外, 基因组系统发生树与全S蛋白、多聚酶和全X蛋白的氨基酸系统发生树结构相似, 而在全C蛋白的氨基酸系统发生树分析过程中, 发现部分B基因型病毒株与C基因型的全C蛋白的系统发生特征相近. 从上述图形结构的比较而言, 可以认为限定区域内核苷酸的变异导致的氨基酸序列的变异程度是不均一的, 两种分子的进化步骤具有非同步性; (3)无论基因组核苷酸系统发生树还是4种病毒蛋白的系统发生树, 各基因型的HBV病毒株均划归于相对独立的分支, 表现出较高的遗传特征, 唯有C型各病毒株的遗传关系显得较为松散, 其系统发生树的地位在各基因型中显得较为古老; (3)结合系统发生树与基因型内病毒株序列一致性的研究结果发现: 只有C基因型各病毒株之间的总一致率低为70. 4%(表1), 其他基因型的总一致率接近或高于84.0%.研究同时发现随着分型内部病毒株的数量增加, 其一致率出现明显下降, C型一致率的水平已低于以往定义的基因型的分型标准, 按目前标准定义的C基因型的病毒株总体的遗传特征不明确, 有必要提出新的分型标准来表示HBV的变异特征.
我们进一步分析了HBV不同基因型的节段差异性, 发现119株不同基因型HBV病毒株基因组序列总一致率为47.7%.区域1-700 nt一致率30.6%, 区域1 103-1 653 nt一致率20.8%, 较总一致率低10-20%, 为高变区;区域1 654-1 950 nt的一致率为74.2%, 可定为高保守区. 我们是在计算机软件分析的基础上, 人工的最小化或最大化各区域的一致率, 具体流程见文献[18], 但划分出来的高变区和高保守区与文献[18]不同, 这可能与选择的基因型与血清型不同有关.本文还进一步确定了各病毒蛋白的高变区和高保守区, 113株全C蛋白多态性的比较结果提示62-129 aa和174-214 aa一致率分别为26.5%和24.4%;30-61 aa和130-173 aa一致率分别为65.6%和70.5%, 较总一致率37.4%有明显区别.118例全S蛋白内多态性的比较结果提示46-187 aa和410-448 aa一致率分别为7.7%和0.0%;188-409 aa一致率43.7%, 较总一致率24.1%有明显区别.118例全X蛋白多态性的比较结果提示57-133 aa一致率54.5%, 明显高于总一致率的27.7%; 117例多聚酶多态性的比较结果提示180-369 aa和465-514 aa一致率分别为12.6%和20.0%; 370-464 aa一致率76.8 %, 较总一致率43.5%有明显区别, 这些数据说明各病毒蛋白内部均有各自特异的高变区和高保守区.全S蛋白总一致率仅24.1%, 其前S1、前S2区一致率小于10%, 目前疫苗的靶区域188-409 aa一致率也仅43.7%, 这提示目前疫苗的覆盖性较差, 需要进一步研制高代表性HBV疫苗.
我们着重研究了HBV 4个ORF的结构特征, 并提出根据病毒蛋白不同的结构特征进行分型的新概念, 目前暂时将这种型别命名为HBV的蛋白表型. 在分型方法的建立过程中, 我们着重强调了前前-S多肽、前-X多肽和前-C多肽的重要性, 这是由于这3段蛋白序列的编码与否在不同病毒株基因组的表现形式是不一样的, 前前-S多肽长度为45 aa, 占全S蛋白全长的10.1%(45/446), 编码前前-S多肽阳性的病毒株占研究总数的33.6%, 分别属于C, F和H基因型; 前-X多肽长度为56 aa, 占全X蛋白全长的26.7%(56/210), 编码前-X多肽阳性的病毒株占研究总数的14.3%, 均属于C基因型;前-C多肽长度29 aa, 占前C-C蛋白全长的13.7%(29/212), 编码前-C多肽阴性的病毒株占研究总数的26.1%, 分别属于A, B, C, D, F和H基因型, G型的9株病毒株的前-C区编码多肽形式特别. 我们发现编码前前-S多肽和前-X多肽在病毒基因组中属于相对少见的情形, 而编码前-C多肽在病毒基因组中属于较常见的现象. 我们以往关于前-C区与C区基因相互关系的研究提出前-C区编码与HBeAg的生物合成有关, 前-C区变异[29]或核心蛋白启动子(CP)的变异[30]可导致HBeAg阴性慢性乙型肝炎(CHB);前-X多肽的编码与原发性肝癌(HCC)的发生有关[31], 但前前-S多肽的功能尚不明了. 蛋白表型的分型方法是确定的, 这与基因型分型日益增多的情形大相径庭, 由于基因型表现出明显的地理特性, 随着研究的深入, 其分型结果会日益增多, 由于蛋白表型的分型方式固定, 将简化分型方式, 为以后的研究设立一个便于比较的平台.
蛋白表型分型重点强调由于基因突变所导致原定义的HBV表面抗原、X蛋白、核心蛋白的前导性序列以及病毒蛋白结构的变化, 同时也强调了病毒蛋白前导性序列的重要性. 按照以往的概念, HBV表面抗原、核心蛋白为病毒的结构蛋白, 其前导性序列可能与病毒蛋白合成后的细胞定位有关; X蛋白的功能尚不明了, 可能与病毒基因调节有关, 作为一种反式激活因子, 定位是在细胞核内.蛋白表型分型强调了基于HBV病毒蛋白基本情况之上的变异方式, 显示核苷酸变异对病毒蛋白编码所产生的重要影响. 117株病毒株的多聚酶的总一致率为43.5%, 高于全S蛋白、全X蛋白和前C-C蛋白的总一致率24.1%, 27.7%和37.4%, 这证实前导性序列对病毒蛋白一致性的影响. 按照我们提议的新的分型方法分析本研究搜集的119株病毒株, 发现为Galibert et al[2]最早解读的HBV基因组序列, 即IV型主要流行表型, 共47例, 占分析总数的39.5%. IV型的蛋白表型特点是表达前-C多肽, 而不表达前前-S多肽和前-X多肽, 除G, H基因型外, 其他基因型病毒株均可见IV型的分布. V型和VII型各有23例, 占分析总数的19.3%, 是重要的流行型别, 前者的特点是表达前前-S多肽和前-C多肽, 但不表达前-X多肽, 后者的特点是三种前导性序列均不表达. II型, III型和VI型是少见的蛋白表型, 其中III型仅1例, 其存在与否需要进一步证实. 由于HBV基因存在高变异现象, 1993年有学者提出HBV准种学说[32-33], 我们的研究[15]证实HBV在患者体内以准种群形式存在, 这种学说同时带来一种研究技术上的要求, 即以单独克隆的HBV基因组测序结果为准, 而不应当是以往研究[34-36]中以直接的PCR结果进行测序来表示全基因组情况, 因为PCR-直接测序方法不能排除准种群导致的位点混杂情况. 有鉴于此, II型、III型和VI型的病毒株测序方法存有一定误差, 其可靠性需要进一步验证, 因此按照本文提出的分型方法, 分型的型别可能将有所减少. 新的分型结果发现有8株HBV基因组序列不能编码完整的1种或1种以上的病毒蛋白, 推断其不具有独立完成生活史的能力, 即需要依靠其他HBV病毒株生存的缺陷型病毒, 我们[37]以往的研究也发现这种现象, 按照本方法无法进行分型, 因此设立无法分型的型别为缺陷型病毒特异性型别. 49株亚洲HBV病毒分离株的蛋白表型分型结果提示: I, IV, V和VII型HBV病毒株所占比例均大于20%, 7种型别均出现于亚洲;36株欧洲HBV病毒分离株的蛋白表型分型结果提示: IV型占58.3%, VII型占25.0%, V型占13.9%, 说明以IV型为主要流行型别, VII型也占重要地位. I, II, III型不出现于欧洲, 在亚洲这三种类型的总和占总数的28. 6%, 这3种类型的共同点是HBV基因组编码前-X多肽, 这3种蛋白表型HBV在亚洲的流行是否是亚洲原发性肝癌发病率高于欧洲的一个原因, 还需要进一步研究.
总之, 我们应用生物信息学技术对GenBank中存储的119个不同基因型HBV病毒株全基因组进行了比较分析, 在此基础上我们提出HBV蛋白表型假说, 蛋白表型着重强调了S基因、X基因、C基因前导性序列的重要性, 通过分型将局部核苷酸替换突变与蛋白结构差异结合起来, 我们试图建立一种区别于现有基因型分型方式, 以展示病毒蛋白结构差异为主的分型方式, 这种分型方式的现实意义需要进一步探讨.
| 1. | Le Bouvier GL, McCollum RW, Hierholzer WJ Jr, Irwin GR, Krugman S, Giles JP. Subtypes of Australia antigen and hepatitis-B virus. JAMA. 1972;222:928-930. [PubMed] [DOI] |
| 2. | Galibert F, Mandart E, Fitoussi F, Tiollais P, Charnay P. Nucleotide sequence of the hepatitis B virus genome (subtype ayw) cloned in E. coli. Nature. 1979;281:646-650. [PubMed] [DOI] |
| 3. | Okamoto H, Tsuda F, Sakugawa H, Sastrosoewignjo RI, Imai M, Miyakawa Y, Mayumi M. Typing hepatitis B virus by homology in nucleotide sequence: comparison of surface antigen subtypes. J Gen Virol. 1988;69:2575-2583. [PubMed] [DOI] |
| 4. | Norder H, Hammas B, Magnius LO. Typing of hepatitis B virus genomes by a simplified polymerase chain reaction. J Med Virol. 1990;31:215-221. [PubMed] [DOI] |
| 5. | Kao JH, Chen PJ, Lai MY, Chen DS. Hepatitis B genotypes correlate with clinical outcomes in patients with chronic hepatitis B. Gastroenterology. 2000;118:554-559. [PubMed] [DOI] |
| 6. | Stuyver L, De Gendt S, Van Geyt C, Zoulim F, Fried M, Schinazi RF, Rossau R. A new genotype of hepatitis B virus: complete genome and phylogenetic relatedness. J Gen Virol. 2000;81:67-74. [PubMed] [DOI] |
| 7. | Arauz-Ruiz P, Norder H, Robertson BH, Magnius LO. Genotype H: a new Amerindian genotype of hepatitis B virus revealed in Central America. J Gen Virol. 2002;83:2059-2073. [PubMed] [DOI] |
| 10. | Sugauchi F, Orito E, Ichida T, Kato H, Sakugawa H, Kakumu S, Ishida T, Chutaputti A, Lai CL, Gish RG. Epidemiologic and virologic characteristics of hepatitis B virus genotype B having the recombination with genotype C. Gastroenterology. 2003;124:925-932. [PubMed] [DOI] |
| 14. | 成 军, 董 菁, 洪 源, 刘 妍, 钟 彦伟, 王 琳, 王 刚, 张 玲霞, 陈 菊梅. 乙型肝炎病毒中国流行株全基因的克隆与序列分析. 世界华人消化杂志. 2003;11:1119-1126. [DOI] |
| 16. | 董 菁, 成 军, 王 勤环, 皇甫 竞坤, 施 双双, 张 国庆, 洪 源, 李 莉, 斯 崇文. 慢性乙型肝炎患者体内乙型肝炎病毒DNA序列异质性及准种特点的研究. 中华医学杂志. 2002;82:81-85. |
| 18. | Günther S, Li BC, Miska S, Krüger DH, Meisel H, Will H. A novel method for efficient amplification of whole hepatitis B virus genomes permits rapid functional analysis and reveals deletion mutants in immunosuppressed patients. J Virol. 1995;69:5437-5444. [PubMed] |
| 20. | 杨 倩, 董 菁, 成 军, 刘 妍, 洪 源, 王 建军, 王 琳, 张 树林. 乙型肝炎病毒基因组中前-X-编码基因启动子序列的确定及转录活性的鉴定. 解放军医学杂志. 2003;28:763-765. |
| 21. | 杨 倩, 董 菁, 成 军, 刘 妍, 洪 源, 王 建军, 张 树林. 乙型肝炎病毒基因组中前-前-S编码基因启动子序列的确定及转录活性的鉴定. 解放军医学杂志. 2003;28:761-762. |
| 24. | 董 菁, 成 军, 王 勤环, 施 双双, 洪 源, 皇甫 竞坤, 王 刚, 李 莉, 斯 崇文. 乙型肝炎病毒逆转录酶区基因序列准种与变异研究. 解放军医学杂志. 2001;26:823-825. |
| 25. | 董 菁, 刘 妍, 皇甫 竞坤, 施 双双, 王 刚, 洪 源, 陈 国凤, 李 莉, 陈 菊梅, 成 军. 乙型肝炎病毒表面抗原一级结构多态性的初步研究. 胃肠病学和肝病学杂志. 2002;11:130-135. |
| 26. | Norder H, Hammas B, Lee SD, Bile K, Couroucé AM, Mushahwar IK, Magnius LO. Genetic relatedness of hepatitis B viral strains of diverse geographical origin and natural variations in the primary structure of the surface antigen. J Gen Virol. 1993;74:1341-1348. [PubMed] [DOI] |
| 29. | 董 菁, 施 双双, 皇甫 竞坤, 成 军, 王 勤环, 王 刚, 洪 源, 李 莉, 斯 崇文. 乙型肝炎病毒前C/C基因准种与变异特点的研究. 解放军医学杂志. 2002;27:122-124. |
| 30. | 董 菁, 施 双双, 张 国庆, 皇甫 竞坤, 洪 源, 成 军, 王 勤环, 李 莉, 斯 崇文. 乙型肝炎病毒C基因启动子区异质性检测初步研究. 临床检验杂志. 2002;20:72-74. |
| 31. | Takahashi K, Akahane Y, Hino K, Ohta Y, Mishiro S. Hepatitis B virus genomic sequence in the circulation of hepatocellular carcinoma patients: comparative analysis of 40 full-length isolates. Arch Virol. 1998;143:2313-2326. [PubMed] [DOI] |
| 32. | Blum HE. Hepatitis B virus: significance of naturally occurring mutants. Intervirology. 1993;35:40-50. [PubMed] [DOI] |
| 33. | Carman W, Thomas H, Domingo E. Viral genetic variation: hepatitis B virus as a clinical example. Lancet. 1993;341:349-353. [PubMed] [DOI] |
| 34. | Hannoun C, Horal P, Lindh M. Long-term mutation rates in the hepatitis B virus genome. J Gen Virol. 2000;81:75-83. [PubMed] [DOI] |
| 35. | Kramvis A, Weitzmann L, Owiredu WK, Kew MC. Analysis of the complete genome of subgroup A' hepatitis B virus isolates from South Africa. J Gen Virol. 2002;83:835-839. [PubMed] [DOI] |
| 36. | Sugauchi F, Mizokami M, Orito E, Ohno T, Kato H, Suzuki S, Kimura Y, Ueda R, Butterworth LA, Cooksley WG. A novel variant genotype C of hepatitis B virus identified in isolates from Australian Aborigines: complete genome sequence and phylogenetic relatedness. J Gen Virol. 2001;82:883-892. [PubMed] [DOI] |