修回日期: 2004-03-01
接受日期: 2004-05-11
在线出版日期: 2004-07-15
目的: 应用抑制性消减杂交(SSH)技术构建HBV DNA聚合酶(DNA P)末端蛋白反式激活基因.
方法: 以RNase H表达质粒pcDNA3.1(-)-RNase H转染HepG2细胞, 以空载体pcDNA3.1(-)为对照; 制备转染后的细胞裂解液, 提取mRNA并逆转录为cDNA, 经RsaI酶切后, 将实验组cDNA分成两组, 分别与两种不同的接头衔接, 再与对照组cDNA进行两次消减杂交及两次抑制性PCR, 将产物与T/A载体连接, 构建cDNA消减文库, 并转染大肠杆菌进行文库扩增, 随机挑选克隆PCR扩增后进行测序及同源性分析.
结果: 文库扩增后得到38个白色克隆, 经菌落PCR分析, 得到36个200-1 000 bp插入片段. 对所得片段测序, 并进行同源性分析, 显示33种已知基因编码蛋白和3种未知功能基因序列, 可能是RNase H反式激活靶基因.
结论: 成功构建HBV RNase H反式激活基因差异表达的cDNA消减文库.
引文著录: 王春花, 郎振为, 成军, 吴煜, 杨艳杰, 张黎颖, 党晓燕. 应用抑制性消减杂交技术筛选HBV DNA聚合酶中RNase H的反式调节基因. 世界华人消化杂志 2004; 12(7): 1564-1568
Revised: March 1, 2004
Accepted: May 11, 2004
Published online: July 15, 2004
AIM: To construct a subtractive cDNA library of genes transactivated by RNase H protein of hepatitis B virus (HBV) DNA polymerase with suppression subtractive hybridization (SSH) technique.
METHODS: The mRNA was isolated from HepG2 cells transfected with pcDNA3.1(-)-RNase H and pcDNA3.1(-) empty vector, respectively, and then cDNA was synthesized. After restriction enzyme RsaI digestion, small sizes cDNAs were obtained. Tester cDNA was divided into two groups and ligated to the specific adaptor 1 and adaptor 2, respectively. After tester cDNA was hybridized with driver cDNA twice and underwent two times of nested PCR and then was subcloned into T/A plasmid vectors to set up a subtractive library. Amplification of the library was carried out with E. coli strain JM109. The cDNA was sequenced and analyzed in GenBank with Blast search after PCR.
RESULTS: The amplified library contained 38 positive clones. Colony PCR showed that 36 clones contained 200-1 000 bp inserts. Sequence analysis suggested that 33 kinds of known and three kinds of novel cDNA sequences were the target genes transactivated by RNase H protein of HBV DNA P.
CONCLUSION: The subtractive library of genes transac-tivated by RNase H protein is successfully constructed.
- Citation: Wang CH, Lang ZW, Cheng J, Wu Y, Yang YJ, Zhang LY, Dang XY. Suppression subtractive hybridization for cloning of genes transactivated by RNase H protein of HBV DNA polymerase. Shijie Huaren Xiaohua Zazhi 2004; 12(7): 1564-1568
- URL: https://www.wjgnet.com/1009-3079/full/v12/i7/1564.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i7.1564
乙型肝炎病毒(HBV)是嗜肝DNA病毒的一种, HBV DNA的长度为3.2 kb, 具有4个开放读码框架(ORF), 分别编码HBV的表面抗原蛋白, 核心/e抗原蛋白、X蛋白以及HBV DNA聚合酶(HBV DNA P)[1]. 目前, HBV与肝细胞之间相互作用的分子生物学机制尚不完全清楚, 其中, 病毒编码的蛋白通过各种级联反应能够影响多种细胞信号转导途径, 从而进一步影响肝细胞某些基因的表达、调控, 可能是病毒感染导致肝细胞损伤和肝细胞癌发生、发展的重要原因[2-3]. HBV DNA P基因在ORF中最长, 并且与C, S, X基因区有重叠, 其编码的P蛋白含有3个功能域和1个无意义的隔离片(SP), 排列顺序为N-末端蛋白, 隔离片, RT/DNA聚合酶和核糖核酸酶. N-末端蛋白和隔离片对嗜肝病毒聚合酶是独特的, 逆转录酶(RT)和核糖核酸酶(RNase H)包含两个已知的酶活性位点, 与其他相关的逆转录病毒和逆转录因子的聚合酶是一致的[4-15]. Kim et al[16] 对人HBV聚合酶的N-末端或C-末端和结构域同时删除形成变异株, 并将变异株和野毒株一起在大肠杆菌中表达, 经直链淀粉柱层析法纯化后, 二者纯化蛋白的DNA依赖性的DNA聚合酶活性进行比较, 证明N-末端蛋白或隔离片删除分别可以使酶活性减少到70%, 而RNase H删除对聚合酶活性的影响要大于前二者. Walton et al[17]认为RNase H在HBV复制过程中的作用表现为能识别并有效裂开DNA-RNA杂交的RNA链, 但是缺乏序列选择性. 既往的研究多集中于RNase H 参与病毒复制的作用机制, 而RNase H蛋白在肝细胞中的表达对于肝细胞的基因表达谱的影响研究较少, 而这种反式激活作用也是病毒型肝炎、肝细胞癌(HCC)发病机制的主要机制
抑制性消减杂交技术(suppression subtractive hybri-dization, SSH)是1990年代后期建立的一种基因克隆的新技术, 可以快速有效地检测到差异表达的基因[18]. 本实验应用SSH技术, 构建RNase H作用于肝母细胞瘤细胞系HepG2细胞后反式调节的cDNA消减文库, 筛选差异表达的基因片段, 并应用生物信息学(bioinformatics)技术对所得片段进行序列同源性分析, 获得其全长基因序列. RNase H反式激活相关靶基因的克隆, 为研究HBV的致病(癌)机制提供新的理论依据.
肝母细胞瘤细胞系HepG2细胞及大肠杆菌JM109(本室保存), pcDNA3.1(-)真核表达载体(Invitrogen), Lipofectamine Plus 转染试剂(Gibco), mRNA Purification试剂盒(Amersham Pharmacia Biotech), PCR-Select cDNA Subtraction试剂盒, 50×PCR Enzyme Mix、Advantage PCR Cloning试剂盒(Clontech), High Pure PCR Product Purification试剂盒(Boehringer Mannheim), T7、SP6通用引物及pGEM-Teasy载体(Promega). RNase H真核表达质粒pcDNA3.1(-)- RNase H由本室构建. DNA序列测定由上海博亚公司完成.
用Lipofectamine Plus 转染试剂将2 g pcDNA3.1(-)-RNase H及pcDNA3.1(-)空载体分别转染35 mm平皿HepG2细胞, 48 h后收获细胞. 使用mRNA Purification试剂盒, 直接提取转染了重组表达质粒及空载体的HepG2细胞mRNA, 经琼脂糖凝胶电泳及分光光度计进行定性、定量分析. 采用PCR-Select cDNA Subtraction Kit, 常规SSH方法按说明书进行: 以转染了重组表达质粒及空载体的HepG2 细胞mRNA为模板逆转录合成双链cDNA(dscDNA), 并分别标记为Tester和Driver, dscDNA经RsaⅠ(一种识别4碱基序列的内切酶)消化, 产生相对较短的平端片段, 纯化酶切产物. 将Tester的cDNA分为两份, 分别连接试剂盒提供的特殊设计的寡核苷酸接头Adapter 1和Adapter 2, 然后与过量的Driver cDNA进行杂交; 合并两种杂交产物后再与Driver cDNA作第2次杂交; 然后将杂交产物做选择性PCR扩增, 使Tester cDNA中特异性表达或高表达的片段得到特异性扩增.
1.2.1 消减文库扩增及克隆分析: 扩增产物与pGEM-Teasy载体连接, 转化JM109感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养18 h. 挑取白色菌落, 增菌, 以pGEM-Teasy载体多克隆位点两端T7/SP6引物进行菌落PCR扩增, 证明含有插入片段后(200-800 bp), 测序. 应用生物信息学将测得序列与GenBank数据库进行同源性分析, 通过序列同源性搜索比对和电子拼接, 根据基因起始密码子的Kozak规则和终止密码子下游保守的多聚腺苷酸信号序列, 确定完整的编码基因序列.
使用高质量的mRNA是保证cDNA高产量的前提. 紫外分光检测显示, 转染了真核表达质粒及空载体的HepG2 细胞mRNA分别为5.12 g和4.92 g, A260/A280 = 1.25.1.0%琼脂糖凝胶电泳见 mRNA为大于0.5 kb清晰慧尾片状条带.
dscDNA与接头连接效率的高低是决定SSH成败的最关键步骤. 将连接有adaptor l和adaptor 2的两组dscDNA分别用不同的特异性引物(看家基因甘油三磷酸脱氢酶G3PDH引物)进行28个循环扩增, 产物用2.0%琼脂糖凝胶电泳鉴定. 结果显示两组dscDNA扩增产物浓度相当, 说明dscDNA已与接头高效率连接.
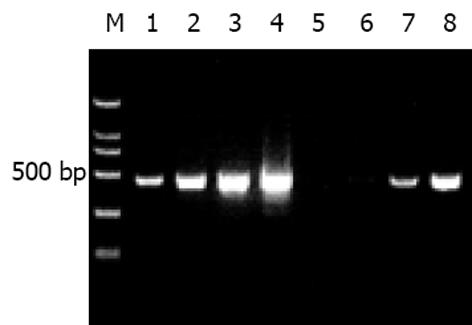
分别以消减及未消减PCR产物为模板, 用G3PDH引物进行PCR扩增, 分别在18, 23, 28, 33次循环结束时从体系中吸取5 L进行电泳鉴定.结果显示, 与未消减组PCR产物相比, 消减组PCR产物中G3PDH基因产物大大减少, 说明所构建的消减文库具有很高的消减效率(图1).
杂交产物经两轮PCR扩增后, 菌落PCR扩增结果显示为200-1 000 bp大小不等的插入片段, 所获得的38个克隆中几乎均含有插入片段, 这些条带可能代表差异表达的基因片段(图2).
挑选36个克隆测序, 与GenBank数据库进行初步比较. 应用生物信息学技术分析发现, 其中3个克隆与未知功能基因序列高度同源, 并在GenBanK数据库注册, GenBanK号为AY450390, AY450394, AY450392. 另一个克隆未检索到任何对应的相似序列, 未知基因结构及功能分析正在进行中. 其余33个克隆均与已知基因的部分序列高度同源(96-100%), 详细结果(见表1).
| 已知的同源序列编码蛋白 | 相同克隆数 | 同源性(%) |
| 热休克蛋白Hsp 90 | 1 | 99 |
| CD164 | 1 | 98 |
| HMGB2 (high-mobility group box 2) | 1 | 100 |
| 硫氧还蛋白 | 1 | 99 |
| 微小组织相容性抗原H13 | 1 | 98 |
| glypican 3 | 1 | 98 |
| NADH脱氢酶 | 6 | 99 |
| Nephropontin前体 | 1 | 96 |
| NB4凋亡分化相关抗原 | 1 | 100 |
| RTN4异构体G(RTN4) | 1 | 99 |
| DEAD盒(Asp-Glu-Ala-Asp)多肽5 | 1 | 98 |
| 凝血因子XI | 1 | 98 |
| 阿朴脂蛋白M | 2 | 99 |
| BCL2/腺病毒E1B相互作用蛋白3 | 1 | 100 |
| Ras相关C3肉毒毒素作用物1 | 1 | 99 |
| 纤维粘连蛋白 | 1 | 98 |
| 电子转移黄素蛋白 | 1 | 100 |
| 原肌球蛋白4 | 1 | 97 |
| 真核翻译延伸因子 | 1 | 99 |
| HIV-1 Rev 结合蛋白 | 1 | 98 |
| 甲胎蛋白 | 2 | 99 |
| ERH(enhancer of rudimentary homolog) | 1 | 98 |
| N-乙酰氨基转移酶-6作用底物 | 1 | 99 |
| peroxiredoxin 3 | 1 | 98 |
| TATA盒结合蛋白相关因子(TAF13) | 1 | 99 |
| Crk作用物相关蛋白CAS-L | 1 | 98 |
HBV DNA P存在于Dane颗粒核心内, 是一种依赖于DNA的DNA P, 其功能与修补及延伸双链DNA的短链有关. 患者血清中HBV DNA P活性增高常伴有HBV增生. 在急性乙肝的潜伏期内, 血清ALT升高之前, 血清DNA P活力即已升高. 急性肝炎患者在发病1 mo后若HBV 活力仍持续升高, 是肝炎转为慢性的征兆. 病毒复制离不开HBV DNA P, 而HBV的持续性感染是慢性肝炎、肝硬化和HCC的首要原因.既往的研究多集中于HBV DNA P在病毒生活周期中的作用机制, 但随着对HBV致病机制研究的深入, 发现许多病毒基因组的反式激活蛋白, 以其表达产物的间接方式参与另外一些基因的功能调节. 可以对基因组内部的基因片段, 甚至可以对另外细胞或病毒的基因组有调控作用. 例如, HBxAg蛋白就是一种具有广泛活性的转录激活因子, 能够反式激活同源/异源的病毒/细胞转录调节序列, 对乙型肝炎的慢性化和促进肝细胞的转化有密切关系[19-24]. 那么, 肝细胞内表达的RNase H蛋白, 是否也能激活某些特定基因的表达? 这些表达产物对细胞的损害、增生分化的作用如何? 基于上述研究目的, 我们采用抑制性消减杂交方法, 克隆HBVDNA聚合酶的RNase H基因片段转染细胞, 反式激活差异表达的基因, 构建其cDNA消减文库, 为今后进一步分析、研究病毒蛋白的致病机制奠定基础.
SSH方法是近年发展起来的一项新的基因克隆技术, 与传统的方法比较, 具有实验周期短、易操作、可靠性高、假阳性率低等特点, 能有效地分离扩增低丰度特异表达的基因, 可以在较短的时间内获得较理想的实验结果.为研究RNase H蛋白的反式激活作用及其作用机制提供了新的研究方向和思路.实验将真核表达载体pcDNA3.1(-)- RNase H, 转染肝母细胞瘤细胞系HepG2, 并以转染空白载体的相同细胞系作为对照, 以2种转染的细胞系中提取的mRNA为起始材料, 应用SSH方法成功地构建了RNase H反式激活相关基因差异表达的cDNA消减文库, 随机挑选36个克隆测序并与GenBank数据库进行同源性比较分析, 结果主要包括2种类型, 第一种是已知基因的序列, 与GenBank中数据高度同源, 其中包括一些已知的看家基因序列和一些与细胞生长调节密切相关的基因序列; 甲胎蛋白、硫氧还蛋白、过氧化氧化还原蛋白(peroxiredoxin) 3、糖苷蛋白(glypican)3等可能与肝细胞恶性转化密切相关; 纤维粘连蛋白、原肌球蛋白4等细胞基质蛋白提示RNase H可能参与肝纤维化的发生发展过程; CD164、NB4凋亡分化相关抗原、BCL2/腺病毒E1B相互作用蛋白3、Ras相关C3肉毒毒素作用物1、电子转移黄素蛋白、N-乙酰氨基转移酶-6作用底物、Crk作用物相关蛋白CAS-L等与细胞周期生长调节及信号转导途径密切相关; 阿朴脂蛋白M可能参与了肝脏脂肪变性的发病机制; 凝血因子XI的表达上调也可能参与了RNase H致病机制; 真核翻译延伸因子、NADH脱氢酶、TATA盒结合蛋白相关因子、ERH等与细胞的转录、翻译功能密切相关, 其中ERH是一种转录抑制因子, Pogge et al[25]报道将其融合表达于GAL4转录因子的DNA结合结构域时 , ERH能抑制HeLa细胞中GAL4反应性报告基因的活性. 第二种是未知基因序列, 获得3个差异表达的未知序列. 对未知序列, 我们采用生物信息学方法获得了其全长基因序列, 已被GenBank收录.
在实验中我们观测到分子伴侣家族的表达上调. 分子伴侣[26-28]为一类与其他蛋白不稳定构象相结合并使之稳定的蛋白, 他们通过控制结合和释放来帮助被结合多肽在体内的折叠、组装、转运或降解等. 哺乳类胞质中的热休克蛋白 Hsp90属于分子伴侣家族, Hsp 90主要与细胞中不稳定的非活化蛋白结合, 促进其迅速活化或阻止其降解. 他可以与胞质中的类固醇激素受体结合, 封闭受体的 DNA 结合域, 阻碍其对基因转录调控区的激活作用, 使之保持在天然的非活性状态, 但也使受体保持着对激素配体的高亲和力. Hsp90 还与 Ras 信号途径中许多信号分子的折叠与组装密切相关, 主要通过与 Hsp90的结合与解离, 介导了这些分子在非活性形式与活性形式间的转化. 如转化型酪氨酸激酶 pp60v-src 或在一定条件下从与Hsp90 等形成的复合物中释放, 才能转位至胞膜行使激酶的活性功能. Hu et al[29]研究表明HBV DNA P与宿主因子如Hsp90复合物相互作用, 这对病毒基因组的复制及病毒粒子的装配是关键的一步. 他们用纯化的反转录酶和宿主因子在体外重建了RNP及蛋白质引物, 结果显示Hsp90能识别RT上对RNP及蛋白质引物必须的两个区域, Hsp90的功能对于建立及保持RT的RNA结合功能是必需的, 但Hsp90对RT的合成及翻译后活化不是必要的. 由此推测, Hsp90作为一个内部桥梁辅助RT的两个区域形成稳定而平衡的RNP结合形式. Cho et al[30]通过共转染及免疫共沉淀实验证实, Hsp90 N末端(aa 1-302)能与HBV Pol的末端蛋白(TP)及反转录区结合, 而C-末端(aa 483-723)只与反转录区结合, 中间(aa 327-438)一段与Hsp90没有相互作用. 我们发现RNase H的反式激活作用可引起Hsp90的表达上调, 结合Hu的实验结果提示RNase H反式激活Hsp90使其表达上调, 级联反应辅助DNA P发挥生物学作用, 此作用机制参与了HBV DNA的复制过程.
在筛选结果中我们也注意到多种与肝脏肿瘤发生有关的癌胚抗原, 除了众所周知的甲胎蛋白, 硫氧还蛋白、过氧化氧化还原蛋白3、糖苷蛋白3也是近年来研究的热点. 硫氧还蛋白, 过氧化氧化还原蛋白3特异性定位于线粒体, 作为抵御呼吸作用产生的H2O2的第一道防线, 在调节细胞内氧化还原状态方面具有重要作用; 而细胞内氧化还原状态的改变是参与细胞生长和死亡过程的一种关键因子, 并可导致肿瘤的发生. Choi et al[31]用免疫组织化学方法研究了58例石蜡包埋组织中硫氧还蛋白、过氧化氧化还原蛋白3的表达情况, 结果发现二者在肝癌组织中的表达水平明显高于邻近的正常肝组织, 作者认为由于增强了硫氧还蛋白, 过氧化氧化还原蛋白3的表达而改变了细胞内氧化还原状态, 这可能与肝细胞肿瘤的形成与发展有关. Nonn et al[32]的研究认为过氧化氧化还原蛋白3的表达增强还能保护肿瘤细胞逃避由于低氧和药物而诱导的H2O2依赖的细胞凋亡. 糖苷蛋白3是一种存在于细胞表面的硫酸肝素类蛋白多糖(HSPGs), HSPGs可作为一种共受体与生长因子相互作用并调节其活性, Midorikawa et al[33]报道过量表达的GPC是通过抑制纤维细胞生长因子2与骨形态发生相关蛋白7的活性而调节细胞增生, 作者认为通过对生长因子的调节可能有助于揭示GPC在肝脏肿瘤发生机制中的作用. Nakatsura et al[34], Sung et al[35]相继报道发现在肝肿瘤发生时, GPC尤其过量表达, 甚至许多学者[34-36]提议将其作为一种新型的肝细胞癌的血清学和组织化学的标志物, 对GPC 与AFP的同时检测可能明显提高肝细胞癌的诊断敏感性.这部分筛选结果提示RNase H的反式激活作用可能与肝细胞恶性转化密切相关, 为研究肝细胞癌的发病机制提供了理论依据.
已知不论在急性和慢性HBV感染, HBV DNA都能够整合到肝细胞染色体DNA中, HBV整合到细胞 DNA中, 能使细胞 DNA发生缺失、插入、转位、突变或易位等改变. 我们筛选到的HMGB2(high-mobility group box 2)的表达上调似与此过程有关; Ueda et al[37]曾报道HMGB2参与了非同源错误的重组, 其研究结果揭示了HMGB2的一种新的功能, 即能促进质粒DNA稳定整合到宿主基因组中, 作者认为这种作用对于自然条件下DNA整合到活细胞中非常重要. HMGB2的筛选发现对于研究HBV DNA的整合机制提供了新的突破口.
由于SSH不以研究对象的已知基因序列为前提, 所以为反式激活靶基因的研究提供了全新的探索方向, 对基因克隆的研究也具有重要的启迪作用. 当然, 应用这种方法只能对反式激活的靶基因进行初步分析, 实验中获得的新基因序列是否是真正的RNase H反式激活靶基因, 尚需进一步的研究; 如果得到证实, 那么本实验结果对于阐明HBVDNA聚合酶RNase H蛋白反式激活作用与HBV感染的慢性化及致肝细胞癌的发生之间的关系具有重要的理论意义.
编辑: N/A
| 1. | 成 军, 杨 守纯. 现代肝炎病毒分子生物学, 第1版. 北京: 人民军医出版社 1997; 83-103. |
| 2. | Rossner MT. Review: hepatitis B virus X-gene product: a promiscuous transcriptional activator. J Med Virol. 1992;36:101-117. [PubMed] [DOI] |
| 4. | Wang X, Grammatikakis N, Hu J. Role of p50/CDC37 in hepadnavirus assembly and replication. J Biol Chem. 2002;277:24361-24367. [PubMed] [DOI] |
| 5. | Stuyver LJ, Locarnini SA, Lok A, Richman DD, Carman WF, Dienstag JL, Schinazi RF. Nomenclature for antiviral-resistant human hepatitis B virus mutations in the polymerase region. Hepatology. 2001;33:751-757. [PubMed] [DOI] |
| 6. | Jasper ZP, Robert EL, Rolf IC, Lena N. Properties of monoclonal antibodies directed against hepatitis B virus polymerase protein. J Virol. 1999;73:4188-4196. |
| 7. | Wang XT, Hu JM. Distinct requirement for two stages of protein-primed initiation of reverse transcription in hepadnaviruses. J Virol. 2002;76:5857-5865. [DOI] |
| 12. | 刘 妍, 董 菁, 成 军, 夏 小兵, 李 克, 王 琳, 施 双双, 段 惠娟, 杨 继珍. 乙肝病毒X基因在真核细胞中的表达及反式激活SV40病 毒早期启动子的研究. 解放军医学杂志. 2001;26:404-406. |
| 14. | 董 菁, 刘 妍, 黄 甫竞坤, 施 双双, 王 刚, 洪 源, 陈 国凤, 李 莉, 陈 菊梅, 成 军. 慢性乙肝病毒感染患者乙肝病毒表面抗原基本 结构多态性研究. 胃肠病学和肝病学杂志. 2002;11:130-135. |
| 16. | Kim Y, Hong YB, Jung G. Hepatitis B virus: DNA polymerase activity of deletion mutants. Biochem Mol Biol Int. 1999;47:301-308. [PubMed] [DOI] |
| 17. | Walton CM, Wu CH, Wu GY. A ribonuclease H-oligo DNA conjugate that specifically cleaves hepatitis B viral messenger RNA. Bioconjug Chem. 2001;12:770-775. [PubMed] [DOI] |
| 18. | Luda D, Yun-Fai CL, Aaron PC. Suppression subtractive hybridization : a method for generating differentially regulated or tissue-specific cDNA probes and libraries. Proc Natl Acad Sci USA. 1996;93:6025-6030. [DOI] |
| 19. | Spandau DF, Lee CH. trans-activation of viral enhancers by the hepatitis B virus X protein. J Virol. 1988;62:427-434. [PubMed] |
| 20. | Aufiero B, Schneider RJ. The hepatitis B virus X-gene product trans-activates both RNA polymerase II and III promoters. EMBO J. 1990;9:497-504. [PubMed] |
| 21. | Twu JS, Robinson WS. Hepatitis B virus X gene can transactivate heterologous viral sequences. Proc Natl Acad Sci U S A. 1989;86:2046-2050. [PubMed] [DOI] |
| 22. | Seto E, Mitchell PJ, Yen TS. Transactivation by the hepatitis B virus X protein depends on AP-2 and other transcription factors. Nature. 1990;344:72-74. [PubMed] [DOI] |
| 23. | Faktor O, Budlovsky S, Ben-Levy R, Shaul Y. A single element within the hepatitis B virus enhancer binds multiple proteins and responds to multiple stimuli. J Virol. 1990;64:1861-1863. [PubMed] |
| 24. | Faktor O, Budlovsky S, Ben-Levy R, Shaul Y. A single element within the hepatitis B virus enhancer binds multiple proteins and responds to multiple stimuli. J Virol. 1990;64:1861-1863. [PubMed] [DOI] |
| 25. | Pogge von Strandmann E, Senkel S, Ryffel GU. ERH (enhancer of rudimentary homologue), a conserved factor identical between frog and human, is a transcriptional repressor. Biol Chem. 2001;382:1379-1385. [PubMed] |
| 26. | Braig K. Chaperonins. Curr Opin Struct Biol. 1998;8:159-165. [PubMed] [DOI] |
| 27. | Hartl FU. Molecular chaperones in cellular protein folding. Nature. 1996;381:571-579. [PubMed] [DOI] |
| 28. | Burston SG, Clarke AR. Molecular chaperones: physical and mechanistic properties. Essays Biochem. 1995;29:125-136. [PubMed] |
| 29. | Hu J, Toft DO, Seeger C. Hepadnavirus assembly and reverse transcription require a multi-component chaperone complex which is incorporated into nucleocapsids. EMBO J. 1997;16:59-68. [PubMed] [DOI] |
| 30. | Cho G, Suh SW, Jung G. HBV polymerase interacts independently with N-terminal and C-terminal fragments of Hsp90beta. Biochem Biophys Res Commun. 2000;274:203-211. [PubMed] [DOI] |
| 31. | Choi JH, Kim TN, Kim S, Baek SH, Kim JH, Lee SR, Kim JR. Overexpression of mitochondrial thioredoxin reductase and peroxiredoxin III in hepatocellular carcinomas. Anticancer Res. 2002;22:3331-3335. [PubMed] |
| 32. | Nonn L, Berggren M, Powis G. Increased expression of mitochondrial peroxiredoxin-3 (thioredoxin peroxidase-2) protects cancer cells against hypoxia and drug-induced hydrogen peroxide-dependent apoptosis. Mol Cancer Res. 2003;1:682-689. [PubMed] |
| 33. | Midorikawa Y, Ishikawa S, Iwanari H, Imamura T, Sakamoto H, Miyazono K, Kodama T, Makuuchi M, Aburatani H. Glypican-3, overexpressed in hepatocellular carcinoma, modulates FGF2 and BMP-7 signaling. Int J Cancer. 2003;103:455-465. [PubMed] [DOI] |
| 34. | Nakatsura T, Yoshitake Y, Senju S, Monji M, Komori H, Motomura Y, Hosaka S, Beppu T, Ishiko T, Kamohara H. Glypican-3, overexpressed specifically in human hepatocellular carcinoma, is a novel tumor marker. Biochem Biophys Res Commun. 2003;306:16-25. [PubMed] [DOI] |
| 35. | Sung YK, Hwang SY, Park MK, Farooq M, Han IS, Bae HI, Kim JC, Kim M. Glypican-3 is overexpressed in human hepatocellular carcinoma. Cancer Sci. 2003;94:259-262. [PubMed] [DOI] |
| 36. | Capurro M, Wanless IR, Sherman M, Deboer G, Shi W, Miyoshi E, Filmus J. Glypican-3: a novel serum and histochemical marker for hepatocellular carcinoma. Gastroenterology. 2003;125:89-97. [PubMed] [DOI] |
| 37. | Ueda T, Shirakawa H, Yoshida M. Involvement of HMGB1 and HMGB2 proteins in exogenous DNA integration reaction into the genome of HeLa S3 cells. Biochim Biophys Acta. 2002;1593:77-84. [PubMed] [DOI] |