修回日期: 2004-02-09
接受日期: 2004-04-05
在线出版日期: 2004-06-15
目的: 微卫星不稳定(microsatellite instability MSI)是DNA复制错误(replication error RER+)的标记, BAT-26系位于错配修复基因hMSH2中的单腺苷酸重复序列位点, 曾被报道无需正常对照就可用于预测散发性大肠癌RER状况, 我们选择BAT-26并与其他双核苷酸重复序列位点进行比较加以验证与评价.
方法: 经病理确诊的散发性大肠癌肿瘤组织60例, 常规抽提DNA, 采用PCR-银染法分析6个(CA)n双碱基重复微卫星序列和BAT-26位点, 并用自动荧光DNA序列分析法检测BAT-26; 同时以PCR-SSCP法检测RER+病例hMSH2基因5, 7, 8, 12, 13, 15外显子突变; RER+判断依6个(CA)n位点中出现2个或以上MSI为标准.
结果: RER+散发性大肠癌占18%(11/60), 6例RER+肿瘤检出hMSH2基因突变. 含hMSH2基因突变的6例RER+病例中有3例BAT-26不稳定(BAT-26+), 44例RER-散发性大肠癌均为BAT-26-. 与(CA)n位点结果比较, BAT-26预测RER的阳性符合率为50%, 阴性符合率为100%, BAT-26+在预测散发性大肠癌RER+特异性为100%, 敏感性为50%. PCR-银染和自动荧光DNA序列分析两种方法检测BAT-26 MSI结果一致.
结论: 单用BAT-26作为预测RER状况的指标尚有缺陷, 需结合其他微卫星位点进行综合分析.
引文著录: 邱福铭, 宋永茂, 黄建, 郑树. BAT-26预测散发性大肠癌的复制错误. 世界华人消化杂志 2004; 12(6): 1461-1463
Revised: February 9, 2004
Accepted: April 5, 2004
Published online: June 15, 2004
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2004; 12(6): 1461-1463
- URL: https://www.wjgnet.com/1009-3079/full/v12/i6/1461.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i6.1461
微卫星不稳定(MSI)可表现为RER+, 约90%以上遗传性非息肉性大肠癌(Hereditary non-polyposis colorectal cancer HNPCC)和约10-15%散发性大肠癌中存在RER+[1]. BAT-26是位于错配修复基因hMSH2第5内含子内的26个腺苷酸重复序列, 具有准单态性, 有研究表明绝大部分RER+原发性大肠癌表现为BAT-26+[2], 我们选择BAT-26并与其他双核苷酸重复序列位点进行比较加以验证与评价.
1996/1997年浙江大学医学院附属第二医院肿瘤科收治的经病理确诊的散发性大肠癌患者60例. 由于部分样本DNA用尽, 对其中50例样本包括6例RER+标本分别用PCR-银染法、自动荧光DNA序列分析法检测BAT-26位点.
新鲜肿瘤组织和相应正常组织采用酚/氯仿抽提法抽提DNA. PCR-银染法分析6个(CA)n双碱基重复微卫星序列D2S119, D13S160, D8S282, D3S1293, D2S123, D18S58, 各位点引物和反应条件参见文献[3], 如肿瘤组织相对于正常组织出现额外的条带、等位条带产生迁移率的改变就定义为MSI, 在6个位点中出现2个或以上MSI即确定为RER+. PCR-SSCP检测hMSH2基因外显子5, 7, 8, 12, 13, 15突变, PCR反应体系和PCR反应参数见文献[4]. BAT-26位点PCR引物和反应条件见文献[2], PCR产物先采用常规的PCR-银染法检测确定MSI, 再用全自动测序仪(ABI 3777 DNA Sequencer)分析: TAMARA标记引物, 基因扫描分析软件genescan分析, 若肿瘤组织出现相应正常组织没有的峰即可确定为MSI.
散发性大肠癌60例, 采用6个双核苷酸重复序列(CA)n位点, 检出11例RER+病例, 阳性率为18%. 其中6例RER+病例均发现有hMSH2第5外显子突变, 其他外显子未检测到突变.
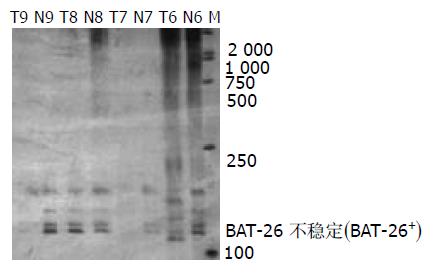
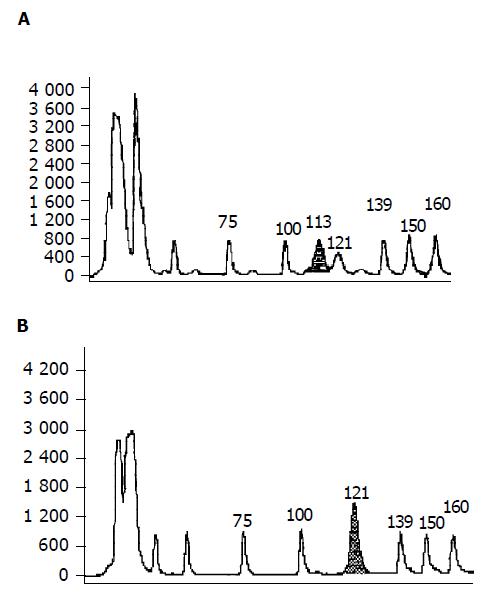
分别采用PCR-银染法和自动荧光全自动测序仪分析50例(6例RER+和44例RER-)散发性大肠癌BAT-26, 均发现仅在相同的3例病例出现BAT-26+, 且均为RER+病例, 其余的44例均为BAT-26-. 图1仅第6号肿瘤出现异常条带(箭头所指),第6号肿瘤样本自动荧光DNA序列仪分析目的片段长度(113 bp)比正常组织(121.0 bp)缩短了8 bp (图2).
6个(CA)n双碱基重复微卫星序列和BAT-26对RER+状况预测的阳性率(RER+病例中各微卫星位点MSI+的比例)和假阳性率(RER-病例中各微卫星位点MSI+的比例)进行比较(表1), 阳性率超过50%的微卫星位点有D3S1293, D3S1293, BAT-26, 假阳性率为零的微卫星位点有D13S160, D18S58, BAT-26. 在有hMSH2基因突变的RER+散发性大肠癌仅有3例BAT-26不稳定(表2). 与(CA)n位点结果比较, BAT-26预测RER状况的阳性符合率为50%, 阴性符合率为100%, BAT-26+在预测散发性大肠癌RER+特异性为100%, 敏感性为50%.
| D2S119 | D13S160 | D8S282 | D3S1293 | D2S123 | D18S58 | BAT26 | |
| 阳性率% | 5/11(45) | 4/11(36) | 3/11(27) | 7/11(64) | 6/11(55) | 5/11(45) | 3/6(50) |
| 假阳性率% | 3/49(6.1) | 0/49(0) | 4/49(8.2) | 2/49(4.1) | 1/49(2.0) | 0/49(0) | 0/44(0) |
| NO. | 基因突变 | BAT-26 |
| 5 | + | - |
| 6 | + | + |
| 17 | + | - |
| 28 | + | + |
| 29 | + | - |
| 48 | + | + |
大肠癌错配修复途径主要涉及绝大部分遗传性肿瘤和少部分散发性肿瘤, 错配修复缺陷即RER+大肠癌具有不同于RER-肿瘤的诸多临床病理特点, 确定大肠癌RER状况对治疗选择和预后判断有一定指导作用. BAT-26具有准单态性, 一般在正常细胞内其碱基长度变化不超过2个碱基. 在HNPCC相关大肠癌中, Loukola et al[5]报告BAT-26预测大肠癌的RER阳性率为55%(27/49); Stone et al[6]比较单和双核苷酸重复序列位点预测RER状况, 发现BAT-25、BAT-26敏感性更高; Hoang et al[2]研究显示高达97%的RER+大肠癌表现为BAT-26+(片段大小缩短>2 bp), 提出BAT-26可单独作为预测RER状况的检测指标, 不需设正常对照. 然而, 日本的一项研究发现8例RER+大肠癌仅有4例出现BAT-26位点单个碱基的改变, 提示单独应用BAT-26预测RER+存在缺陷, 认为需结合其他微卫星位点进行准确检测[7]. 迄今, 有关国人的相关研究尚为数不多, 为此我们选择BAT-26并与其他双核苷酸重复序列位点进行比较加以验证与评价. 我们选择了6个(CA)n双碱基重复微卫星序列D2S119、D13S160、D8S282、D3S1293、D2S123、D18S58测定散发性大肠癌RER状况, 有11例大肠癌呈RER+, 其中6例标本发现有hMSH2基因突变. 同时, PCR-SSCP银染和自动荧光DNA序列仪对上述6例标本分析BAT-26位点, 均证实相同3例病例为BAT-26+, 其余无一表现为BAT-26+. 由于错配修复基因突变是MSI的分子基础, 如果将hMSH2基因突变作为RER状况的金标准, BAT-26预测的特异性虽为100%, 但敏感性仅有50%, 所选的(CA)n微卫星位点如D13S160、D18S58也有类似的特异性和敏感性; 与(CA)n位点结果比较, BAT-26预测RER状况的阳性符合率为50%, 阴性符合率为100%, 因此BAT-26单独预测散发性大肠癌RER状况, 由于其敏感性较低, 可在一定程度上造成RER+病例的遗漏, 故仍需结合其他位点进行检测来提高准确性. 目前国际上推荐五个微卫星位点也就是Bethesda微卫星标记 (BAT-25, BAT-26, D5S346, D2S123, 和D17S250)进行检测[8]. 在BAT-26的MSI检测方面, 两种方法检测结果完全符合. 但自动荧光DNA序列仪分析法与PCR-SSCP银染法相比较, 具有明显的特点: 能准确、定量和比较客观地反映MSI, 能达到测定单个碱基改变的精确度[9]; 利用不同荧光标记, 两个样本可在同一泳道上同时进行电泳, 从而避免了不同泳道间细微差异可能造成的误差; 此外, 如果选用更多的荧光标记, 在同一泳道上最多可同时进行3对样本电泳, 极大地提高了检测效率. 因此, 自动荧光DNA序列分析法为准确、快速、高通量检测MSI的有效方法.
| 2. | Hoang JM, Cottu PH, Thuille B, Salmon RJ, Thomas G, Hamelin R. BAT-26, an indicator of the replication error phenotype in colorectal cancers and cell lines. Cancer Res. 1997;57:300-303. [PubMed] |
| 3. | Huang J, Papadopoulos N, McKinley AJ, Farrington SM, Curtis LJ, Wyllie AH, Zheng S, Willson JK, Markowitz SD, Morin P. APC mutations in colorectal tumors with mismatch repair deficiency. Proc Natl Acad Sci USA. 1996;93:9049-9054. [PubMed] [DOI] |
| 5. | Loukola A, Eklin K, Laiho P, Salovaara R, Kristo P, Järvinen H, Mecklin JP, Launonen V, Aaltonen LA. Microsatellite marker analysis in screening for hereditary nonpolyposis colorectal cancer (HNPCC). Cancer Res. 2001;61:4545-4549. [PubMed] |
| 6. | Stone JG, Tomlinson IP, Houlston RS. Optimising methods for determining RER status in colorectal cancers. Cancer Lett. 2000;149:15-20. [PubMed] [DOI] |
| 7. | Morifuji M, Hiyama E, Murakami Y, Imamura Y, Sueda T, Yokoyama T. Fluorescent-based BAT-26 analysis for distinct screening of microsatellite instability in colorectal cancers. Int J Oncol. 2003;22:807-813. [PubMed] [DOI] |
| 8. | Boland CR, Thibodeau SN, Hamilton SR, Sidransky D, Eshleman JR, Burt RW, Meltzer SJ, Rodriguez-Bigas MA, Fodde R, Ranzani GN. A National Cancer Institute Workshop on Microsatellite Instability for cancer detection and familial predisposition: development of international criteria for the determination of microsatellite instability in colorectal cancer. Cancer Res. 1998;58:5248-5257. [PubMed] |
| 9. | Toh Y, Oki E, Oda S, Tomoda M, Tomisaki S, Ichiyoshi Y, Ohno S, Sugimachi K. An integrated microsatellite length analysis using an automated fluorescent DNA sequencer. Cancer Res. 1996;56:2688-2691. [PubMed] |