修回日期: 2003-12-01
接受日期: 2003-12-06
在线出版日期: 2004-04-15
抑制性消减杂交技术(Suppressive subtractive hybridization, SSH)是近年来兴起的一种检测差异表达基因的方法, 广泛用于寻找疾病及肿瘤相关基因、生长和发育相关基因、药物的筛选和靶向治疗及其明确基因的功能等. 本文从SSH技术的原理、效率、存在的问题和展望及其在感染病学中的应用等方面进行了综述.
引文著录: 王方, 王宇明. 抑制性消减杂交技术及其在感染病学中的应用. 世界华人消化杂志 2004; 12(4): 972-975
Revised: December 1, 2003
Accepted: December 6, 2003
Published online: April 15, 2004
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2004; 12(4): 972-975
- URL: https://www.wjgnet.com/1009-3079/full/v12/i4/972.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i4.972
新近人类基因组计划(human genome project, HGP)顺利完成, 通过对人类基因组结构的了解, 建立了较为系统的庞大数据库; 其他物种的基因组结构亦在不断地阐明与完善. 然而, HGP的完成仅仅是阐明人类众多基因的基础和必经之路. 基因是细胞的遗传物质, 决定细胞的生物学性状. 细胞的生物学功能最终是由大量的基因表达产物即蛋白质来完成的. 基因表达是一个复杂而精细的网络调控过程, 细胞的生物学事件是在某一特定的时间与空间里, 多种基因表达及其相互调控的结果. 明确细胞在生理与病理条件下的基因表达及其调控过程, 将对细胞的生理与病理状态的干预提供极大的帮助.高等真核生物约含有10万个不同基因, 但在生物体的发育过程中只有15%的基因得以表达, 而这大约15 000个基因的表达是按时间和空间顺序有序地进行着, 这种表达方式即为基因的差别表达(differential expression). 而分子生物学的首要任务之一便是要比较不同细胞或不同基因型在基因表达上的差异, 这不仅是研究生命过程分子机制的基础, 亦是分离克隆目的基因的前提.为了更有效地发现差异表达基因, 近年来发展了一系列方法, 包括差异显示PCR (differential display PCR, DD-PCR)、RNA指纹技术(RNA fingerprinting)、抑制性消减杂交技术(suppressive subtractive hybridization, SSH)、基因系列表达分析(sequence analyses of gene expression, SAGE)、Real-time Quantitative PCR (TaqMan) 、DNA微阵列(DNA microarray, 或 gene chip)等. 尽管每一种方法均有各自的优缺点, 但总的来说筛选差异表达基因的方法已从既费时又费力的方法, 如聚丙酰胺凝胶为基础的差异显示发展到自动高通量检测方法, 如DNA微阵列, 但微阵列只能检测已知基因, 不能发现未知基因. 消减杂交(subtractive hybridization, SH)是一种富集差异表达基因的有效方法, 这种方法最初是60年代中期由Bautz和Reilly使用, 用于纯化噬菌体T4 mRNA[1], 这种单纯杂交方法有一定的限制, 因为需要大量的mRNA进行杂交, 杂交后微量的cDNA进行克隆亦较困难. 这种方法被Duguid 和 Dinauer进一步优化, 在cDNA后加接头, 可以进行选择性PCR扩增[2]. Diatchenko和Gurskaya et al[3-4]又进一步改进了抑制性杂交PCR技术, 即SSH 方法, 经过几轮循环可以将差异表达基因扩增1 000倍.
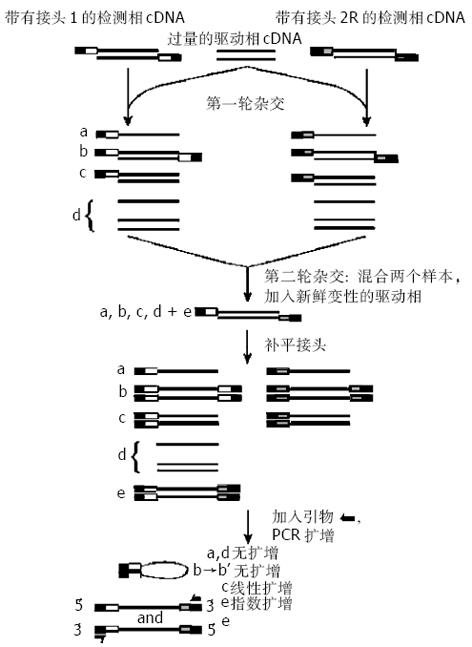
SSH是抑制性PCR与消减杂交技术相结合的更简单、更快速的分离差异基因的方法. 该方法运用了杂交二级动力学原理, 即丰度高的单链DNA在退火时产生同源杂交的速度快于丰度低的单链DNA, 从而使原来在丰度上有差别的单链DNA相对含量达到基本一致. 而抑制PCR则是利用链内退火优于链间退火的特点, 使非目的序列片段两端反向重复序列在退火时产生类似发卡的互补结构, 无法作为模板与引物配对, 从而选择性抑制非目的基因片段的扩增. 这样既利用了消减杂交技术的消减富集, 又利用了抑制性PCR技术进行了高效率的动力学富集.具体操作流程见图1.
第1步: SSH技术首先将由mRNA逆转录来的检测子cDNA(Tester)和驱动子cDNA(Driver)分别用同一种识别四碱基序列的限制性内切酶RsaⅠ消化形成平均长度大于500 bp的cDNA片段, 这样可增加有差别表达基因检出的可能性, 以防止长链片段形成的复杂结构对消减杂交的干扰.
第2步: 连接接头(Adaptor). 将Tester cDNA均分为两份, 分别接上接头Adaptor 1和Adaptor2. Adaptor设计十分巧妙, 由一长链(40 nt)和一短链(10 nt)组成的一端是平端的双链DNA片段, 且双链5'端均无磷酸化基团, 保证了Adaptor以惟一方向与cDNA片段连接. 接头外侧序列与第1次PCR引物序列相同, 而内侧序列则与第2次PCR(巢式PCR)引物序列相同, 有利于后续PCR的筛选扩增. 此外接头上含有T7启动子序列及内切酶识别位点, 为以后克隆和测序提供方便.
第3步: 两轮消减杂交. 用过量Driver cDNA样品与上述两份连有接头的Tester cDNA样品进行第1次消减杂交, 分别得到4种产物. 这种不充分杂交使得单链cDNA分子在浓度上基本相同. 同时由于Tester cDNA与Driver cDNA序列中相同片段大都形成异源双链分子c, 使得Tester cDNA中差异表达基因得到第一次富集; 然后混合上述两份杂交样品, 同时加入新的变性Driver cDNA进行第2次消减杂交.此次杂交只有第1次杂交后经扣除和丰度均等化的单链Tester cDNA能与Driver cDNA形成双链分子. 这一次杂交进一步富集了差异表达的cDNA, 并且还形成了2个5′端分别接不同接头的双链分子e.
第4步: 两次PCR反应. 5'端填平末端, 加入根据接头设计的引物进行两次PCR. 第1次PCR基于抑制反应, 只有两端连接有2个不同接头的双链cDNA片段(如e)才能得到指数扩增. 第2次PCR实际上是巢式PCR, 极大地提高了扩增特异性, 使差异表达的目的基因片段得到大量富集.然后利用接头上存在的酶切位点, 插入适当载体, 转化细菌, 初步筛选出具有插入的克隆, 再经杂交筛出具有差异表达的克隆, 进行测序、同源分析等系列工作, 最后完成全长基因的克隆、鉴定.
对于一个理想的消减系统, 不管目的基因丰度如何, 均能产生有效富集, 且在耗时进行筛选之前, 就能鉴定消减效率, 以最大限度地降低假阳性. 那么, SSH技术的消减效率究竟如何呢?为了进一步指导SSH技术的应用, Ji et al[5]首次用数学模型和实验模型全面研究了其实际的效率和局限性.
Ji et al 根据杂交二级动力学原理推算出的数学模型表明, 影响SSH效率的一个主要因素是靶基因的浓度和其在Tester 和Driver中的比例R, 浓度越高, 比例越大, SSH 富集的效率越高; 靶基因富集的效率和R3成正比. 实验模型用来进一步证实理论模型的结论.
在实验模型中, 首先检测SSH富集存在于Tester而不存于Driver中的靶基因的效率. 将fx174 DNA 和成纤维细胞(Fibroblast)cDNA混合后作为Tester, Fibroblast cDNA作为Driver, fx174 DNA为靶基因. 结果当fx174 DNA的含量大于总Tester的0.01%时, 凝胶电泳可以看到清晰的fx174 DNA条带; 当fx174 DNA含量在总Tester的0.1-1%之间时, 凝胶电泳看到fx174 DNA条带的清晰度同前, 说明SSH PCR富集fx174 DNA的效率同前; 而当fx174 DNA含量小于总Tester的0.01%时, 凝胶电泳看不到清晰的fx174 DNA条带, 这和理论模型不太一致, 此时富集的不是靶基因fx174 DNA, 而是随机扩增的Fibroblast cDNA片段, 原因可能是当Tester中fx174 DNA含量低于0.01%时, 在第二次杂交时2条链不能有效退火, 而在后续的PCR扩增中双链cDNA是必需的. 进一步检测在Tester和 Driver中均存在但丰度不同的靶基因被富集的效率. Tester 中含固定的1% 的靶基因fx174 DNA, 而Driver中所含靶基因fx174 DNA的浓度依次从0%递减到 0.1%. 结果发现当Tester中靶基因的含量比Driver中高5倍以上时, 凝胶电泳可以看到清晰的靶基因条带, 当Tester中的靶基因含量低于Driver中的5倍时, 则看不到靶基因条带; 再进一步改变靶基因在Tester和Driver中的浓度比例和丰度: Tester cDNA中含 0.1% fx174 DNA, 而Driver cDNA中fx174 DNA的浓度依次从0.1%到0, 结果和前一次相同, 有效的靶基因富集主要取决于靶基因在Tester和 Driver中的浓度比, 而和靶基因的绝对含量没有关系, 即当Tester中fx174 DNA浓度大于Driver中的5倍时, 则靶基因得到有效富集.
该实验模型结果对于有效地应用SSH技术和合理解释SSH PCR的实验结果很有意义. 要获得有效的靶基因富集, 对于只存在于Tester中的靶基因而言, 靶基因至少应占Tester的0.01%, 对于表达丰度不同的基因, 靶基因在Tester中的浓度应大于Driver中的5倍. 实际的PCR扩增效率也可能影响到SSH PCR的效率, 但为了简化模型, 没有在模型中考虑.
SSH技术可以研究细菌病原体的致病株和非致病株或弱致病株之间的基因序列差异. 这种基因序列差异可能和细菌的毒力岛(pathogenicity islands, PIs)有关. PIs是存在于致病株的基因序列, 和细菌的致病性有关. PIs具有一些共同的结构特征, 包括非典型的鸟嘌呤加胞嘧啶的含量百分比, 侧翼区存在重复或插入序列等结构特征[6].
SSH技术最初由Akopyants et al[7]应用于研究幽门螺杆菌致病株的致病基因, 后来相继应用于大肠杆菌[8]、亲水气单胞菌[9]、B. pseudomallei菌[10]、肺炎杆菌的致病株毒力相关基因[11]. Janke et al[8]用SSH技术检测致病性大肠埃希氏杆菌株536和非致病株K-12 株MG1655之间的差异表达基因, 发现有22个毒力株536特异性差异表达基因, 其中5个和已知毒力决定簇具有同源性, 可能代表部分毒力岛基因. Reckseidler et al[10]用SSH技术检测毒力株B. pseudomallei 和无毒株B. thailandensis之间的差异表达基因, 结果发现了B. pseudomallei株编码荚膜多糖的糖基转移酶基因, 当糖基转移酶基因被失活以后, 在动物模型中B. pseudomallei株和B. thailandensis株的LD50值相似, 说明这个糖基转移酶基因和荚膜的产生对细菌毒力很重要. Reckseidler还发现用SSH技术鉴定的消减克隆基因组具有非典型的鸟嘌呤加胞嘧啶含量百分比, 而此非典型特征是PIs岛的一个特征, 表明该方法对于检测细菌毒力株的特异序列十分有效. 最近, Han et al[12]用SSH技术发现从胃溃疡患者和非溃疡患者分离出的幽门螺杆菌株的基因序列存在一些特异的差别, 这些差别和菌株的致病性有关.
徐晓春et al[13]将被约氏疟原虫感染的斯氏按蚊作检测子, 未被感染的斯氏按蚊作驱动子, 二者之间做SSH, 寻找与约氏疟原虫感染相关的应答基因. 表达上调的24个克隆中, 12个为功能已知基因, 4个为功能未知基因, 7个为新基因, 其中有9个基因与经过细菌脂多糖(lipopolysaccharide, LPS)处理的免疫细胞来源的基因序列具有同源性, 表明在疟原虫感染斯氏按蚊过程中涉及多种生物化学过程, 尤其与免疫系统和能量代谢有关. Patzwahl et al[14]用SSH技术分离出相同程度病理改变的HCV感染肝组织和非病毒性肝脏疾病肝组织之间的差异应答基因, 分别是-干扰素(-IFN)诱导的化学趋化因子基因、干扰素诱导蛋白10 (IP-10)基因、/-IFN诱导的抗病毒MxA基因、/-IFN诱导的p44基因、//-IFN诱导的IFI-56K基因, 同非病毒感染引起的肝脏疾病相比, 这些基因的表达增高50倍. Riemer et al[15]用同样的方法检测瘙痒病感染后脑组织应答基因, 发现有19个基因表达上调, 其中有相当一部分基因的表达可以被IFN诱导, 如IP-10、2', 5'-寡核苷酸合成酶、Mx蛋白、IIGP蛋白、HLA-Ⅰ和Ⅱ、补体、2-微球蛋白等, 表明干扰素反应在瘙痒病的发病机制中起重要作用.
了解病原体在不同发育期的差异表达基因对于了解病原体的生理发育和致病机制具有重要意义. Dessens et al[16]用SSH技术成功扩增了疟原虫子孢子在蚊子中肠期的差异表达基因. 将存在于蚊子中肠期的虐原虫P. berghei ANKA 克隆234作检测子, 血液中无性阶段的非配子体克隆233作驱动子, 经过筛选和鉴定发现4个差异表达基因; 而Matuschewski et al[17]则发现有30个基因在唾液腺子孢子内上调表达, 其中一个基因编码蛋白激酶, 这些基因编码的产物可能与疟原虫在肝细胞内感染和发育有关. Bellatin et al[18]用同样的方法发现2个基因显著表达于利什曼原虫无鞭毛体阶段: 新基因A600和微管蛋白基因, Northern印迹法检测到这2个基因的表达升高6倍. 序列分析表明A600包含1个293 bp的开放读码框架, 编码一个膜蛋白或一个分泌蛋白, 可能对于无鞭毛体的分化或在吞噬溶酶体内的生存起重要作用.
刘妍et al[19]用SSH技术在转染HBX的pcDNA3.1(-)-X载体和空载体pcDNA3.1(-)之间寻找HBX转染相关基因. 结果共有85个阳性克隆, 将其中的65个克隆进行序列分析, 有19个功能已知基因和15个功能未知基因, 正在进一步研究其功能. 刘杰et al[20]在转染编码HBxAg基因的重组逆转录病毒载体的HepG2 x细胞和转染空载体的HepG2Cat细胞之间做SSH, 克隆和HBxAg有关的全长基因片段, 用原位杂交和Northern印迹进行差异筛选, 用RACE PCR扩增全长, 结果发现一个C2基因的表达在HepG2 x细胞被抑制, 其全长为1.35 kb, 编码一个含113个氨基酸的小蛋白. 将C2基因导入HepG2细胞可以抑制细胞的生长, 在裸鼠可以抑制肿瘤的生长. C2基因也表达于正常肝脏, 但肝癌组织中无表达. 结果表明HBx可以通过调节基因表达而促进或抑制细胞生长.
SSH综合了DD PCR和RDA两种技术的特点, 因此具备两项技术各自的优点且相应地改进了各自的缺点. Liu et al[21]用DNA 阵列、SSH、DD-PCR 3种方法寻找用全反式维甲酸作用于急性早幼粒细胞白血病NB4细胞系的差异表达基因. 用588个已知基因构成的DNA阵列中, 99个基因的信号强度大于2倍, 37个表达上调, 62个表达下调; 用DD-PCR法检测到的30个克隆经Northern 杂交分析只有13个代表差异应答基因; 用SSH方法得到703个克隆, 代表316个基因, 其中278个表达上调, 38个表达下调; 159个是已知基因, 132个是新序列片断. 经逆向Northern 印迹和半定量RT-PCR筛选, 159个已知基因中63个差异表达, 132个新基因中, 49个差异表达. 用3种不同的方法, 共检测出169个差异应答基因, 100个上调, 69个下调, 结果是可信的. 如RIG-E, RIG-G和细胞色素P450基因用DD-PCR和SSH检测结果一致, 有16个基因, 如MCP、IL-8、 Src样蛋白和 Bfl-1等用cDNA阵列和SSH均得到证实. 3种方法中, cDNA阵列仅含588个已知基因, 范围有限, 而DD-PCR 仅得到部分差异表达基因, 只有SSH比较全面地检测了差异表达基因.
SSH也存在一些缺点和不足. 既往人们普遍认为用SSH寻找差异表达基因时, Tester和Driver之间的背景要尽可能相同, 如同一个体的病变和正常组织间, 同一组织的不同发育或治疗时期, 但我们从Ji et al[5]的试验模型和理论模型可以看出, SSH 检测低丰度表达的靶基因的能力是有限的. 要获得有效的靶基因富集, 对于只表达于Tester的基因, 靶基因含量至少应占Tester的0.01%, 对于表达丰度不同的基因, Tester中靶基因浓度应大于Driver的5倍. 所以Ji et al认为SSH方法主要用于富集高度差异表达的基因, 如病毒感染前后或2种不同组织中的差异表达基因谱; 而同一组织患病前后或同一组织的不同发育或治疗时期, 基因表达的变化可能和生理学变化有关, 丰度较低, 用SSH PCR不能有效富集, 用其他方法筛选大量SSH PCR文库既费时又费力. Li et al[22]用鼠血脑屏障的毛细血管与鼠的肝和肾之间做SSH寻找与血脑屏障有关的特异表达基因, 以期从分子水平阐明脑血管病变的机制及发现促进药物通过血脑屏障的新靶标, 就是一个用SSH在不同的组织间寻找差异表达基因的典范; 而Nagayama et al 用SSH在正常个体和患病个体间成功地寻找到与自身免疫性肝炎发病相关的基因IP-10. Patzwahl et al[14] 用SSH在不同病因引起的病理改变基本相同的不同个体间成功地发现了与慢性HCV感染有关的应答基因.
除此之外, SSH还存在一些其他缺点和不足, 如对起始材料相对要求较多, 不适于来源困难的样品, 且若用cDNA做SSH则需来源样品保持新鲜以防RNA降解造成信息丢失; 非酶切位点区域内的点突变, 小缺失或插入均不能有效被发现; 得到的片段尚需扩增其全长; 筛选过程较繁杂. 在实际应用中, 选择合适的内切酶消化是关键, 提高接头与片段的连接效率则是整个筛选过程有效性的保证, 选择高效的筛选方法可以提高靶基因的检出率; 用不同方法相互结合可以取长补短, 提高差异应答基因的检出率. 如Nishizuka et al 先后用DD-PCR及SSH检测非致瘤性和致瘤性HeLa x纤维母细胞CGL1和CGL3细胞株间的差异应答基因, 用SSH筛选出了用DD法未筛选出的p16基因, 但用DD-PCR和SSH技术均未发现的IAP基因, 却用寡核苷酸微阵列检出了.
随着人类基因组计划和微生物全基因组测序的逐渐完成及基因芯片技术的成熟, SSH与微阵列结合将促进差异基因的大规模快速筛选, 而且筛选到的特异序列可与相应的基因序列库中EST进行同源比较或通过计算机分析查找到基因的全序列, 进而推测、鉴定基因表达蛋白的功能, 进一步把基因克隆的研究推向基因功能的研究.
编辑: N/A
| 1. | Bautz EK, Reilly E. Gene-specific messenger RNA: isolation by the deletion method. Science. 1966;151: 328-330. [PubMed] [DOI] |
| 2. | Duguid JR, Dinauer MC. Library subtraction of in vitro cDNA libraries to identify differentially expressed genes in scrapie infection. Nucleic Acids Res. 1990;18: 2789-2792. [PubMed] [DOI] |
| 3. | Diatchenko L, Lau YF, Campbell AP, Chenchik A, Moqadam F, Huang B, Lukyanov S, Lukyanov K, Gurskaya N, Sverdlov ED. Suppression subtractive hybridization: a method for generating differentially regulated or tissue-specific cDNA probes and libraries. Proc Natl Acad Sci U S A. 1996;93: 6025-6030. [PubMed] [DOI] |
| 4. | Gurskaya NG, Diatchenko L, Chenchik A, Siebert PD, Khaspekov GL, Lukyanov KA, Vagner LL, Ermolaeva OD, Lukyanov SA, Sverdlov ED. Equalizing cDNA subtraction based on selective suppression of polymerase chain reaction: cloning of Jurkat cell transcripts induced by phytohemaglutinin and phorbol 12-myristate 13-acetate. Anal Biochem. 1996;240: 90-97. [PubMed] [DOI] |
| 5. | Ji W, Wright MB, Cai L, Flament A, Lindpaintner K. Efficacy of SSH PCR in isolating differentially expressed genes. BMC Genomics. 2002;3: 12. [PubMed] [DOI] |
| 6. | Winstanley C. Spot the difference: applications of subtractive hybridisation to the study of bacterial pathogens. J Med Microbiol. 2002;51: 459-467. [PubMed] [DOI] |
| 7. | Akopyants NS, Fradkov A, Diatchenko L, Hill JE, Siebert PD, Lukyanov SA, Sverdlov ED, Berg DE. PCR-based subtractive hybridization and differences in gene content among strains of Helicobacter pylori. Proc Natl Acad Sci USA. 1998;95: 13108-13113. [DOI] |
| 8. | Janke B, Dobrindt U, Hacker J, Blum-Oehler G. A subtractive hybridisation analysis of genomic differences between the uropathogenic E. coli strain 536 and the E. coli K-12 strain MG1655. FEMS Microbiol Lett. 2001;199: 61-66. [PubMed] [DOI] |
| 9. | Zhang YL, Ong CT, Leung KY. Molecular analysis of genetic differences between virulent and avirulent strains of Aeromonas hydrophila isolated from diseased fish. Microbiology. 2000;146 (Pt 4): 999-1009. [PubMed] [DOI] |
| 10. | Reckseidler SL, DeShazer D, Sokol PA, Woods DE. Detection of bacterial virulence genes by subtractive hybridization: identification of capsular polysaccharide of Burkholderia pseudomallei as a major virulence determinant. Infect Immun. 2001;69: 34-44. [DOI] |
| 11. | Lai YC, Yang SL, Peng HL, Chang HY. Identification of genes present specifically in a virulent strain of Klebsiella pneumoniae. Infect Immun. 2000;68: 7149-715. [DOI] |
| 12. | Han FC, Gong M, Ng HC, Ho B. Identification of H. pylori strain specific DNA sequences between two clinical isolates from NUD and gastric ulcer by SSH. World J Gastroenterol. 2003;9: 1747-1751. [PubMed] [DOI] |
| 13. | 徐 晓春, 瞿 逢伊, 宋 关鸿. 蚊媒感染疟原虫后应答性表达增高基因的富集和筛选. 中国寄生虫学与寄生虫病杂志. 2001;19: 325-329. |
| 14. | Patzwahl R, Meier V, Ramadori G, Mihm S. Enhanced expression of interferon-regulated genes in the liver of patients with chronic hepatitis C virus infection: detection by suppression-subtractive hybridization. J Virol. 2001;175: 1332-1338. [DOI] |
| 15. | Riemer C, Queck I, Simon D, Kurth R, Baier M. Identification of upregulated genes in scrapie-infected brain tissue. J Virol. 2000;74: 10245-10248. [PubMed] [DOI] |
| 16. | Dessens JT, Margos G, Rodriguez MC, Sinden RE. Identification of differentially regulated genes of Plasmodium by suppression subtractive hybridization. Parasitol Today. 2000;16: 354-356. [PubMed] [DOI] |
| 17. | Matuschewski K, Ross J, Brown SM, Kaiser K, Nussenzweig V, Kappe SH. Infectivity-associated changes in the transcriptional repertoire of the malaria parasite sporozoite stage. J Biol Chem. 2002;277: 41948-41953. [PubMed] [DOI] |
| 18. | Bellatin JA, Murray AS, Zhao M, McMaster WR. Leishmania mexicana: identification of genes that are preferentially expressed in amastigotes. Exp Parasitol. 2002;100: 44-53. [PubMed] [DOI] |
| 21. | Liu TX, Zhang JW, Tao J, Zhang RB, Zhang QH, Zhao CJ, Tong JH, Lanotte M, Waxman S, Chen SJ. Gene expression networks underlying retinoic acid-induced differentiation of acute promyelocytic leukemia cells. Blood. 2000;96: 1496-1504. |
| 22. | Li JY, Boado RJ, Pardridge WM. Blood-brain barrier genomics. J Cereb Blood Flow Metab. 2001;21: 61-68. [PubMed] [DOI] |