修回日期: 2003-12-01
接受日期: 2003-12-16
在线出版日期: 2004-04-15
目的: HCV NS5A病毒蛋白反式激活作用的新的靶基因NS5ATP2及其不同剪接体基因序列的确立、克隆化研究.
方法: 依据我室构建的NS5A反式激活基因差异表达的cDNA消减文库, 利用生物信息学技术获得, 提取HepG2细胞的总RNA, 进行反转录(RT-PCR), 扩增产物与原核表达载体连接, 进行测序鉴定.
结果: 经测序鉴定成功获得新基因的编码序列, 并意外发现了NS5ATP2的不同剪接体, 对NS5ATP2基因组进行分析, 获得剪接体的编码序列, 并成功进行了克隆化研究.
结论: 利用分子生物信息学技术, 发现并鉴定了HCV NS5A反式激活作用的新的靶基因NS5ATP2(615)及其可变剪接体NS5ATP2(216), 为研究新基因的生物学功能及丙肝发病机制提供新的依据.
引文著录: 杨倩, 成军, 刘妍, 洪源, 王建军, 张树林. 丙型肝炎病毒非结构蛋白NS5A反式激活基因2基因组DNA结构分析及其不同剪切体的克隆化研究. 世界华人消化杂志 2004; 12(4): 801-804
Revised: December 1, 2003
Accepted: December 16, 2003
Published online: April 15, 2004
AIM: To clone and identify a new gene NS5ATP2 and its spliced variant transactivated by hepatitis C virus non-structural protein 5A.
METHODS: On the base of subtractive cDNA library of genes transactivated by NS5A protein of hepatitis C virus, the coding sequence of new gene and its spliced variant were obtained by bioinformatics methods. Polymerase chain reaction (PCR) was conducted to amplify NS5ATP2 gene.
RESULTS: The coding sequence of new gene and its spliced variant were cloned and Identification successfully.
CONCLUSION: A novel gene has been recognized as the new target transactivated by HCV NS5A protein. These results bring some new clues for studying the biological functions of the new gene and pathogenesis of the viral proteins.
- Citation: Yang Q, Cheng J, Liu Y, Hong Y, Wang JJ, Zhang SL. Cloning and identification of NS5ATP2 gene and its spliced variant transactivated by hepatitis C virus non-structural protein 5A. Shijie Huaren Xiaohua Zazhi 2004; 12(4): 801-804
- URL: https://www.wjgnet.com/1009-3079/full/v12/i4/801.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i4.801
丙型肝炎病毒(HCV)是含外膜蛋白的单股正链RNA病毒, 属黄病毒科肝炎病毒类. 在大多数感染人群中丙型肝炎病毒表现为持续性感染, 并可导致慢性肝炎、肝硬化, 或肝细胞癌[1-4]. 在体外实验中证实NS5A对细胞周期及细胞生长具有调节作用, 可能与慢性肝炎、肝细胞癌的发生发展有着密切的关系[5-6]. 我室利用抑制性消减杂交技术, 对于NS5A表达载体转染的肝母细胞瘤细胞系HepG2的基因表达谱变化进行比较研究, 发现了NS5A蛋白可以反式激活一些基因的表达. 其中包括未知功能基因, 命名为NS5ATP2[7-10]. 我们对其进行了克隆化研究, 成功获得了NS5ATP2的编码序列, 在此过程中我们意外发现了NS5ATP2基因的不同剪切体, 对NS5ATP2基因组进行分析, 确定了该基因剪切体的编码序列, 并进行了克隆化研究.
肝母细胞瘤细胞系HepG2和感受态大肠杆菌DH5为本室保存, Taq酶购自Promega公司, pGEM-T载体购自Promega公司. 玻璃奶回收试剂盒购自博大公司. DNA序列测定由上海申友公司完成.
利用抑制性消减杂交技术构建NS5A反式激活基因差异表达的cDNA消减文库, 挑选90个克隆测序, 与GenBank数据库进行初步比较. 15个克隆未检索到任何对应的相似序列, 可能代表了某些新基因. 利用美国国立卫生院(NIH)国立医学图书馆(NLM)国立生物工程中心(NCBI)建立的核苷酸序列数据库(GenBank)及其同源基因序列的搜索(BLASTN), 发现其中一个新基因与GenBank中注册的已知功能基因序列没有同源. 电子拼接推定该基因的开放读码框架获得相应的全长编码基因, 将该未知功能基因命名为NS5ATP2. 对NS5ATP2进行电子拼接, 发现其开放读码框长615 bp, 编码207氨基酸残基[11-13].
根据NS5ATP2的全长编码基因, 设计上下游引物. 上游引物5'-GGA TTC ATG GCT TCG GTC TCC TCT GC-3', 下游引物5'-GGT ACC TCA GGA GTG TGG CTC ACT GG -3'. 以反转录产物为模板进行PCR, PCR参数如下: 94 ℃ 2 min预变性, 94 ℃ 60 s变性, 60 ℃ 60 s退火, 72 ℃ 60 s延伸, 共35个循环, 72 ℃再延伸10 min.
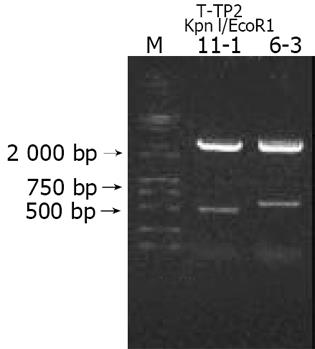
克隆目的片段 将PCR产物在0.9%琼脂糖凝胶中电泳, 切取目的片段, 玻璃奶法回收PCR产物, 与pGEM-T载体连接, 转化DH5感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养18 h. 挑取白色菌落, 增菌. 提取质粒进行限制性酶切分析鉴定, 发现两个大小不一致的酶切片段, 挑选相应菌落送测序.
将测序得到的两个核苷酸序列进行分析, 其中一个与我们预测的NS5ATP2序列完全一致, 而另一个序列与NS5ATP2部分同源, 利用美国国立卫生院(NIH)国立医学图书馆(NLM)国立生物工程中心(NCBI)建立的核苷酸序列数据库(GenBank)进行搜索, 确定了两个序列的染色体定位, 并对其基因组进行了分析.
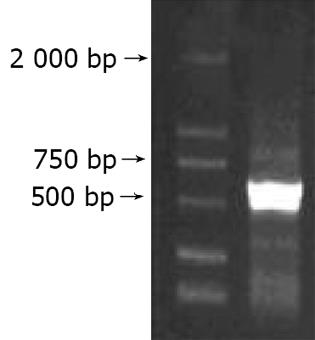
以HepG2细胞系cDNA文库为模板, PCR反应后经0.9%琼脂糖凝胶电泳, 可见长度为615 bp和500 bp的两种电泳条带, 见图1. PCR产物与T-载体连接, 转化大肠杆菌, 提取质粒进行酶切鉴定, 结果见图2.
将连接两个不同大小片段的质粒进行测序, 其中一个结果完全符合NS5ATP2的拼接序列, 编码序列全长为615 bp, 表明我们已顺利得到NS5ATP2基因编码序列. 同时我们测序得到另一个与NS5ATP2部分同源序列, 测序全长为513 bp, 利用生物信息学技术推断其编码序列为219 bp.
2.3 将NS5ATP2(615 bp)与NS5ATP2(513 bp)编码序列进行比较, 结果见图3:
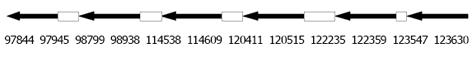
登录美国国立卫生院(NIH)国立医学图书馆(NLM)国立生物工程中心(NCBI)建立的核苷酸序列数据库(GenBank)(http://http://www.ncbi.nlm.nih.gov/), 确定NS5ATP2 (615 bp)与NS5ATP2(513 bp)的染色体定位, 并进行比较分析. 发现二者均定位于6q22.1-23.3, 但内含子和外显子结构不同, NS5ATP2(615 bp)基因位于123 630-97 844 nt, NS5ATP2 (513 bp) 基因位于123 630-122 235 nt, 见图4, 5:
人类基因组的精细完整序列图谱公布, 是人类生命科学史上的重要的里程碑, 基因组序列的阐明能从根本上影响我们对生物学的认识和对医学的实践. 利用基因组DNA序列可以为科研提供很多方面的方便, 这些基因组序列可以用来帮助寻找新基因[14]. 人类基因大约有25 000-35 000条, 通过与基因组序列的对比, 我们还能发现基因不同的剪切形式, 为进一步实验验证提供信息[15-18]. 一个基因可以通过如下几种方式产生多个转录体, 如不同的转录起始位点, 可变剪接, 选择不同的加尾信号位点, RNA编辑等. 可变剪接包括3种类型: (1) 内含子的保留; (2)可变外显子的保留或切除; (3) 3'-和5'-剪接位点的转移(shift)导致外显子的增长或缩短[19-21]. 可变剪接是从相对简单的基因组提高蛋白质组多样性的重要机制, 蛋白质组的多样性与多细胞高等生物的复杂性相适应. 从可变剪接涉及的基因分布格局分析, 可变剪接多发生在参与信号传导和表达调节等复杂过程的基因上, 如受体, 信号传导通路(凋亡), 转录因子等[22-26]. 对个体分化发育和一些关键的细胞生理过程如凋亡、细胞兴奋等的精确调控有重要意义[27-29]. 一个基因通过可变剪接产生多个转录异构体, 各个不同的转录异构体编码结构和功能不同的蛋白质, 他们分别在细胞/个体分化发育不同阶段, 在不同的组织, 有各自特异的表达和功能. 因此, 可变剪接是一种在转录后RNA水平调控基因表达的重要机制[30].
我室在进行病毒性肝炎发病机制的研究过程中, 利用抑制性消减杂交技术、酵母双杂交技术、酵母单杂交技术、基因芯片技术, 筛选出乙型肝炎、丙型肝炎病毒蛋白反式激活的新基因、与病毒蛋白-蛋白相结合的新基因, 如NS5A-TP4、NS5A-TP6、HCBP6、SPBP1等, 在对上述基因进行功能研究的过程中, 我们发现这些新基因与细胞信号传导、细胞生长、癌基因及抑癌基因的表达有着密切的联系, 为研究病毒性肝炎的发病机制提供了新的方向、新的线索[31-34]. NS5A是非结构蛋白的一种, 目前认为NS5A蛋白是在细胞蛋白激酶作用下发生磷酸化修饰后发挥生物调节作用的. 近年研究发现, NS5A是转录反式激活因子, 其羧基末端富含酸性氨基酸及脯氨酸, 这是真核细胞转录因子特有的结构特征. 尽管NS5A蛋白是存在于胞质中, 但其具有功能性核定位信号(nucleic localization signal, NLS)序列, 即PPRKKRVV(354-362 aa), 因此有核信号转导功能[35-36]. 为进一步研究其对细胞基因表达的调节作用, 我室利用抑制性消减杂交技术筛选出HCV NS5A反式激活基因差异表达的cDNA消减文库, 对挑选的克隆进行分析发现其中一个新基因与GenBank中注册的已知功能基因序列没有同源. 将该未知功能基因命名为NS5ATP2. 电子拼接推定该基因的开放读码框架获得相应的全长编码基因, TP2的ORF长度为615 bp, 编码204个氨基酸残基. 根据这一序列设计引物, 以人肝癌细胞cDNA为模板, PCR反应扩增TP2基因序列. 在NS5A-TP2基因的克隆化研究过程中, 随机挑选的两个克隆进行酶切鉴定时, 凝胶电泳得到600 bp和500 bp两个DNA片段, 将相应的克隆进行测序, 其中一个克隆序列与NS5A-TP2推定序列完全一致, 表明我们已顺利得到NS5A-TP2, 另一个克隆序列测序全长为513 bp与TP2部分同源, 登陆美国国立卫生院(NIH)国立医学图书馆(NLM)国立生物工程中心(NCBI)建立的核苷酸序列数据库(GenBank)(http://http://www.ncbi.nlm.nih.gov/), 确定NS5ATP2(615 bp)与NS5ATP2(513 bp)的染色体定位, 并进行比较分析. 发现二者均定位于6q22.1-23.3, NS5ATP2(615 bp)基因位于123 630-97 844 nt, NS5ATP2(513 bp) 基因位于123 630-122 235 nt, NS5ATP2(615 bp)与NS5ATP2 (513 bp)是由同一基因编码的不同剪切体. 对NS5ATP2 (513 bp)进行生物信息学分析推定该基因的开放读码框架为216 bp, 编码72个氨基酸, 我们根据其开放读码框架将NS5ATP2 (513 bp)重新命名为NS5ATP2 (216 bp). 得到NS5ATP2基因及其剪切体的编码序列, 是该基因的生物学研究的第一步, 对其进行深入的研究为丙型肝炎的发病机制的研究提供了新的研究方向[37].
编辑: N/A
| 1. | 成 军, 杨 守纯. 现代肝炎病毒分子生物学. 第1版. 北京: 人民军医出版社 1997; 83-88. |
| 2. | 成 军. 丙型肝炎病毒基因组的翻译及其产物的加工. 国外医学微生物学分册. 1995;18:14-16. |
| 4. | 成 军, 张 玲霞. 抗HCV的基因治疗方案-从HCV-肝细胞相互作用的分子生物学机制设计. 国外医学. 流行病学传染病学分册. 1999;26:59-61. |
| 5. | Gong GZ, Waris G, Tanveer R. Human hepatitis C virus NS5A protein alters intracellular calcium levels, induces oxidative stress, and activates STAT-3 and NF-B. Proc Natl Acad Sci USA. 2001;98:9599-9604. [PubMed] [DOI] |
| 6. | Ghosh AK, Steele R, Meyer K. Hepatitis C virus NS5A protein modulates cell cycle regulatory genes and promotes cell growth. J Gen Virol. 1999;80:1179-1183. [PubMed] [DOI] |
| 7. | Diachenko L, Lau YC, Campbell AP, Chenchik A, Moqadam F, Huang B, Lukyanov S, Lukyanov K, Gurskaya N, Sverdlov ED. Suppression subtractive hybridization: a method for generating differentially regulated or tissue-specific cDNA probes and libraries. Proc Natl Acad Sci USA. 1996;93:6025-6030. [PubMed] [DOI] |
| 9. | 刘 妍, 成 军, 王 刚, 李 克, 段 惠娟, 王 琳, 董 菁, 洪 源, 张 跃新, 李 莉. 应用抑制性消减杂交技术克隆丙型肝炎病毒核心蛋白反式激活基因. 解放军医学杂志. 2001;26:880-883. |
| 13. | 杨 倩, 刘 妍, 成 军, 王 建军, 王 春花, 纪 冬, 党 晓燕, 张 树林. 丙型肝炎病毒非结构蛋白5A反式激活基因4的克隆化研究. 胃肠病学和肝病学杂志. 2003;12:258-250. |
| 14. | Mount SM. Genomic sequence, splicing, and gene annotation. Am J Hum Genet. 2000;67:788-792. [PubMed] [DOI] |
| 15. | Aznarez I, Chan EM, Zielenski J, Blencowe BJ, Tsui LC. Characterization of disease-associated mutations affecting an exonic splicing enhancer and two cryptic splice sites in exon 13 of the cystic fibrosis transmembrane conductance regulator gene. Hum Mol Genet. 2003;12:2031-2040. [PubMed] [DOI] |
| 16. | Pennacchio LA. Insights from human/mouse genome comparisons. Mamm Genome. 2003;14:429-436. [PubMed] [DOI] |
| 17. | Austin CP. The completed human genome: implications for chemical biology. Curr Opin Chem Biol. 2003;7:511-515. [PubMed] [DOI] |
| 18. | Tran P, Leclerc D, Chan M, Pai A, Hiou-Tim F, Wu Q, Goyette P, Artigas C, Milos R, Rozen R. Multiple transcription start sites and alternative splicing in the methylenetetrahydrofolate reductase gene result in two enzyme isoforms. Mamm Genome. 2002;13:483-492. [PubMed] [DOI] |
| 19. | Slaugenhaupt SA. Tissue-specific expression of a splicing mutation in the IKBKAP gene. Clin Auton Res. 2002;12:I15-I19. [PubMed] [DOI] |
| 20. | Stiles JK, Hicock PI, Shah PH, Meade JC. Genomic organization, transcription, splicing and gene regulation in Leishmania. Ann Trop Med Parasitol. 1999;93:781-807. [PubMed] [DOI] |
| 21. | O'Neill JP, Rogan PK, Cariello N, Nicklas JA. Mutations that alter RNA splicing of the human HPRT gene: a review of the spectrum. Mutat Res. 1998;411:179-214. [PubMed] [DOI] |
| 22. | Verma M, Kurl RN, Blass C, Davidson EA. RNA-RNA interaction and gene splicing. Cancer Biochem Biophys. 1997;15:211-220. [PubMed] |
| 23. | Kornblihtt AR, Pesce CG, Alonso CR, Cramer P, Srebrow A, Werbajh S, Muro AF. The fibronectin gene as a model for splicing and transcription studies. FASEB J. 1996;10:248-257. [PubMed] |
| 24. | Smith CW, Patton JG, Nadal-Ginard B. Alternative splicing in the control of gene expression. Annu Rev Genet. 1989;23:527-577. [PubMed] [DOI] |
| 25. | Benjamin PR, Burke JF. Alternative mRNA splicing of the FMRFamide gene and its role inneuropeptidergic signalling in a defined neural network. Bioessays. 1994;16:335-342. [PubMed] [DOI] |
| 26. | Jiang Z, Tang H, Havlioglu N, Zhang X, Stamm S, Yan R, Wu JY. Mutations in tau gene exon 10 associated with FTDP-17 alter the activity of an exonic splicing enhancer to interact with Tra2 beta. J Biol Chem. 2003;278:18997-19007. [PubMed] [DOI] |
| 27. | Wang-Johanning F, Frost AR, Jian B, Epp L, Lu DW, Johanning GL. Quantitation of HERV-K env gene expression and splicing in human breast cancer. Oncogene. 2003;22:1528-1535. [PubMed] [DOI] |
| 28. | Logette E, Wotawa A, Solier S, Desoche L, Solary E, Corcos L. The human caspase-2 gene: alternative promoters, pre-mRNA splicing and AUG usage direct isoform-specific expression. Oncogene. 2003;22:935-946. [PubMed] [DOI] |
| 29. | Trott JF, Hovey RC, Koduri S, Vonderhaar BK. Alternative splicing to exon 11 of human prolactin receptor gene results in multiple isoforms including a secreted prolactin-binding protein. J Mol Endocrinol. 2003;30:31-47. [PubMed] [DOI] |
| 30. | Ezquieta B, Cueva E, Oyarzabal M, Oliver A, Varela JM, Jariego C. Gene conversion (655G splicing mutation) and the founder effect (Gln318Stop) contribute to the most frequent severe point mutations in congenital adrenal hyperplasia (21-hydroxylase deficiency) in the Spanish population. Clin Genet. 2002;62:181-188. [PubMed] [DOI] |
| 31. | 李 克, 王 琳, 成 军, 张 玲霞, 段 惠娟, 陆 荫英, 杨 继珍, 刘 妍, 邵 得志, 夏 小兵. 筛选与克隆丙型肝炎病毒核心蛋白结合蛋白6基因. 中华实验和临床病毒学杂志. 2002;16:51-54. |
| 33. | 刘 妍, 成 军, 张 跃新, 段 惠娟, 牟 劲松, 韩 萍, 李 莉, 张 玲霞, 陈 菊梅. 截短型HBsAg中蛋白反式激活基因的克隆. 中华传染病杂志. 2002;20:218-221. |
| 34. | 杨 倩, 刘 妍, 成 军, 王 建军, 杨 艳杰, 张 树林. 丙型肝炎病毒核心蛋白上调层粘连蛋白B1链基因启动子表达活性的研究. 世界华人消化杂志. 2003;11:955-958. [DOI] |