修回日期: 2003-12-01
接受日期: 2003-12-16
在线出版日期: 2004-04-15
目的: 探讨乙型肝炎病毒(HBV)基因组是否存在前-X编码基因, 并探讨前-X基因与HBV基因型之间的关系.
方法: 自慢性乙型肝炎患者血清中提取乙型肝炎病毒基因组, 首先应用多引物-多聚酶链反应(PCR)进行HBV基因型分型. 之后将前-X区扩增, TA克隆到pGEM Teasy载体后进行单克隆测序, 利用Vector 8.0软件进行序列分析.
结果: 17例患者中, A型者1例, B型3例, B/C混合型者3例, C型10例. 克隆测序的45个克隆中, 27个(60%) 克隆编码前-X多肽, 其中18个(66.7%)克隆来自C基因型. 有3种形式突变导致前-X区编码不能. 前-X区多肽具有个体化突变现象.
结论: 前-X区编码在B、C基因型中均为普遍存在的现象, 其编码具有C型倾向性.
引文著录: 董菁, 杨倩, 成军. 型肝炎病毒基因组前-X区的分子流行病学研究. 世界华人消化杂志 2004; 12(4): 794-800
Revised: December 1, 2003
Accepted: December 16, 2003
Published online: April 15, 2004
AIM: To identify pre-X region in hepatitis B virus (HBV) genome and to study the relationship between the genotype and the existence of pre-X region.
METHODS: HBV genomic DNA was extracted from the sera of patients with HBV infection. Multiple-primers polymerase chain reaction (PCR) method was used to identify the genotype of HBV strains. Pre-X region was amplified by PCR method, and TA cloning technique was used to clone the DNA fragment into pGEM Teasy vector. After sequencing of the target region, Vector 8.0 software was used to analysis the sequences.
RESULTS: Seventeen samples were collected in this study. One genotype A, 3 genotype B, 10 genotype C and 3 B/C genotype mixture were identified. After sequencing, 27 clones from 45 clones (60%) were found encoding the pre-X peptide. Eighteen from 27 clones (66.7%) pre-X coding sequences were found from C genotype. Three types of replacement mutation led to pre-mature coding of pre-X gene. The mutation in pre-X peptide had feature of individually mutation.
CONCLUSION: Coding of the pre-X gene is popular in HBV genome.
- Citation: Dong J, Yang Q, Cheng J. Molecular epidemiological features of pre-X region in hepatitis B virus genome. Shijie Huaren Xiaohua Zazhi 2004; 12(4): 794-800
- URL: https://www.wjgnet.com/1009-3079/full/v12/i4/794.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i4.794
Gelibertet al[1-2]于1979年第一次解读了乙型肝炎病毒(HBV)基因组, 全长在3 200个核苷酸(nt)左右, 为部分双链DNA病毒. 之后学者[3-4]将HBV基因组划分出4个开放读码框架(ORF), 分别命名为S、C、P、X区. 4个ORF中表达的氨基酸长度不同, S基因编码HBV囊膜蛋白, 包裹HBV病毒; C基因编码核心蛋白, 系HBV基因组的附着部位; P基因编码多聚酶, 在基因组复制过程中起重要作用; X基因编码产物的功能不明确, 研究发现具有反式激活作用[5-6], 可激活宿主或病毒的基因表达. 我国学者[7-8]于1984年报道了中国大陆HBV全序列, 近来我们利用长距离-精确多聚酶链反应(LA-PCR)方法[9]研究了乙型肝炎患者血清中存在的HBV病毒基因组, 确定了前-前-S基因[10]和前-X基因[11]的以及上述2个基因的启动子[12-13], 并以文献流行病学方法确定了前-前-S基因和前-X基因的普遍性[14]. 本文以分子流行病学实验方法证实了前-X区的存在状态.
1.1.1 血清采集: 血清来源: 17例患者, 诊断为病毒性肝炎, 乙型, 慢性, 符合中华医学会传染病与寄生虫病学分会、中华医学会肝病学分会2000年联合制定的《病毒性肝炎防治方案》(试行)[15], 临床初步检测HBV DNA阳性.
1.1.2 DNA提取: 自静脉中采集5 mL全血, 分离血清, 200 L血清加入蛋白酶K及其缓冲液, 65 ℃消化过夜, 饱和酚: 氯仿(1: 1)抽提总DNA, 无水乙醇沉淀, 加入无DNA酶的RNA酶消化后, -20 ℃保存备用.
1.2.1 HBV基因型分析: 参考Naitoet al[16]设立的6种主要基因型分型方法, 建立HBV基因型的检测方法. 简言之, 应用巢式-多引物-多聚酶链反应, 外引物: 上游引物序列: 5'- AGC ATG GGA GGT TGG TCT TC-3', 下游引物序列: 5'- AAG GCA TCA AGG CAG GAT AGC-3', 目的片段长度约1 429 bp, 自前-S1区起始处至S区终止子下游226 bp. 内引物: 上游引物序列: 5'- GGC TCA AGT TCC GGA ACA GT-3', A基因型特异性下游引物: 5'- CTC GCG GAG ATT GAC GAG ATG T-3', B基因型特异性下游引物: 5'-CAG GTT GGT GAG TGA CTG GAG A-3', C基因型特异性下游引物: 5'-GGT CCT AGG AAT CCT GAT GTT G-3'.
外引物扩增PCR程序参数如下: 94 ℃ 1 min预变性, 94 ℃ 1 min 30 s变性, 59 ℃ 1 min 30 s退火, 72 ℃ 2 min延长, 共30个循环, 72 ℃延长10 min. 内引物扩增PCR程序参数如下: 94 ℃ 1 min预变性, 94 ℃ 30 s变性, 58 ℃ 30 s退火, 72 ℃ 30 s延长, 共35个循环. NT-PCR产物经过3%琼脂糖凝胶电泳后判断HBV基因型.
1.2.2 前-X区的克隆及序列分析: 采用本研究组以往报告[17-19]的PCR-TA克隆-测序方法. PCR扩增前-X区基因: 前-X上游引物为P1: 5'- CCA AGT GTT TGC TGA CGC AAC C', 下游引物为P2: 5'- GGA TCC AGT TGG CAG CAC ACC-3', P1下游第31-33 nt处为前-X区起始密码子ATG, P2上游30-32 nt处为前S1区起始密码子ATG. 前-X区PCR参数如下: 94 ℃ 1 min预变性, 94 ℃ 60 s变性, 58 ℃ 60 s退火, 72 ℃ 60 s延长, 共35个循环, 72 ℃再延长10 min.
克隆目的片段: 将PCR产物在1%琼脂糖凝胶中电泳, 切取目的片段, 玻璃奶法回收PCR产物, 与Promega公司所产的pGEM Teasy载体连接过夜. 将连接好的重组质粒转入细菌JM109, 氨苄青霉素(Amp)和X-gal蓝白斑法筛选阳性菌落, 进一步提取质粒鉴定. DNA测序: 选择经鉴定pGEM Teasy内插入230 bp产物的菌落送检测序, 由上海博亚公司完成. 测序引物为pGEM Teasy载体自有的T7、SP6测序引物.
1.2.3 序列分析: 应用Vector 8.0版软件对进行了测序分析的HBV前-X基因序列进行碱基序列和氨基酸序列一致性分析. 该软件比较后提供两个数据: 阳性率和一致率, 阳性率是选定区域推定序列的核苷酸(氨基酸)序列数目与区域总长度之比, 推定序列是软件自动比较所有序列, 参考每个核苷酸(氨基酸)位点上每个克隆的编码方式, 由大多数克隆(多于70%)编码的核苷酸(氨基酸)组成的一致性序列, 阳性率用于展示区域内部的插入突变或/和缺失突变, 以及较少见的单一位点多种核苷酸(氨基酸)编码方式; 一致率是选定区域全部克隆均为一致的核苷酸(氨基酸)数目与最长的单一克隆核苷酸(氨基酸)序列数目之比, 表示该段区域核苷酸(氨基酸)序列一致性, 用于展示区域内部的替换突变或/和缺失突变.
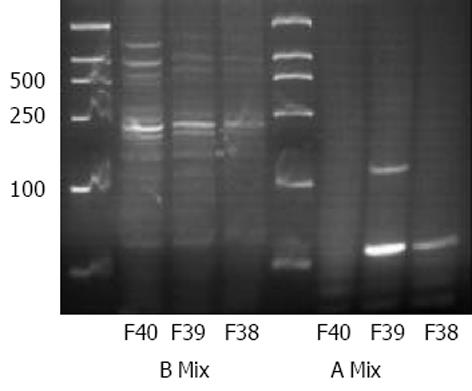
获得的内引物PCR产物电泳结果见图1.
应用该基因型分型方法, 电泳时展示的HBV A基因型PCR产物长度为68 bp, B型为281 bp, C型为122 bp. 本研究的17例患者中, 基因型为A型者1例, 基因型为B型者3例, B/C混合型者3例, C型10例.
前-X基因全长168 bp, 45株核苷酸序列比较见图2. 本研究扩增的靶区域全长231 nt, 包括前-X基因上游30 nt和下游X基因30 nt. 靶区域除去引物区全长187 bp, 比较结果提示阳性率为99.5%, 一致率为78.1%, 其中154 nt编码方式有A/G/C3种; 前-X基因编码框架自第31至第198 nt, 阳性率为99.4%, 一致率为76.8%; 在序列比较中, 我们发现来自同一患者体内的克隆之间的一致性大于95%, 符合准种表现.
图2显示了所有的45个克隆前-X区的核苷酸序列, 以及由这45个克隆推断出的一致性序列. 这45个克隆中, 有27个克隆编码前-X多肽, 占观察序列的60%. 导致前-X基因编码不能的原因在于A2608→C/T替换突变或C/A2733→T替换突变或上述位点的双突变[14], 研究结果提示双替换突变是导致前-X区编码不能的主要原因, 占22.2%.
前-X基因的编码与否与基因型之间的关系见表1. 研究提示A型仅有1个克隆, 是由于双替换突变导致了编码失败; B型8个克隆中, 6个克隆是由于双替换突变导致了编码失败; C型26个克隆中, 5个克隆由于A2608→C/T替换突变, 3个克隆由于C/A2733→T替换突变导致了编码失败. C型测序的26个克隆中有18个克隆编码前-X区, 占69.2%; B型测序的8个克隆中仅有2个克隆编码前-X区, 占25%. 选择测序的45个克隆中有27个克隆编码前-X区, 其中B型2个, C型18 个, B/C混合型7例.
| A2608→C/T替换突变 | C/A2733→T替换突变 | 双替换突变 | 编码前-X区 | |
| A型 | 0 | 0 | 1 | 0 |
| B型 | 0 | 0 | 6 | 2 |
| C型 | 5 | 3 | 0 | 18 |
| B/C | 0 | 0 | 3 | 7 |
| 比例% | 11.1 | 6.7 | 22.2 | 60 |
值得注意的是患者F298, 共选择了4个克隆进行测序, 其中克隆F298-1发生A2608→C替换突变, 导致前-X区编码不能, 其他3个克隆未发生替换突变.
45株克隆中有27个克隆编码的前-X多肽, 其氨基酸序列比较见图3. 前-X多肽全长56 aa, 总一致率为73.2%. 经过比较, 按照一致率的差异分为3部分, 1-10 aa为高变区, 一致率为60.0%; 11-36 aa为高保守区, 一致率为88.5%, 其中19-36 aa是保守区中的保守区, 一致率为94.4%; 37-56 aa为另一高变区, 一致率为60.0%.
氨基酸序列中的部分替换突变具有来源个体化现象, 我们[20]曾命名其为HBV个体化突变.第26 aa, 来自其他患者的病毒株均编码P, 而来自患者Y931的2个克隆编码S; 相应的, 第40 aa, 来自其他患者的病毒株均编码Y, 而来自患者Y983的2个克隆编码H.
氨基酸的变异还表现为一些位点的变异具有相对的普遍意义, 比如第5年a的Y(10)/H(17)(括号中为编码该氨基酸残基的克隆数, 下同), 第6 aa的W(12)/R(15), 第37 aa的G(11)/S(16), 第38 aa的E(15)/K(12), 第42 aa的N(14)/H(13).
本研究组[21-29]在近期的研究中着重阐述了HBV准种现象, 在此基础上, 我们发现以往界定的HBV前-S1基因之前存在一个ORF, 将其命名为前-前-S基因[10]. 同时发现X基因之前存在一融合表达的ORF, 命名为前-X区[11], 为了证实前-X基因的实际存在以及氨基酸序列特点, 我们采取横断面研究方式对慢性乙型肝炎患者体内感染的HBV基因组进行了研究.
本研究选择了17例HBV DNA阳性的患者, 按照Naito et al[13]建立的快速分型鉴定方案进行了HBV基因型分型研究, 试图探讨HBV基因型别与前-X区编码之间的关系. 经过分型研究, 本研究抽取的17例患者中, 基因型为A型者1例, 基因型为B型者3例, B/C混合型者3例, C型10例. 自17例患者的克隆库中随机选择了共45个克隆进行核酸序列测定, 其中27个克隆编码前-X区, 18个克隆不编码前-X基因. 编码前-X基因的克隆中以C基因型为最多, 有18个克隆, 来自7例患者; 其中B型2个克隆, 来自1例患者; B/C混合型7个克隆, 来自2例患者. 10例患者体内的HBV病毒基因组编码前-X基因, 占采样总数的58.8%(10/17), 提示多数患者体内的HBV基因组编码前-X基因. 进行HBV基因型分型的研究试图阐明前-X基因的存在与HBV基因型之间的关系, 结果发现C型测序的26个克隆中有18个(69.2%)克隆编码前-X区, B型测序的8个克隆中仅有2个(25%)克隆编码前-X区, 提示前-X基因的编码具有一定的HBV C基因型倾向性, 但并不具有基因型特异性.
我们[14]之前的研究发现前-X基因编码不能主要由于3种突变所导致, 即A2608→C/T替换突变、C/A2733→T替换突变以及上述2个位点的双替换突变. 第一种变异导致前-X区起始密码子ATG突变为C/TTG, 病毒X蛋白自动以下一个ATG为起始密码子; 第二种变异导致前-X区ATG与原X区之间ATG之间第44位氨基酸编码子发生终止突变, C/AGA突变为TGA, 导致前-X基因表达被提前终止; 双替换突变是指上述的2种突变同时发生. 表1初步揭示了HBV基因型与前-X基因编码不能之间的关系. 结果发现A基因型1个克隆、B基因型6个克隆和B/C混合型3个克隆发生了双替换突变, 而C基因型前-X区编码不能的8个克隆均表现为单位点替换突变, 其中A2608→C/T突变5个, C/A2733→T突变3个, 本组研究结果初步提示C基因型HBV基因组与其他基因型导致前-X编码不能的机制是不同的, 相对而言, C基因型的变异是初步的, 而A、B基因型的变异是进一步的. 值得注意的是患者F298, 在选择的4个测序克隆中, 其中1个克隆发生A2608→C替换突变而导致前-X区编码不能, 其他3个克隆未发生替换突变, 这提示前-X区变异是渐变的, 并不是一成不变的.
针对前-X多肽氨基酸序列进行的分析发现前-X多肽存在2个高度变异区和一个高度保守区. 前-X多肽全长56 aa, 氨基酸序列总一致率为73.2%. 高度变异区存在于肽段的两端, 分别为1-10 aa, 一致率为60.0%; 37-56 aa为另一高变区, 一致率为60.0%. 上述2个区域的一致率较前-X多肽的总一致率低13.2%. 前-X多肽的中央部分为高保守区, 11-36 aa段一致率为88.5%, 19-36 aa的一致率高达94.4%. 这些发现对以后前-X多肽的检测有重要意义. 前-X多肽之中的变异以替换突变为主, 而之中部分位点的变异展示出HBV个体化突变特征[20]. 如第26 aa, 来自其他患者的病毒株均编码P, 而来自患者Y931的2个克隆编码S; 第40 aa, 来自其他患者的病毒株均编码Y, 而来自患者Y983的2个克隆编码H, 这些变异提示HBV基因组的变异就象人类的指纹, 这在HBV生活史中具有什么样的意义, 还需要进一步研究.
前-X区多肽氨基酸的变异还表现为一些位点的变异具有相对的普遍意义, 比如第5年a的Y(10)/H(17)(括号中为编码该氨基酸残基的克隆数, 下同), 第6 aa的W(12)/R(15), 第37 aa的G(11)/S(16), 第38 aa的E(15)/K(12), 第42 aa的N(14)/H(13). 这些位点的变化可能具有进一步分型的意义, 需要进一步研究.
总之, 本研究应用分子流行病学研究方法, 自17例患者的血清中提取了HBV DNA基因组, 之后应用HBV基因型分型技术和PCR-TA克隆-测序技术展示了前-X基因的存在, 这些样本来自普通HBV感染者, 而不是来自原发性肝癌患者[30-31], 提示前-X基因的存在是一种普遍现象.结合本研究组针对前-X基因的研究[11,13-14], 我们认为应当加强对前-X区的研究, 将Loncarevic et al[32]最初的研究提高到蛋白水平. 本研究还提示前-X基因存在HBV C基因型倾向性, 但不存在基因型的特异性.
编辑: N/A
| 1. | Galibert F, Mandart E, Fitoussi F, Tiollais P, Charnay P. Nucleotide sequence of the hepatitis B virus genome (subtype ayw) cloned in E. coli. Nature. 1979;281:646-650. [PubMed] [DOI] |
| 2. | Charnay P, Mandart E, Hampe A, Fitoussi F, Tiollais P, Galibert F. Localization on the viral genome and nucleotide sequence of the gene coding for the two major polypeptides of the hepatitis B surface antigen (HBs Ag). Nucleic Acids Res. 1979;7:335-346. [PubMed] [DOI] |
| 4. | Kumar U, Jayasuryan N, Kumar R. A truncated mutant (residues 58-140) of the hepatitis B virus X protein transactivitation function. Proc Natl Acad Sci USA. 1996;93:5647-5652. [PubMed] [DOI] |
| 5. | 刘 妍, 董 菁, 成 军, 夏 小兵, 李 克, 王 琳, 施 双双, 段 惠娟, 杨 继珍. 乙肝病毒X基因在真核细胞中的表达及反式激活SV40病毒 早期启动子的研究. 解放军医学杂志. 2001;26:404-406. |
| 8. | 董 菁, 李 进, 施 双双, 皇甫 竞坤, 成 军, 王 勤环, 洪 源, 王 业东, 李 莉, 斯 崇文. 乙型肝炎病毒基因组准种与变异特点的研究. 解放军医学杂志. 2002;27:116-118. |
| 11. | 杨 倩, 董 菁, 成 军, 刘 妍, 洪 源, 王 建军, 张 树林. 乙型肝炎病毒基因组中前-前-S-编码基因启动子序列的确定及转录活性的鉴定. 解放军医学杂志. 2003;28:761-762. |
| 15. | Naito H, Hayashi S, Abe K. Rapid and specific genotyping system for hepatitis B virus corresponding to six major genotypes by PCR using type-specific primers. J Clin Microbiol. 2001;39:362-364. [DOI] |
| 16. | 董 菁, 成 军, 王 勤环, 皇甫 竞坤, 施 双双, 张 国庆, 洪 源, 李 莉, 斯 崇文. 乙型肝炎病毒DNA序列异质性及准种特点的研究. 中华医学杂志. 2002;82:81-85. |
| 17. | Dong J, Cheng J, Wang QH, Liu Y, Wang G, Shi SS, Xia XB, Shao Q, Si CW. The preliminary study on hepatitis B virus (HBV) quasispecies in patients with chronic HBV infection. Chin J Infect Dis. 2001;19:199-203. |
| 18. | 董 菁, 成 军, 王 勤环, 施 双双, 皇甫 竞坤, 王 刚, 洪 源, 李 莉, 斯 崇文. 乙型肝炎病毒前C/C基因准种与变异特点的研究. 解放军医学杂志. 2002;27:122-123. |
| 21. | 董 菁, 施 双双, 张 国庆, 皇甫 竞坤, 洪 源, 成 军, 王 勤环, 李 莉, 斯 崇文. 乙型肝炎病毒C基因启动子区异质性检测的初步研究. 临床检验杂志. 2002;20:72-74. |
| 22. | 董 菁, 施 双双, 张 国庆, 皇甫 竞坤, 成 军, 王 勤环, 李 莉. 乙型肝炎病毒表面抗原/抗体同时阳性患者体内S基因序列的分析研究. 中国公共卫生. 2002;18:535-537. |
| 23. | 董 菁, 成 军, 王 勤环, 施 双双, 洪 源, 皇甫 竞坤, 王 刚, 李 莉, 斯 崇文. 乙型肝炎病毒逆转录酶区基因序列准种与变异研究. 解放军医学杂志. 2001;26:823-825. |
| 28. | 董 菁, 刘 妍, 皇甫 竞坤, 施 双双, 王 刚, 洪 源, 陈 国凤, 李 莉, 陈 菊梅, 成 军. 乙型肝炎病毒表面抗原基因多态性的初步研究. 胃肠病学和肝病学杂志. 2002;11:130-135. |
| 29. | Takahashi K, Kishimoto S, Ohori K, Yoshizawa H, Akahane Y, Okamoto H, Mishiro S. A unique set of mutations in the "preX" region of hepatitis B virus DNA frequently found in patients but not in asymptomatic carriers: implication for a novel variant. Int Hepatol Commun. 1995;3:131-138. [DOI] |
| 30. | Takahashi K, Akahane Y, Hino K, Ohta Y, Mishiro S. Hepatitis B virus genomic sequence in the circulation of hepatocellular carcinoma patients: comparative analysis of 40 full-length isolates. Arch Virol. 1998;143:2313-2326. [PubMed] [DOI] |
| 31. | Loncarevic IF, Zentgraf H, Schroder CH. Sequence of a replication competent hepatitis B virus genome with a preX open reading frame. Nucleic Acids Res. 1990;18:4940. [PubMed] [DOI] |