修回日期: 2003-08-10
接受日期: 2003-08-16
在线出版日期: 2004-02-15
目的: 应用基因芯片技术, 检测丙型肝炎病毒(HCV)非结构蛋白5A(NS5A)反式激活基因NS5ATP3的表达对肝母细胞瘤细胞HepG2基因表达谱的影响, 进一步阐明NS5ATP3蛋白可能的分子生物学功能.
方法: 设计并合成NS5ATP3基因序列特异性的引物, 应用聚合酶链反应(PCR)技术扩增NS5ATP3蛋白编码基因片段, 以常规的分子生物学技术将获得的NS5ATP3编码基因片段克隆到TA载体中进行核苷酸序列的测定, 构建真核表达载体pcDNA3.1(-)-NS5ATP3. 以脂质体转染肝母细胞瘤细胞系HepG2, 提取mRNA, 逆转录为cDNA, 与转染空白表达载体pcDNA3.1(-)的HepG2细胞进行cDNA芯片分析.
结果: 构建的表达载体经过限制性内切酶分析和DNA序列测定, 证实准确无误. 提取高质量的mRNA, 逆转录为cDNA, 进行DNA芯片技术分析. 在1 152个基因表达谱的筛选中, 发现有6个基因表达水平显著上调, 18个基因表达水平显著下调.
结论: 应用基因表达谱芯片技术成功筛选了NS5ATP3转染细胞后差异表达基因, 为进一步阐明NS5ATP3蛋白可能的生物学功能提供依据.
引文著录: 刘妍, 杨倩, 成军, 王建军, 纪冬, 党晓燕, 王春花. 基因表达谱芯片筛选NS5ATP3转染细胞差异表达基因. 世界华人消化杂志 2004; 12(2): 306-310
Revised: August 10, 2003
Accepted: August 16, 2003
Published online: February 15, 2004
AIM: NS5ATP3 obtained from suppression subtractive hybridization screeening is a novel gene transactivated by nonstructural protein 5A (NS5A) of hepatitis C virus (HCV), which possesses unknown function. To study the difference in gene expression in human hepatoblastoma cell line HepG2 cells transfected with NS5ATP3-expressing plasmid and further elucidate its potential molecular biological function, we compared the differentially expressed genes between the HepG2 transfected by pcDNA3.1(-)-NS5ATP3 and pcDNA3.1(-), respectively by cDNA microarray technique.
METHODS: Sequence specific primers were designed and synthesized and the NS5ATP3 DNA fragment was amplified with polymerase chain reaction (PCR) technique. The expressive vector of pcDNA3.1(-)-NS5ATP3 was constructed by routine molecular biological methods. cDNA microarray technology was employed to detect the mRNA from the HepG2 cells transfected with pcDNA3.1(-)-NS5ATP3 and pcDNA3.1(-), respectively using lipofectamine.
RESULTS: The expressive vector has been constructed and confirmed by restriction enzyme digestion and DNA sequencing analysis. High quality mRNA and cDNA were prepared and successful microarray screening was conducted. The scanning results indicated that among 1 152 genes which were gotten from gene expression profile analysis, there were 21 differences in which 6 genes were up-regulated and 18 genes were down-regulated in NS5ATP3-expressing HepG2 cells. These genes differentially regulated by NS5ATP3 included human genes encoding proteins involved in cell signal transduction, cell apoptosis, cell proliferation and differentiation.
CONCLUSION: cDNA microarray technology is successfully used to screen the genes differentially expressed in NS5ATP3-expressing HepG2 cells, which brings some new clues for studying the potential molecular mechanism of NS5ATP3 protein.
- Citation: Liu Y, Yang Q, Cheng J, Wang JJ, Ji D, Dang XY, Wang CH. Screening of genes differentially expressed in HepG2 cells transfected with gene 3 transactivated by hepatitis C virus nonstructural protein 5A (NS5ATP3) using cDNA microarray. Shijie Huaren Xiaohua Zazhi 2004; 12(2): 306-310
- URL: https://www.wjgnet.com/1009-3079/full/v12/i2/306.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i2.306
丙型肝炎病毒(HCV)感染, 与肝纤维化和肝细胞癌(he-patocellular carcinoma, HCC)发生发展过程密切相关, 而病毒基因组编码的蛋白与宿主肝细胞蛋白之间的相互作用, 可能是HCV致病(癌)的重要分子机制. 研究发现, HCV非结构蛋白5A(HCV NS5A)基因(位于6 258-7 601 nt之间)编码的56 kD的NS5A蛋白(448 aa)除了参与HCV多蛋白的成熟和RNA的复制过程外, 还具有多种生物学功能. 不同磷酸化形式的NS5A蛋白具有反式激活作用更是目前关于NS5A生物学功能研究的热点, NS5A蛋白是一种作用很强的转录激活因子, 能够影响细胞信号转导途径, 激活多种病毒及细胞基因启动子, 调控着细胞基因的转录, 推测NS5A蛋白的这种反式激活作用, 可能是HCV感染与肝细胞癌发生发展密切相关的重要分子生物学机制[1-3]. 本室在利用抑制性消减杂交技术(suppression subtractive hybridization, SSH)筛选NS5A蛋白反式激活作用的部分靶基因时, 结合生物信息学技术(bioinformatics)克隆了NS5A蛋白反式激活作用的新靶基因, 即HCV NS5A蛋白反式激活基因3(NS5ATP3), 该新基因的开放读码框架(ORF)长度为1 572个核苷酸(nt), 编码产物由524个氨基酸残基(aa)组成[4-6].
为了探索NS5ATP3的生物学功能, 深入了解HCV NS5A蛋白的反式激活作用, 我们构建了NS5ATP3基因真核表达载体, 应用基因表达谱芯片技术, 筛选NS5ATP3基因转染细胞后差异表达的基因, 检测NS5ATP3蛋白的表达对肝细胞基因表达谱的影响, 推测其在体内可能存在功能的线索, 为研究HCV的致病机制及探索未知基因的功能提供了新的方向.
肝母细胞瘤细胞系HepG2细胞由本室保存, 细胞培养相关试剂、总RNA提取试剂Trizol及真核表达载体pcDNA3.1(-)均购自Invitrogen公司. 人类基因组分类I芯片包括原癌基因和抑癌基因、免疫调节相关基因、细胞凋亡和应激反应蛋白相关基因、细胞信号转导相关基因等1 152个cDNA, 由上海联合基因有限公司提供. mRNA纯化试剂Oligotex mRNA Midi Kit 购自Qiagen公司.
1.2.1 真核表达载体构建及细胞转染: 设计并合成NS5ATP3基因序列特异性引物, 上下游引物序列分别为: 5'-GAA TTC ATG GTG CCG CCA CGA CGC CAT C-3', 5'-GGA TCC CTA ACT AGC TCC ATC CTC CTG-3'下划线部分为引物两端的酶切位点, EcoRI和BamHI. 以转染了HCV NS5A表达载体的HepG2细胞cDNA作为模板, 应用聚合酶链反应(PCR)技术扩增NS5ATP3的全长编码基因. 先克隆到TA载体中进行序列测定, 然后再亚克隆到真核表达载体pcDNA3.1(-)中, 构建真核表达载体pcDNA3.1(-)-NS5ATP3. 在35 mm培养皿中常规培养HepG2细胞, 细胞生长至对数期时, 分别以脂质体转染试剂Lipofectamine PLUS将2 μg pcDNA3.1(-)- NS5ATP3和空载体pcDNA3.1(-) 转染HepG2细胞, 48 h后收获细胞, 每5×106个细胞加入1 mL Trizol试剂. 立即于液氮中保存.
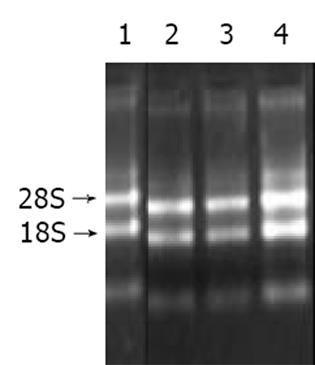
1.2.2 总RNA提取及mRNA纯化: 使用Trizol试剂一步法提取转染pcDNA3.1(-)-NS5ATP3和空载体pcDNA3.1(-)的细胞HepG2总RNA(分别标记为实验组和对照组), 样品经分光光度计检测吸光度A值, 并行热稳定实验, 于-20 ℃和70 ℃保温1 h后, 经琼脂糖凝胶电泳检测28 S、18 S条带变化. 纯化mRNA并行电泳检测.
1.2.3 探针标记及芯片制备: 常规方法逆转录标记cDNA探针并纯化. Cy3-dUTP标记对照组细胞mRNA (5 μg), Cy5-dUTP标记实验组细胞mRNA(5 μg). 乙醇沉淀后溶解在20 μL 5×SSC+0.2% SDS杂交液中. 芯片包含的1 152个cDNA以通用引物进行PCR扩增, PCR产物长度为1 000-3 000 bp. 靶基因以0.5 μg/μL溶解于3×SSC溶液中, 用Cartesian公司的Cartesian 7 500 点样仪及TeleChem公司的硅烷化玻片进行点样. 玻片经水合(2 h)、室温干燥(0.5 h), UV交联, 再分别用0.2% SDS、水及0.2% 的硼氢化钠溶液处理10 min, 晾干备用.
1.2.4 杂交及洗涤: 将基因芯片和杂交探针在95 ℃水浴变性5 min, 将混合探针加在基因芯片上, 置于60 ℃杂交15-17 h. 依次以2×SSC+ 0.2% SDS、0.1%×SSC +0.2% SDS、0.1%×SSC洗涤10 min, 室温晾干.
1.2.5 检测与分析: 用General Scanning公司的ScanArray 3 000扫描芯片. 用预先选定的内参照基因(24条管家基因, 每个基因点2个点, 共48个点)对Cy3和Cy5的原始提取信号进行均衡和修正. 用ImaGene3.0软件分析Cy3、Cy5两种荧光信号的强度, 计算Cy5/Cy3比值. 阳性结果判断: Cy5/Cy3>1.9, 红色荧光, 显示表达增强; Cy5/Cy3<0.6, 为绿色荧光, 显示表达减弱.
真核表达载体pcDNA3.1(-)-NS5ATP3经过限制性内切酶作图分析和核苷酸序列的测定, 证实含有完整的开放读码框架, 序列准确无误[6].
实验组和对照组总RNA的吸光度比值A260/A280分别为1.998和2.014, 热稳定实验70 ℃保温1 h与-20 : 1 h电泳条带比较, 显示28 S条带无明显降解, 电泳结果证实已抽提高纯度的总RNA(图1). mRNA主要集中于0.9-4.0 kb的连续条带.
在芯片上共有1 152个cDNA. 为了监控芯片杂交技术体系的整个过程, 在芯片上设置了阴性对照(8条水稻基因, 共8个点), 这些点的杂交信号均很低, 证实了数据的可靠性. 由于实验组探针标记Cy5荧光素(呈红色), 对照组探针标记Cy3荧光素(呈绿色), 红绿颜色的差异就显示该基因在实验组和对照组中基因表达水平上的差异, 黄色代表表达水平无差异. 按阳性标准, 从1 152个基因中筛选出差异表达基因共24条, 其中6条基因表达增强, 18条基因表达降低.
NS5ATP3上调基因类型: 在基因芯片的扫描分析中, 如果荧光染料的Cy5/Cy3比值在1.900以上, 就判断为NS5ATP3的上调基因. 在本研究中发现有6种基因的表达水平上调(表1).
| 序号 | GenBank登录号 | 编码蛋白 | Cy5/Cy3 |
| 1 | NM_013230 | 小细胞肺癌相关抗原CD24 | 1.955 |
| 2 | D50683 | 转化生长因子βII受体α | 1.983 |
| 3 | L04569 | L型电压依赖的钙离子通道α1亚单位hHT | 1.991 |
| 4 | NM_004661 | 细胞周期分裂蛋白CDC23 | 2.068 |
| 5 | NM_002736 | cAMP依赖的蛋白激酶调节亚单位IIβPRKAR2B | 2.073 |
| 6 | AF012086 | Ran 结合蛋白RanBP2α | 2.183 |
NS5ATP3下调基因类型: 在基因芯片的扫描分析中, 如果荧光染料的Cy5/Cy3比值在0.500以下, 就判断为NS5ATP3的下调基因. 在本研究中发现有18种基因的表达水平下调(表2).
| 序号 | GenBank登录号 | 编码蛋白 | Cy5/Cy3 |
| 1 | AL117234 | 定位于X染色体上的新基因 | 0.147 |
| 2 | NM_003034 | 唾液转移酶8A | 0.240 |
| 3 | NM_014337 | 肽基辅氨酰异构PPIL2 | 0.280 |
| 4 | NM_001764 | CD1B抗原β多肽 | 0.307 |
| 5 | NM_002982 | 小的可诱导细胞因子A2 | 0.326 |
| 6 | NM_022110 | FK506结合蛋白样蛋白 | 0.355 |
| 7 | NM_007177 | TU3A 蛋白 | 0.355 |
| 8 | NM_000844 | 谷氨酸酯受体 | 0.358 |
| 9 | NM_014451 | 甲状旁腺激素应答的骨肉瘤B1蛋白 | 0.361 |
| 10 | NM_000851 | 谷胱甘肽S转移酶 | 0.371 |
| 11 | NM_001775 | CD38 抗原 | 0.374 |
| 12 | NM_001648 | 前列腺特异性抗原 | 0.376 |
| 13 | NM_003254 | 金属蛋白酶1组织抑制因子 | 0.395 |
| 14 | BC004309 | RAS 癌基因家族成员RAB4 | 0.414 |
| 15 | NM_000732 | CD3D 抗原δ多肽 | 0.414 |
| 16 | NM_014020 | LR8蛋白 | 0.434 |
| 17 | NM_002183 | 白介素3受体αIL3RA | 0.447 |
| 18 | NM_000609 | 基质细胞来源因子1 | 0.493 |
HCV基因组含有单一的开放读码框架, 编码3 010-3 033个氨基酸残基的多蛋白肽前体, 两侧是5'-非翻译区及3'-非翻译区, 多肽前体至少被加工为10种结构蛋白和非结构蛋白[7-9]. 临床和实验研究显示, HCV 核心蛋白、非结构蛋白3(NS3)具有多种调控细胞、病毒基因表达、细胞生长以及免疫调节等功能, 分别在HCV感染及致肝细胞癌(HCC)发生发展过程中扮演重要的角色[10-11]. 非结构蛋白5A(NS5A)蛋白也不例外[4-5]. HCV NS5A位于HCV多蛋白的羧基末端, 是丝氨酸磷酸化蛋白质, 依磷酸化程度的不同而产生两种不同分子量大小的多肽p56和p58. 由于NS5A表现出对抗干扰素α(IFNα)的治疗效应而引起人们广泛的关注, NS5A能够与肝细胞中的IFNα刺激蛋白-双链RNA依赖的激酶(PKR)相互作用, 抑制PKR的功能, 从而下调IFNα刺激的抗病毒效应[12-13]. 近年研究发现, NS5A是转录反式激活因子, 其羧基末端富含酸性氨基酸及脯氨酸, 这是真核细胞转录因子特有的结构特征. 但是其参与细胞转录调节的机制仍不十分清楚. 一般来说, 转录的反式激活因子是在细胞核中起作用的, 而NS5A定位于细胞内质网(ER), 因此推测NS5A必定参与了细胞信号传导途径. NS5A能够反式激活核转录因子NF-κB及STAT3, 在细胞炎症反应、肿瘤发生及转移过程中起重要作用[14]. Ghosh et al[15]研究发现, NS5A蛋白能够抑制细胞周期调节基因p21WAF1, 激活人肝癌细胞中增生的细胞核抗原基因(PCNA), 从而调节细胞凋亡, 促进细胞增生. NS5A cDNA能够使转染的小鼠成纤维细胞NIH 3T3具有转化特性, 且转化细胞移植入裸鼠体内可形成纤维肉瘤灶, 这一证据直接证明了HCV NS5A蛋白的恶性转化潜能. 可见, HCV非结构蛋白NS5A的反式激活功能在HCV致病中发挥了重要作用, 那么, 肝细胞内表达的NS5A蛋白, 究竟激活了哪些特定基因的表达?这些表达产物对细胞的损害、增生分化的作用如何?
基于上述研究目的, 我们采用抑制性消减杂交方法, 成功筛选并克隆了HCV非结构蛋白NS5A基因转染细胞反式激活基因cDNA文库, 对挑选的克隆进行分析发现其中一个新基因与GenBank中注册的已知功能基因序列没有同源. 电子拼接推定该基因的开放读码框架, 获得相应的全长编码基因, 将该未知功能基因命名为NS5ATP3. 根据这一序列设计引物, 以人肝癌细胞cDNA为模板, PCR反应扩增NS5ATP3基因序列. 反应产物经测序完全符合计算机分析结果, 这表明我们已顺利得到了NS5ATP3编码序列. NS5ATP3的ORF长度为1 572 bp, 编码524个氨基酸残基的蛋白, 然而NS5ATP3基因的生物学功能及其在丙型肝炎发病机制中的作用目前还不清楚, 为进一步研究NS5ATP3的生物学功能, 我们利用基因芯片技术对其上调、下调基因进行分析[16-18]. 结果表明, 6种基因的表达水平上调, 18种基因的表达水平下调.这些基因包括细胞生长、细胞凋亡、信号转导、免疫调节、肿瘤发生等基因.
分析表达增强的基因, B淋巴细胞表面的分化抗原CD24是B细胞表面的信号转导分子, 能够调节多种信号活化的应答, 能诱导B淋巴细胞凋亡, 下游信号分子是丝裂素活化的蛋白激酶有丝分裂原激活蛋白基酶(MAPK)的激活以及细胞外信号调节的激酶1(ERK1)的活化. 研究还发现, CD24抗原是肝细胞癌细胞中高度表达的基因, 与p53基因突变及肿瘤分化高度相关, CD24抗原是潜在的肝细胞肿瘤早期标志基因[19-20]. 细胞分裂周期蛋白CDC23与酿酒酵母的CDC23高度同源, 是促进细胞分裂后期复合物(anaphase-promoting complex, APC)的成员, 对于细胞周期由G2期向M期过渡是必不可少的, APC能够催化细胞周期蛋白B与泛素(cyclin B-ubiquitin)形成共轭复合物, 在泛素介导的细胞周期蛋白B的蛋白水解过程中起重要作用, 推测NS5ATP3通过上调CDC23的表达对细胞周期有调控作用[21]. L型电压依赖的钙离子通道是细胞电信号转导途径中重要的调节因子[22], cAMP依赖的蛋白激酶调节亚单位IIβPRKAR2B是参与细胞信号转导途径主要因子[23]. Ran是真核细胞核运输过程中必需的小GTP酶, 在细胞mRNA加工, 细胞周期调节等方面发挥作用, Ran结合蛋白2即RanBP2是核孔蛋白, 在细胞核浆物质转运过程中起重要作用[24], NS5ATP3通过上调上述基因的表达, 提示该蛋白可能对细胞信号传递途径及核内外物质运输等过程有正调控作用, 而促进某些与细胞转录调节密切相关的基因, 如癌基因的转录, 在HCV感染过程中发挥一定的作用.
分析表达降低的基因, 胸腺细胞抗原CD1B是参与胸腺细胞分化以及抗原递呈的非专职递呈抗原, 能够向T细胞递呈各种不同的短链糖脂类抗原, 而树突状细胞(DC)递呈的是长链糖脂类抗原[25-26]. TU3A是编码144个氨基酸残基的肿瘤抑制因子, 在肾细胞肿瘤中该基因的表达下调, 并发现广泛存在的缺失突变, 该基因是否在其他肿瘤如肝细胞肿瘤中也具有同样的生物行为有待于进一步研究[27-28]. 甲状旁腺激素(PTH)应答的骨肉瘤B1蛋白是PTH刺激下调的造骨细胞功能形成过程中表达的一个新蛋白, 在骨形成时参与甲状旁腺激素的调节[29]. 谷胱甘肽S转移酶M5属于mu类, 具有对亲电子化合物的解毒功能, 包括致癌物质、治疗药物、环境毒素和氧化应激产物等有害物质通过与谷胱甘肽结合而发挥其解毒功效, 该基因的变异可以改变个体对致癌物质和毒物的易感性, 同时也影响某些药物的毒性和药效[30]. CD38蛋白是广泛表达于各种细胞和组织尤其是白细胞的一种多功能的胞外酶, 参与细胞黏附、信号转导和钙信号传递等生物过程[31]. CD3D抗原δ多肽是与T淋巴细胞表面的抗原受体结合的小完整膜蛋白, 能将活化信号转移到细胞内, 参与免疫信息的传递[32-33]. LR8蛋白是应用差异显示方法从肺纤维细胞克隆的未知功能蛋白[34]. 已知趋化因子是一类与靶细胞表面特异性受体结合而活化并介导白细胞迁移的蛋白, 依据半胱氨酸残基的位置不同而分为4类: C、CC、CXC和CX3C. 而CXCR4受体的配体-基质细胞来源因子SDF1是直接激活并介导白细胞迁移的趋化因子, 又称为CXC趋化因子, 是免疫缺陷病毒HIV-1入侵T细胞的辅助受体分子, 是通过G蛋白耦连的7个跨膜受体而行使淋巴细胞趋化效应, 同时这7个跨膜受体也是免疫缺陷病毒HIV-1入侵T细胞的辅因子. SDF1是HIV-1入侵细胞的辅因子LESTR的生物配体, 能够激活细胞内游离钙, 抑制HIV-1病毒感染T细胞[35-36]. NS5ATP3通过下调上述基因的表达水平, 推测其在调节细胞免疫应答、信号转导及物质代谢等方面具有一定作用.
总之, 利用基因表达谱芯片分析了NS5ATP3蛋白对肝细胞基因表达谱的影响, NS5ATP3蛋白能够上调或下调HepG2细胞中许多不同基因表达的变化, 这些基因变化是复杂的, 有的甚至是矛盾的, 这些基因与细胞信号转导、细胞增生与分化、免疫应答、物质运输、细胞凋亡等生物过程密切相关. 本实验结果对于初步了解新基因NS5ATP3在肝细胞中的生物学效应提供某些线索, 当然, 还需要进一步的实验加以深入研究.
编辑: N/A
| 1. | Neddermann P, Clementi A, De Francesco R. Hyperphosphorylation of the hepatitis C virus NS5A protein requires an active NS3 protease, NS4A, NS4B, and NS5A encoded on the same polyprotein. J Virol. 1999;73:9984-9991. [PubMed] |
| 2. | Rosenberg S. Recent advances in the molecular biology of hepatitis C virus. J Mol Biol. 2001;313:451-464. [PubMed] [DOI] |
| 3. | Kato N, Yoshida H, Ono-Nita SK, Kato J, Goto T, Otsuka M, Lan K, Matsushima K, Shiratori Y, Omata M. Activation of intracellular signaling by hepatitis B and C viruses: C-viral core is the most potent signal inducer. Hepatology. 2000;32:405-412. [PubMed] [DOI] |
| 4. | 刘 妍, 段 惠娟, 成 军, 王 建军, 陆 荫英, 牟 劲松, 王 琳, 张 玲霞. 丙型肝炎病毒非结构蛋白NS5A反式激活SV40病毒早期启 动子的研究. 军医进修学院学报. 2003;24:81-83. |
| 6. | 刘 妍, 杨 倩, 成 军, 王 建军, 纪 冬, 王 春花, 党 小燕, 张 玲霞. 丙型肝炎病毒非结构蛋白NS5A反式激活基因NS5ATP3的克隆化研究. 解放军医学杂志. 2004;(待发表). |
| 7. | Kato N. Molecular virology of hepatitis C virus. Acta Med Okayama. 2001;55:133-159. [PubMed] |
| 8. | De Francesco R. Molecular virology of the hepatitis C virus. J Hepatol. 1999;31 Suppl 1:47-53. [PubMed] [DOI] |
| 9. | Kim JE, Song WK, Chung KM, Back SH, Jang SK. Subcellular localization of hepatitis C viral proteins in mammalian cells. Arch Virol. 1999;144:329-343. [PubMed] [DOI] |
| 10. | 刘 妍, 成 军, 王 刚, 李 克, 段 惠娟, 王 琳, 董 菁, 洪 源, 张 跃新, 李 莉. 应用抑制性消减杂交技术克隆丙型肝炎病毒核心蛋白反式激活基因. 解放军医学杂志. 2001;26:880-883. |
| 11. | 刘 妍, 成 军, 牟 劲松, 陆 荫英, 王 建军, 李 克, 王 琳, 张 玲霞. 丙型肝炎病毒非结构蛋白NS3反式激活SV40病毒早期启动子的研究. 解放军医学杂志. 2003;28:44-46. |
| 12. | Koch JO, Bartenschlager R. Modulation of hepatitis C virus NS5A hyperphosphorylation by nonstructural proteins NS3, NS4A, and NS4B. J Virol. 1999;73:7138-7146. [PubMed] |
| 13. | Sato C. [Effects of hepatitis C virus proteins on the interferon-stimulated signal transduction]. Nihon Rinsho. 2001;59:1271-1276. [PubMed] |
| 14. | Gong G, Waris G, Tanveer R, Siddiqui A. Human hepatitis C virus NS5A protein alters intracellular calcium levels, induces oxidative stress, and activates STAT-3 and NF-kappa B. Proc Natl Acad Sci USA. 2001;98:9599-9604. [PubMed] [DOI] |
| 15. | Ghosh AK, Steele R, Meyer K, Ray R, Ray RB. Hepatitis C virus NS5A protein modulates cell cycle regulatory genes and promotes cell growth. J Gen Virol. 1999;80:1179-1183. [PubMed] [DOI] |
| 16. | Lakhani SR, Ashworth A. Microarray and histopathological analysis of tumours: the future and the past? Nat Rev Cancer. 2001;1:151-157. [PubMed] [DOI] |
| 17. | Kamiński S. DNA microarrays - a methodological breakthrough in genetics. J Appl Genet. 2002;43:123-130. [PubMed] |
| 19. | Kay R, Rosten PM, Humphries RK. CD24, a signal transducer modulating B cell activation responses, is a very short peptide with a glycosyl phosphatidylinositol membrane anchor. J Immunol. 1991;147:1412-1416. [PubMed] |
| 20. | Huang LR, Hsu HC. Cloning and expression of CD24 gene in human hepatocellular carcinoma: a potential early tumor marker gene correlates with p53 mutation and tumor differentiation. Cancer Res. 1995;55:4717-4721. [PubMed] |
| 21. | Zhao N, Lai F, Fernald AA, Eisenbart JD, Espinosa R, Wang PW, Le Beau MM. Human CDC23: cDNA cloning, mapping to 5q31, genomic structure, and evaluation as a candidate tumor suppressor gene in myeloid leukemias. Genomics. 1998;53:184-190. [PubMed] [DOI] |
| 22. | Schultz D, Mikala G, Yatani A, Engle DB, Iles DE, Segers B, Sinke RJ, Weghuis DO, Klockner U, Wakamori M. Ctional expression of the alpha 1 subunit of the L-type voltage-dependent calcium channel from normal human heart. Proc Natl Acad Sci USA. 1993;90:6228-6232. [DOI] |
| 23. | Solberg R, Sistonen P, Träskelin AL, Bérubé D, Simard J, Krajci P, Jahnsen T, de la Chapelle A. Mapping of the regulatory subunits RI beta and RII beta of cAMP-dependent protein kinase genes on human chromosome 7. Genomics. 1992;14:63-69. [PubMed] [DOI] |
| 24. | Nothwang HG, Rensing C, Kübler M, Denich D, Brandl B, Stubanus M, Haaf T, Kurnit D, Hildebrandt F. Identification of a novel Ran binding protein 2 related gene (RANBP2L1) and detection of a gene cluster on human chromosome 2q11-q12. Genomics. 1998;47:383-392. [PubMed] [DOI] |
| 25. | Moody DB, Briken V, Cheng TY, Roura-Mir C, Guy MR, Geho DH, Tykocinski ML, Besra GS, Porcelli SA. Lipid length controls antigen entry into endosomal and nonendosomal pathways for CD1b presentation. Nat Immunol. 2002;3:435-442. [PubMed] [DOI] |
| 26. | Sugita M, Cao X, Watts GF, Rogers RA, Bonifacino JS, Brenner MB. Failure of trafficking and antigen presentation by CD1 in AP-3-deficient cells. Immunity. 2002;16:697-706. [PubMed] [DOI] |
| 27. | Wang L, Darling J, Zhang JS, Liu W, Qian J, Bostwick D, Hartmann L, Jenkins R, Bardenhauer W, Schutte J. Loss of expression of the DRR 1 gene at chromosomal segment 3p21.1 in renal cell carcinoma. Genes Chromosomes Cancer. 2000;27:1-10. [PubMed] [DOI] |
| 28. | Yamato T, Orikasa K, Fukushige S, Orikasa S, Horii A. Isolation and characterization of the novel gene, TU3A, in a commonly deleted region on 3p14.3--> p14.2 in renal cell carcinoma. Cytogenet Cell Genet. 1999;87:291-295. [PubMed] [DOI] |
| 29. | Adams AE, Rosenblatt M, Suva LJ. Identification of a novel parathyroid hormone-responsive gene in human osteoblastic cells. Bone. 1999;24:305-313. [PubMed] [DOI] |
| 30. | Xu S, Wang Y, Roe B, Pearson WR. Characterization of the human class Mu glutathione S-transferase gene cluster and the GSTM1 deletion. J Biol Chem. 1998;273:3517-3527. [PubMed] [DOI] |
| 31. | Munshi CB, Graeff R, Lee HC. Evidence for a causal role of CD38 expression in granulocytic differentiation of human HL-60 cells. J Biol Chem. 2002;277:49453-49458. [PubMed] [DOI] |
| 32. | Gold DP, Clevers H, Alarcon B, Dunlap S, Novotny J, Williams AF, Terhorst C. Evolutionary relationship between the T3 chains of the T-cell receptor complex and the immunoglobulin supergene family. Proc Natl Acad Sci USA. 1987;84:7649-7653. [PubMed] [DOI] |
| 33. | Kastrup J, Pedersen LØ, Dietrich J, Lauritsen JP, Menné C, Geisler C. In vitro production and characterization of partly assembled human CD3 complexes. Scand J Immunol. 2002;56:436-442. [PubMed] [DOI] |
| 34. | Lurton J, Rose TM, Raghu G, Narayanan AS. Isolation of a gene product expressed by a subpopulation of human lung fibroblasts by differential display. Am J Respir Cell Mol Biol. 1999;20:327-331. [PubMed] [DOI] |
| 35. | Bleul CC, Farzan M, Choe H, Parolin C, Clark-Lewis I, Sodroski J, Springer TA. The lymphocyte chemoattractant SDF-1 is a ligand for LESTR/fusin and blocks HIV-1 entry. Nature. 1996;382:829-833. [PubMed] [DOI] |
| 36. | Barbero S, Bajetto A, Bonavia R, Porcile C, Piccioli P, Pirani P, Ravetti JL, Zona G, Spaziante R, Florio T. Expression of the chemokine receptor CXCR4 and its ligand stromal cell-derived factor 1 in human brain tumors and their involvement in glial proliferation in vitro. Ann N Y Acad Sci. 2002;973:60-69. [PubMed] [DOI] |