修回日期: 2002-12-10
接受日期: 2002-12-16
在线出版日期: 2003-09-15
哺乳动物基因组中, DNA甲基化是指CpG二核苷酸中的胞嘧啶第5位碳原子被甲基化. DNA甲基化是一种基因外修饰, 不改变DNA的一级结构; 他在细胞正常发育、基因表达模式以及基因组稳定性中起着至关重要的作用. 全基因组低甲基化, 维持甲基化模式酶的调节失控和正常非甲基化CpG岛的高甲基化是人类肿瘤中普遍存在的现象. DNA高甲基化是导致抑癌基因失活的又一个机制. 本文综述了抑癌基因的高甲基化、DNA修复基因的高甲基化、甲基化与转录的关系以及导致转录失活可能存在的作用机制、寻找甲基化相关基因的依据原则、甲基化的检测方法、肿瘤甲基化图谱的特征、甲基化与突变的相互作用、导致甲基化产生的原因、及其广泛的应用前景.
引文著录: 刘仲敏, 刘芝华, 吴旻. DNA高甲基化与抑癌基因. 世界华人消化杂志 2003; 11(9): 1420-1424
Revised: December 10, 2002
Accepted: December 16, 2002
Published online: September 15, 2003
N/A
- Citation: N/A. N/A. Shijie Huaren Xiaohua Zazhi 2003; 11(9): 1420-1424
- URL: https://www.wjgnet.com/1009-3079/full/v11/i9/1420.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v11.i9.1420
在哺乳动物基因组中, 胞嘧啶的第5位碳原子可以被的胞嘧啶[1]. DNA甲基化是一种基因外(epigenetic)修饰, 他不改变DNA的一级结构并且在细胞正常发育、基因表达模式以及基因组的稳定性中起着至关重要的作用.DNA甲基化模式是动态的. 例如, 在早期发育阶段, 受精后发育的特征是在全基因组范围内发生去甲基化, 接下来又发生区域性的甲基化, 这种基因组范围的DNA甲基化改变使甲基化模式得以建立. 甲基化模式的混乱与异常发育有关, 包括癌症.
DNA甲基化的改变, 与遗传因素改变一样都是人类肿瘤的特点之一. 许多基因, 尤其是管家基因的启动子区富含CpG二核苷酸, 诸如此类启动子区定义为CpG岛. 除定位于失活X染色体上的基因和印迹基因外, 正常细胞的CpG岛由于被保护而处于非甲基化状态.全基因组低甲基化, 维持甲基化模式酶的调节失控和正常非甲基化CpG岛的高甲基化是人类肿瘤中普遍存在的现象. 以往的研究证明启动子区的高甲基化导致抑癌基因失活是人类肿瘤所具有的共同特征之一, 而且这种高甲基化是导致抑癌基因失活的又一个机制.1986年, 有文章报道在肺癌和淋巴瘤中发现了位于11号染色体短臂的calcitonin基因存在高甲基化现象. 这一发现使更多的学者致力于CpG岛异常甲基化的研究中.
人类肿瘤中, 抑癌基因CpG岛高甲基化1989年首次在视网膜母细胞瘤(Rb)基因中发现, 继首次在H-ras基因中发现突变之后. 后来证明Rb基因启动子的高甲基化可以显著下调体外报告基因的表达. 除了视网膜母细胞瘤中未发现该基因的甲基化.
直到1994年发现VHL基因的失活也与甲基化相关, 才确定肿瘤中启动子CpG岛高甲基化是基因失活的一个作用机制. 但是, 肿瘤中基因外失活现阶段的研究是始于Stephen Baylin et al发现抑癌基因p16的失活通常都采用CpG岛高甲基化的作用机制. p16基因, 即CDKN2A基因, 一种细胞周期素依赖性激酶抑制蛋白, 他可以抑制通过磷酸化使Rb蛋白失活细胞周期素D依赖性激酶的活性. p16基因的失活将导致细胞丧失激活Rb基因的能力进而使细胞生长失去控制. p16基因位于染色体9p的区域内, 许多肿瘤中该区段经常发生缺失、纯合性缺失和一些点突变, 这一现象提示该基因在肿瘤形成中发挥重要作用. 但是, 大量的研究表明在很多类型原位癌中很少能检测到该基因的纯合性缺失, 且在肿瘤中也很少能检测到p16基因的点突变. 事实上, 与p16基因失活相关的高甲基化是肿瘤中经常发生的事件之一. 最初有关p16基因甲基化的报道主要存在于肺癌、头颈癌、神经胶质瘤、结直肠癌和乳腺癌中.后来这些研究几乎涉及所有上皮来源的癌, 但p16基因的甲基化不存在于小细胞肺癌和部分血癌中.
与p16基因相邻的p15基因, 在Rb基因的磷酸化调节中具有与其相似的结构和功能. p15基因与p16基因经常同时缺失. 但是, 上述涉及的肿瘤中p15基因很少通过甲基化失活. 在许多血癌中可以检测到p15基因的高甲基化频繁发生.
乳腺癌散发性病例的乳腺癌易感基因BRCA1中没有检测到体细胞突变, 这一点提示可能是甲基化导致该基因的失活. 近来的研究工作已经证明在一小部分乳腺癌和乳腺癌细胞系的CpG岛中存在甲基化[2-4]. 但没有发现BRCA2基因存在甲基化[5].
p73基因, p53基因的同源基因, 定位于1号染色体. 大量的报道表明, 与p53基因不同, 在该基因中很少能检测到突变. 近来的两篇报道已经发现在急性淋巴细胞白血病和淋巴瘤中p73基因表达失活与甲基化有关[6,7]. p14ARF基因具有调节p53蛋白稳定性的作用, 在部分结直肠癌细胞系中发生甲基化[8].
在肿瘤中, 可以发现参与DNA修复的基因经常由于启动子区高甲基化而失活. 第一个在前列腺癌中被证明的此类基因是谷胱甘肽转移酶(GSTπ), 实验证明甲基化异常发生在大多数前列腺癌中, 并且他还是导致该基因失活的原因. 同时也在乳腺癌和肾癌中发现这一基因被甲基化, 但在其他的肿瘤中很少发现这一现象[9].
第二个具有肿瘤特异性甲基化的DNA修复基因是O6-甲基鸟嘌呤DNA甲基转移酶(MGMT). 近来的研究证明存在于该基因启动子区或第一外显子内的CpG岛甲基化与其转录失活有关[10], 而且这种变化可以使染色质进入关闭状态, 进而导致核小体定位异常[11]. 该变化不仅存在于细胞系中, 也发生在原位癌中. MGMT基因的甲基化异常多见于结直肠癌、部分淋巴瘤、肺癌以及脑肿瘤[12,13].
最后一个 DNA修复基因是MLH1.许多工作发现存在于散发性结直肠癌、子宫内膜癌和胃癌中的微卫星不稳定性经常是该基因被甲基化后造成的[14-16].
除上述基因外, 还发现其他一些基因, 如细胞因子信号抑制子(SOCS1)[17]、骨形成蛋白(BMP3B)[18]、RAS效应同源物(RASSF1)[19]、E-钙黏素、胆固醇受体、雌激素受体、雄激素受体、维甲酸受体b、CD44、FHIT等基因在不同肿瘤中存在着甲基化现象.
现在已知DNA甲基化参与了基因的转录调节. 一些具有CpG二核苷酸结合位点转录因子间的相互作用属于甲基化敏感型. 但是甲基化DNA对转录的影响更多情况下是通过与甲基化CpG结合蛋白和改变染色质结构的相关因子之间的相互作用实现的.
MeCP1 和MeCP2是首先鉴定出的两个甲基化CpG结合蛋白. MeCP2是单一多肽链, 包含一个与甲基化CpG结合的结构域(MBD)和一个转录抑制结构域(TRD)[20]. 另外四个甲基化CpG结合蛋白也具有MBD(MBD1, MBD2, MBD3, MBD4), 并且MBD2具有使MeCP1复合物更易结合甲基化DNA的作用; 这些甲基化CpG结合蛋白具有直接抑制转录的作用, 也能够与转录共抑制子形成复合物; 后来发现DNA甲基转移酶(DNMTs)也具有这两种功能. 具有甲基化CpG结合活性蛋白抑制转录的机制有待于研究. 有证据表明这一过程是由许多复杂机制完成的. 一个主要机制包括甲基化DNA在MBD介导下与Sin3或Mi-2/NuRD形成共抑制复合物, 他们反过来再与HDAC1、HDAC2、两个Rb相关的组蛋白结合蛋白、RbAP46 和RbAP48组成核心组蛋白去乙酰化复合物[21-24]. 此外还存在其他共抑制复合物, 但是还没有证明他们在转录失活机制中的扮演什么样的角色.HDACs可以使组蛋白H3和H4的N末端赖氨酸残基去乙酰化[25], 从而导致组蛋白所带正电荷增加.该组蛋白与带负电荷的DNA相互作用造成染色质结构压缩, 进一步可能通过限制转录因子的结合抑制转录作用. Mi-2/NuRD共抑制复合物也包含通过ATP依赖机制重新包装染色质的因子. 上述活动可以使核小体在DNA上的定位发生改变, 进而可能限制DNA和转录因子之间的相互作用.
这一描述可能是比较简单.首先, 染色质的重包装和组蛋白的去乙酰化是相互依赖的.其次, DNA甲基化可能需要HDAC的活动或染色质的重包装中的成分的参与. 第三, 事实上, HDACs产生序列特异性的转录抑制因子导致基因失活, 因而通过HDACs发挥作用的转录抑制因子可能不仅仅作用于甲基化相关的位点. 类似地, 乙酰化和甲基化介导的抑制作用也可能单独发挥功能. 综合起来, 这些现象进一步体现了抑制基因的表达是DNA甲基化、组蛋白乙酰化、染色质的重包装和序列特异的失活机制彼此之间复杂的相互作用所导致的.
尽管甲基化通常与转录失活有关, 但在印迹座位中至少有两个例子是例外的, 他们的甲基化可以使转录激活.
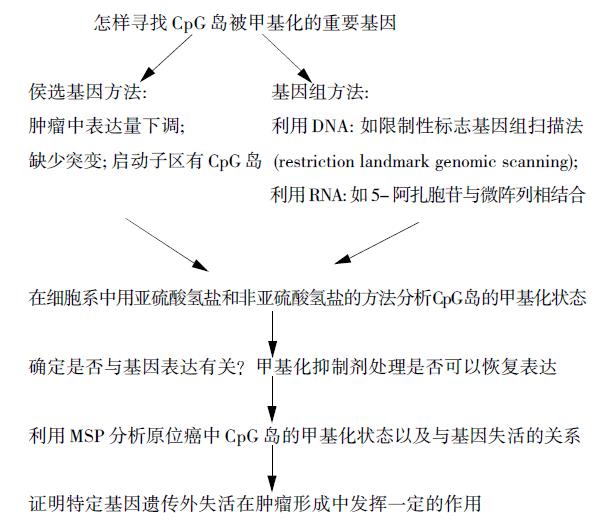
科研工作者的首要目标应该是对通常情况下认为CpG岛的甲基化与转录失活相关的甲基化进行研究. 图1列出在肿瘤中寻找甲基化相关基因的一个依据原则.
有关特定基因RNA转录起始位点的描述为发现DNA 甲基化可能在哪里发挥作用提供了线索. 如果GenBank中没有此方面的报道, 可以参考其他物种如老鼠启动子的相关信息. 同时也要考虑假基因的影响, 以便避免产生假阳性结果.最后是以往研究发现并没有确凿的证据证明在体外利用去甲基化抑制剂使细胞系中失活的基因恢复表达, 该基因的失活就一定与甲基化有关. 原因在于该药可能间接地诱导基因表达. 获得确凿的证据的唯一方法是分析CpG岛的甲基化状态.
原则上首先可以利用亚硫酸氢盐和非亚硫酸氢盐的方法在肿瘤细胞系详细地研究CpG岛的甲基化.亚硫酸氢盐测序的研究有利于设计出区别甲基化和非甲基化模式的引物. 细胞系也允许利用去甲基化抑制剂对其进行处理, 以便研究基因去甲基化和恢复表达的情况. 对于原位癌, 诸如甲基化特异性PCR(MSP)的方法可进行大量标本的筛查.
要对CpG岛的DNA 甲基化模式进行较深入的了解, 必须利用亚硫酸氢盐修饰过的DNA进行研究. 亚硫酸氢盐可以使非甲基化的胞嘧啶转变成尿嘧啶, 而甲基化的胞嘧啶不发生改变. 这种方法造成甲基化和非甲基化DNA序列的差异. 该方法中DNA的完全变性以及胞嘧啶完全转变为尿嘧啶均可避免产生假阳性结果.
以亚硫酸氢盐修饰为基础, 用于CpG岛甲基化分析的研究方法包括以下两种: (1)测序. 测定亚硫酸氢盐修饰过DNA的序列是检测胞嘧啶甲基化最直接的方法. 利用PCR反应扩增修饰过的DNA, 获得目的基因双链DNA. 可以对该产物直接进行序列分析; 也可以将其克隆测序获得单链DNA的甲基化图谱. (2)MSP. 在研究CpG岛甲基化中, MSP是应用最广泛的一种技术. 利用亚硫酸氢盐修饰造成甲基化和非甲基化DNA序列差异, 可以设计出各自特异性引物. 该技术的极高灵敏度允许他对小量样本的DNA进行甲基化研究, 包括来自石蜡包埋样品或显微切割组织的DNA. 由于MSP方法的多种优点, 他被认为在人类肿瘤甲基化分析中是一种快速、高效临床研究手段. 最后, 另外一种MSP是"Methylight", 他利用荧光标记的时PCR技术进行检测[26], 荧光检测大大提高了该方法的灵敏度. 别的方法包括结合亚硫酸氢盐的限制性分析; 甲基化敏感的单核苷酸引物延伸法; 甲基化敏感的单链构象分析.
肿瘤是涉及多重信号通路和遗传损伤的一种疾病, 他们在肿瘤的形成中是必不可少的. 基因外损伤也同样如此. CpG岛甲基化所涉及的基因分别参与了细胞周期(p16INK4a, Rb), DNA修复(MGMT, hMLH1), 细胞黏附(CDH1)等过程. 过去的3 a中, 肿瘤中启动子发生甲基化的基因数量增加的相当快. 这种不断发现可能导致研究者相信所有他们感兴趣的基因均可能发生异常甲基化, 并且在肿瘤形成中具有同等重要的作用. Esteller同时对以往有关甲基化的部分工作进行了分析, 发现如下规律: (1)根据肿瘤类型可以建立CpG岛甲基化图谱.Esteller et al [27]已经证明这种甲基化的特定模式同样存在于肿瘤起源的组织中.参考以往报道的文献, 可以发现有些类型的肿瘤比别的肿瘤更易发生甲基化: 例如最容易甲基化的肿瘤主要起源于胃肠管道, 而诸如卵巢癌一类的肿瘤则很少发生甲基化[27]. 另外一个结论是具有不同程度CpG岛甲基化的肿瘤呈梯度分布. 因而, 不大可能存在一种所有CpG岛均甲基化的肿瘤. (2)染色体上是否存在异常甲基化的热点?核内是否有控制甲基化的因子?许多最近发表的文章已经报道出通过甲基化导致转录失活的基因都位于3号染色体短臂附近的同一区域[28,29]. 该区段在许多肿瘤中存在很高频率的杂合性丢失, 尤其在非小细胞肺癌中.许多学者对该区段进行突变检测, 但一无所获. 答案似乎是一些基因的失活可能与甲基化有关. 但还无法确定这一区域是否属于异常甲基化热点. (3)大量CpG岛发生甲基化基因的基因组结构具有如下特征: 他们通常具有两个不同的启动子, 且都有CpG岛. 主要转录产物来自于强启动子, 而弱启动子则是少量转录产物起始处.如APC、ER等基因均属于上述类型.
过去一直认为许多肿瘤的形成主要是基因突变引起的.但20年后只发现癌基因K-ras和抑癌基因p53在许多不同类型的肿瘤中存在较高频率的突变. 现在已发现越来越多基因的失活可能与甲基化有关. 但是, 赋予他在肿瘤发生及发展过程中扮演着关键性的角色最重要的发现是甲基化导致特定基因失活的证实. 如DNA修复基因, 他们由于甲基化失活可能改变细胞的整个遗传环境. 甲基化相关MGMT的失活可以引起参与结直肠癌形成过程的癌基因K-ras中G到A的转换[30], 即外基因损伤可以引起在肿瘤发展中起关键性作用基因的遗传损伤[30,31]. 由于启动子甲基化而失活的其他一些基因可能与DNA损伤相关.
遗传外损伤与遗传损伤可能是相互作用的. 遗传改变同样也影响参与甲基化模式建立和维持过程中基因作用. 如DNMT3b基因生殖细胞中的突变与异常甲基化有关, 但与肿瘤无关[32].
CpG岛甲基化经常发生在缺乏基因突变的情况下, 说明二者在导致基因失活的过程中发挥同等重要的作用. 另一个证明基因启动子甲基化在肿瘤细胞中发挥重要作用的途径是研究其恢复表达的效应. 例如通过去甲基化或转基因的方法都可以恢复由于甲基化失活基因的表达.
导致肿瘤甲基化模式改变的机制尚不清楚, 其可能原因有以下几种: (1)保护CpG岛免受甲基化因子的丢失. 从以往的研究可知, Aprt 基因胚胎细胞的CpG岛通过一个尚未证实的保护顺式作用元件与CpG岛边缘的Sp1位点相结合, 而使其处于去甲基化状态[33]. (2)肿瘤中DNA methyltransferase (DNMT)的过度表达.大量证据表明, DNMT1在肿瘤中的表达是上调的[34]. 但也有报道显示CpG岛的高甲基化与DNMT1的表达水平无关. (3)DNMT复合物受到破坏. 另一个潜在的因素可能与DNMT水平升高有关, 即指这些酶的酶相关或抑制成分的调节失控. 该失控可能由于与DNMTs形成复合物的一种或几种因子(例如pRb, DMAP1-DNMT结合蛋白)的丢失所致. (4)复制时序性的破坏. 基因组的复制在细胞周期S期通过空间和短暂模式得以实现. 在此过程中的变化使复制区域和染色质构成的区域无法正常地相互作用.CpG岛可能通过复制时序紊乱发生异常甲基化. 有证据表明, 肿瘤细胞具有一个比较不同步的复制时序.
尽管基因启动子的高甲基化和基因表达失活之间的关系有待于进一步阐明, 但是人们日益重视DNA甲基化在肿瘤早期诊断和预后分析中, 以及在肿瘤的预防和治疗中可能具有的重要作用. 以DNA甲基化为基础的标志物可以应用于肿瘤细胞的检测、肿瘤类型的识别和肿瘤治疗三个主要临床领域中.
与基因突变不同, CpG岛启动子甲基化位点在同一个基因中是恒定的. 对于所有的患者来说, 在某一个已知基因中只需单一类型的引物, 利用甲基化特异性PCR便可检测肿瘤特异性甲基化的变化情况. 且与杂合性缺失不同, 该PCR产物呈阳性, 这一特点为上述检测提供方便.
与基因改变不同, 遗传外改变是可逆的. 恢复启动子甲基化所引起失活关键基因的表达有助于逆转肿瘤细胞的表型. 人们可以为DNA去甲基化设计出象5-Aza C (5-aza-2-deoxycytidine)一样更为特异的药品用于肿瘤治疗. 近期的研究发现, 用HDAC (histone deacetylase)抑制子使高甲基化基因发生部分去甲基化可以恢复其表达活性, 这一实验结果在临床上具有广泛的应用前景.
遗传外改变已经成为肿瘤中最重要的分子事件之一. 某些抑癌基因CpG岛甲基化的发现将DNA甲基化与破坏多种信号通路的遗传改变联系起来. 启动子的高甲基化现在被认为是基因失活的真正的作用机制之一. 但是, 还有许多重要问题有待解决, 如保护CpG岛免受甲基化因子的鉴定; 甲基化通过什么途径导致基因失活等. 最后, 甲基化在肿瘤患者的分子治疗中有广泛的用途, 而且可能成为未来治疗方法的作用靶点.
| 1. | Ramsahoye BH, Biniszkiewicz D, Lyko F, Clark V, Bird AP, Jaenisch R. Non-CpG methylation is prevalent in embryonic stem cells and may be mediated by DNA methyltransferase 3a. Proc Natl Acad Sci USA. 2000;97:5237-5242. [DOI] |
| 2. | Dobrovic A, Simpfendorfer D. Methylation of the BRCA1 gene in sporadic breast cancer. Cancer Res. 1997;57:3347-3350. |
| 3. | Mancini DN, Rodenhiser DI, Ainsworth PJ, O'Malley FP, Singh SM, Xing W, Archer TK. CpG methylation within the 5 regulatory region of the BRCA1 gene is tumor specific and includes a putative CREB binding site. Oncogene. 1998;16:1161-1169. [DOI] |
| 4. | Rice JC, Massey-Brown KS, Futscher BW. Aberrant methylation of the BRCA1 CpG island promoter is associated with decreased BRCA1 mRNA in sporadic breast cancer cells. Oncogene. 1998;17:1807-1812. |
| 5. | Collins N, Wooster R, Stratton MR. Absence of methylation of CpG dinucleotides within the promoter of the breast cancer susceptibility gene BRCA2 in normal tissues and in breast and ovarian cancers. Br J Cancer. 1997;76:1150-1156. [DOI] |
| 6. | Kawano S, Miller CW, Gombart AF, Bartram CR, Matsuo Y, Asou H, Sakashita A, Said J, Tatsumi E, Koeffler HP. Loss of p73 gene expression in leukemias/lymphomas due to hypermethylation. Blood. 1999;94:1113-1120. |
| 7. | Corn PG, Kuerbitz SJ, van Noesel MM, Esteller M, Compitello N, Baylin SB, Herman JG. Transcriptional silencing of the p73 gene in acute lymphoblastic leukemia and Burkitt's lymphoma is associated with 5 CpG island methylation. Cancer Res. 1999;59:3352-3356. |
| 8. | Robertson KD, Jones PA. The human ARF cell cycle regulatory gene promoter is a CpG island which can be silenced by DNA methylation and down-regulated by wild-type p53. Mol Cell Biol. 1998;18:6457-6473. [DOI] |
| 9. | Esteller M, Corn PG, Urena JM, Gabrielson E, Baylin SB, Herman JG. Inactivation of glutathione S-transferase P1 gene by promoter hypermethylation in human neoplasia. Cancer Res. 1998;58:4515-4518. |
| 10. | Qian XC, Brent TP. Methylation hot spots in the 5 flanking region denote silencing of the 06-methylguanine-DNA methyltransferase gene. Cancer Res. 1997;57:3672-3677. |
| 11. | Patel SA, Graunke DM, Pieper RO. Aberrant silencing of the CpG island-containing human 06-methylguanine DNA methyltransferase gene is associated with the loss of nucleosome-like positioning. Mol Cell Biol. 1997;17:5813-5822. |
| 12. | Herfarth KK, Brent TP, Danam RP, Remack JS, Kodner IJ, Wells SA Jr, Goodfellow PJ. A specific CpG methylation pattern of the MGMT promoter region associated with reduced MGMT expression in primary colorectal cancers. Mol Carcinog. 1999;24:90-98. [DOI] |
| 13. | Esteller M, Hamilton SR, Burger PC, Baylin SB, Herman JG. Inactivation of the DNA repair gene 06-methylguanine-DNA methyltransferase by promoter hypermethylation is a common event in primary human neoplasia. Cancer Res. 1999;59:793-797. |
| 14. | Herman JG, Umar A, Polyak K, Graff JR, Ahuja N, Issa JP, Markowitz S, Willson JK, Hamilton SR, Kinzler KW. Incidence and functional consequences of hMLH1 promoter hypermethylation in colorectal carcinoma. Proc Natl Acad Sci USA. 1998;95:6870-6875. [DOI] |
| 15. | Gurin CC, Federici MG, Kang L, Boyd J. Causes and consequences of microsatellite instability in endometrial carcinoma. Cancer Res. 1999;59:462-466. |
| 16. | Yamamoto H, Perez-Piteira J, Yoshida T, Terada M, Itoh F, Imai K, Perucho M. Gastric cancers of the microsatellite mutator phenotype display characteristic genetic and clinical features. Gastroenterology. 1999;116:1348-1357. [DOI] |
| 17. | Yoshikawa H, Matsubara K, Qian GS, Jackson P, Groopman JD, Manning JE, Harris CC, Herman JG. SOCS-1, a negative regulator of the JAK/STAT pathway, is silenced by methylation in human hepatocellular carcinoma and shows growth suppression activity. Nat Genet. 2001;28:29-35. |
| 18. | Dai Z, Lakshmanan RR, Zhu WG, Smiraglia DJ, Rush LJ, Fruhwald MC, Brena RM, Li B, Wright FA, Ross P. Global methylation profiling of lung cancer identifies novel methylated genes. Neoplasia. 2001;3:314-323. |
| 19. | Dammann R, Li C, Yoon JH, Chin PL, Bates S, Pfeifer GP. Epigenetic inactivation of a RAS association domain family protein from the lung tumour suppressor locus 3p21.3. Nat Genet. 2000;25:315-319. [DOI] |
| 20. | Nan X, Ng HH, Johnson CA, Laherty CD, Turner BM, Eisenman RN, Bird A. Transcriptional repression by the methyl-CpG-binding protein MeCP2 involves a histone deacetylase complex. Nature. 1998;393:386-389. |
| 21. | Laherty CD, Yang WM, Sun JM, Davie JR, Seto E, Eisenman RN. Histone deacetylases associated with the mSin3 corepressor mediate mad transcriptional repression. Cell. 1997;89:349-356. [DOI] |
| 22. | Nagy L, Kao HY, Chakravarti D, Lin RJ, Hassig CA, Ayer DE, Schreiber SL, Evans RM. Nuclear receptor repression mediated by a complex containing SMRT, mSin3A, and histone deacetylase. Cell. 1997;89:373-380. |
| 23. | Gibbons RJ, McDowell TL, Raman S, O扲ourke DM, Garrick D, Ayyub H, Higgs DR. Mutations in ATRX, encoding a SWI/SNF-like protein, cause diverse changes in the pattern of DNA methylation. Nat Genet. 2000;24:368-371. |
| 24. | Knoepfler PS, Eisenman RN. Sin meets NuRD and other tails of repression. Cell. 1999;99:447-450. [DOI] |
| 25. | Kuo MH, Allis CD. Roles of histone acetyltransferases and deacetylases in gene regulation. Bioessays. 1998;20:615-626. [DOI] |
| 26. | Eads CA, Danenberg KD, Kawakami K, Saltz LB, Blake C, Shibata D, Danenberg PV, Laird PW. MethyLight: a high-throughput assay to measure DNA methylation. Nucleic Acids Res. 2000;28:E32. [DOI] |
| 27. | Esteller M, Corn PG, Baylin SB, Herman JG. A gene hypermeth-ylation profile of human cancer. Cancer Res. 2001;61:3225-3229. |
| 28. | Zochbauer-Muller S, Fong KM, Maitra A, Lam S, Geradts J, Ashfaq R, Virmani AK, Milchgrub S, Gazdar AF, Minna JD. 5 CpG island methylation of the FHIT gene is correlated with loss of gene expression in lung and breast cancer. Cancer Res. 2001;61:3581-3585. |
| 29. | Tomizawa Y, Sekido Y, Kondo M, Gao B, Yokota J, Roche J, Drabkin H, Lerman MI, Gazdar AF, Minna JD. Inhibition of lung cancer cell growth and induction of apoptosis after reexpression of 3p21.3 candidate tumor suppressor gene SEMA3B. Proc Natl Acad Sci USA. 2001;98:13954-13959. |
| 30. | Esteller M, Gonzalez S, Risques RA, Marcuello E, Mangues R, Germa JR, Herman JG, Capella G, Peinado MA. K-ras and p16 aberrations confer poor prognosis in human colorectal cancer. J Clin Oncol. 2001;19:299-304. [DOI] |
| 31. | Esteller M, Risques RA, Toyota M, Capella G, Moreno V, Peinado MA, Baylin SB, Herman JG. Promoter hypermethylation of the DNA repair gene O(6)-methylguanine-DNA methyltransferase is associated with the presence of G:C to A:T transition mutations in p53 in human colorectal tumorigenesis. Cancer Res. 2001;61:4689-4692. |
| 32. | Xu GL, Bestor TH, Bourc'his D, Hsieh CL, Tommerup N, Bugge M, Hulten M, Qu X, Russo JJ, Viegas-Pequignot E. Chromosome instability and immunodeficiency syndrome caused by mutations in a DNA methyltransferase gene. Nature. 1999;402:187-191. [DOI] |
| 33. | Rountree MR, Bachman KE, Herman JG, Baylin SB. DNA methylation, chromatin inheritance, and cancer. Oncogene. 2001;20:3156-3165. [DOI] |
| 34. | Baylin SB, Herman JG, Graff JR, Vertino PM, Issa JP. Alterations in DNA methylation: a fundamental aspect of neoplasia. Adv Cancer Res. 1998;72:141-196. [DOI] |