修回日期: 2017-11-29
接受日期: 2017-12-03
在线出版日期: 2017-12-28
NSD2也称为MMSET或者WHSC1, 是NSD组蛋白甲基转移酶家族中的一员. 组蛋白的甲基化修饰作为表观遗传学中重要的调控机制, 在转录调控以及染色质重构等多种生物学过程中发挥重要作用. 由t(4;14)(p16;q23)易位所导致的NSD2过表达与多发性骨髓瘤患者预后紧密相关. 此外, 在其他多种恶性肿瘤中也检测到NSD2的高表达. NSD2催化组蛋白的赖氨酸位点发生甲基化并通过参与多种蛋白的相互作用或对靶基因的调控而促使肿瘤的发生发展. NSD2及相关信号分子有望成为多种相关肿瘤的治疗靶点, 深入探究NSD2的作用机制将促进相关靶向药物的发展, 为多种NSD2相关肿瘤提供新的治疗方案.
核心提要: NSD2与肿瘤的关系越来越受重视. 高表达的NSD2与多种肿瘤恶性程度及患者预后紧密相关. 随着相关机制的阐明, 多种药物靶点被开发, 且部分靶向药物已进入临床试验阶段. 本文就其结构、功能、与肿瘤的关系以及相关药物靶点进行总结概括, 为相关研究工作者提供参考.
引文著录: 雷宏维, 陶凯雄. NSD2与肿瘤关系的研究进展. 世界华人消化杂志 2017; 25(36): 3218-3223
Revised: November 29, 2017
Accepted: December 3, 2017
Published online: December 28, 2017
Nuclear receptor binding SET domain-protein 2 (NSD2), also known as MMSET or WHSC1, is a member of the NSD family. Methylation of histone as an important regulatory mechanism in epigenetics plays an important role in transcriptional regulation and chromatin remodeling. The overexpression of NSD2, caused by t(4; 14) (p16; q23) translocation, is closely related to the prognosis of patients with multiple myeloma. In addition, high expression of NSD2 can be detected in many other malignant tumors. NSD2 catalyzes the methylation of the lysine sites of histone, and promotes the initiation and progression of tumors by participating in the interaction among multiple proteins or the regulation of target genes. NSD2 and related signal molecules are expected to be the targets for treatment of various related tumors. Elucidating the mechanism of NSD2 may promote the development of targeted drugs and provide new solutions for the treatment of various NSD2-related tumors.
- Citation: Lei HW, Tao KX. Relationship between NSD2 and tumors. Shijie Huaren Xiaohua Zazhi 2017; 25(36): 3218-3223
- URL: https://www.wjgnet.com/1009-3079/full/v25/i36/3218.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v25.i36.3218
近年来, 国内外多项研究证实核受体结合SET结构域蛋白2(nuclear receptor binding SET domain-protein 2, NSD2)在多种肿瘤的发生发展中发挥重要作用. 最初, 人们发现NSD2是由于其单倍体剂量不足所导致的Wolf-Hirschhorn综合征, 患者表现为生长和智力发育迟缓、先天性心脏病等. 随后, 有研究者发现15%-20%的多发性骨髓瘤(multiple myeloma, MM)患者染色体中存在t(4;14)(p16;q23). 该易位导致NSD2与IgH形成融合基因, 从而使得NSD2受IgH增强子作用而过表达, 这提示NSD2有潜在的致癌作用. 此外, 通过对多类肿瘤表达谱的研究分析, 发现NSD2除了在淋巴造血系统来源的肿瘤中高表达外, 还在脑肿瘤、乳腺癌、胰腺癌、膀胱癌等实体瘤中高表达[1], 并且高表达的NSD2与肿瘤恶性程度及患者预后紧密相关[2]. NSD2可能在肿瘤诊断及靶向治疗发挥重要作用, 现就其结构、功能、与肿瘤的关系以及相关药物靶点作一简要综述.
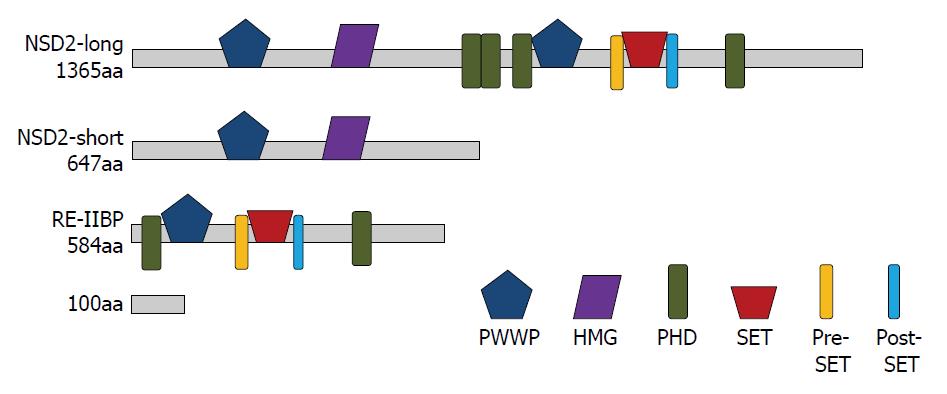
NSD2位于染色体4p16.3上, 大小90 kb, 包含25个外显子. 其剪切产物包括3个主要的异构体--长NSD2(MMSET-Ⅱ)、短NSD2(MMSET-Ⅰ)以及反应元件Ⅱ结合蛋白的内含子转录物(RE-IIBP). NSD2长片段MMSET-Ⅱ编码1365个氨基酸, 由两个PWWP结构域、高迁移率组(high mobility group, HMG)DNA结合结构域、4个PHD锌指结构和SET结构域组成[3-5]. PWWP、HMG、PHD结构域具有核定位、DNA结合、识别组蛋白修饰标记的作用[6], 而SET结构域作为组蛋白赖氨酸甲基转移酶的功能基团, 参与赖氨酸的甲基化. NSD2短片段MMSET-I仅编码647个氨基酸, 且只含有初始PWWP和HMG结构域. RE-IIBP编码584个氨基酸, 因含SET结构域而同样具有甲基化作用. 其中, 参与甲基化的SET结构域又由核心SET结构域、pre-SET结构域以及post-SET结构域共同组成. pre-SET结构域维持整个蛋白结构的稳定性, post-SET结构域提供1个芳香基团形成疏水通道参与构成部分酶活化位点, 而核心SET结构域通过侧面与这两者连接[7](图1).
NSD2的主要功能是参与组蛋白的甲基化修饰. 组蛋白的甲基化修饰主要发生在H3和H4组蛋白的赖氨基酸或精氨酸残基上, 且同一位点可发生单甲基化(me1)、二甲基化(me2)或三甲基化(me3). NSD2在参与基因转录激活过程中可通过其SET结构域催化 H3K4、H3K9、H3K27、H3K36、H4K20、H4K44等发生甲基化, 产生大量的组蛋白甲基化标志物[8]. 其中H3K36是其主要催化目标. 与H3K36me1相比, NSD2会优先催化H3K36me2[9]. 催化H3K36me2的过程首先由NSD2中PWWP结构域的第一个氨基末端特异性结合到H3K36me2并使NSD2稳定到染色质中, 同时PWWP催化SET结构域将基因激活信号传到临近的核小体. 随后NSD2 post-SET结构域经自我抑制环区连接到被催化的SET结构域, 通过进一步与核小体结合缓解其抑制功能从而促进NSD2的甲基转移作用[10].
最近研究[2]表明, NSD2不仅在MM中升高, 在多种实体瘤中也显著增高, 并提示患者预后更差. NSD2通过多种途径参与了肿瘤的发生发展.
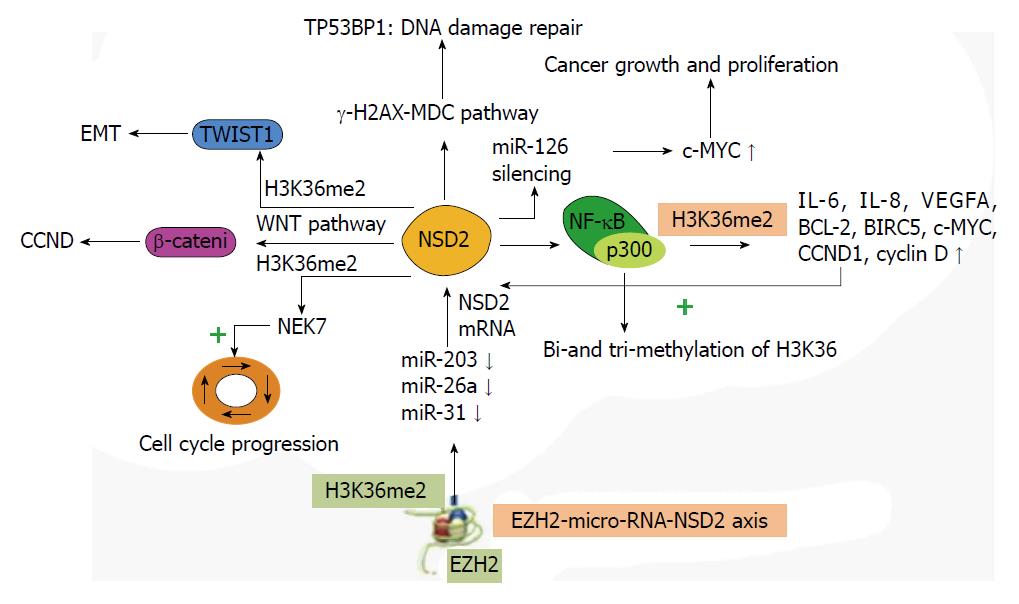
有研究者发现, 15%-20%的MM病例中含t(4;14)易位, 该易位直接导致FGFR3和NSD2与IgH形成融合基因, 受IgH增强子的作用而过度表达[11]. 其中FGFR3和NSD2均有潜在的致癌作用, 而NSD2被认为是主要的致癌驱动因子, 因为大约30%携带这种易位基因的病例其FGFR3正常表达却伴随NSD2的过表达[12,13]. NSD2可在MM细胞中可通过沉默miR-126而介导c-MYC过表达诱导细胞增殖[14]. c-MYC是MM中诱导细胞增殖上调的基因, 而miR-126可通过识别c-MYC的3'非编码区导致其翻译的抑制, 从而间接降低了c-MYC蛋白水平, 因此miR-126的表达可抑制细胞增殖. 但NSD2可与组蛋白脱乙酰酶HDAC1/2结合到miR126的启动子区域并诱导H3K9m3, 随后沉默miR-126导致c-MYC的上调并诱导细胞增殖[14]. EZH2促进NSD2mRNA上调. Asangani等[15]证实了EZH2与NSD2可通过microRNAs达成紧密联系, 形成EZH2-microRNA-NSD2组蛋白甲基转移酶轴. 其中组蛋白甲基转移酶EZH2是多梳蛋白抑制复合体2(polycomb repressive complex, 2PRC2)的一个催化亚基, 其主要通过SET结构域催化H3K27me3[16]. 相关研究表明, miR-203、miR-26a和miR-31可通过降低NSD2 mRNA的稳定性起到抑制肿瘤作用, 但EZH2介导的H3K27m3能抑制miR-203、miR-26a和miR-31的表达, 从而导致NSD2 mRNA的上调[15]. 此外, Popovic等[17]通过研究NSD2的过表达与EZH2的产物H3K27me3表达水平之间的关系证实: 因NSD2而过表达的H3K36me2, 其扩散可导致H3K27me3的全面减少, 形成更加开放的染色质状态. 在敲除了NSD2基因的t(4;14)MM细胞系中, 其细胞增殖、细胞周期进展以及DNA修复都减少了, 但细胞凋亡和黏附却增加了[18], 这一点也证实了NSD2促进肿瘤细胞的增殖. 除多发性骨髓瘤外, 在胃癌、结肠癌、肺癌以及皮肤癌中也检测到NSD2过度表达[19,20]. 且有证据表明, NSD2与神经母细胞瘤、乳腺肿瘤、膀胱肿瘤以及前列腺肿瘤的发生发展及恶性程度相关[21], 这也表明NSD2或许可作为相关肿瘤诊断的理想标志物.
Ezponda等[22]最近的一项研究显示NSD2可促进前列腺癌细胞的侵袭转移. TWIST1是一种基本的螺旋-环-螺旋转录因子, 他可以上调上皮-间质转化(epithelial-mesenchymal transition, EMT)特有的N-钙黏蛋白而参与细胞迁移. 而NSD2可使TWIST1基因体中的H3K36二甲基化并上调其转录, 从而促进前列腺癌细胞的EMT和迁移性[22]. NSD2这种直接结合TWIST1基因座并上调EMT相关因子表达的能力, 在晚期前列腺癌的侵袭转移生物学行为方面起关键作用[23]. 此外, 在t(4;14)MM细胞中也观察到NSD2介导的TWIST1转录上调. Lu等[24]证实NSD2可通过抑制E-钙黏蛋白表达促进骨肉瘤细胞增殖和转移. Asangani等[15]证实EZH2诱导正常乳腺和前列腺细胞的侵袭性, 也是通过增加NSD2表达介导的.
NSD2可通过直接转录上调NEK7促进细胞周期进程. NEK7是一种丝氨酸/苏氨酸蛋白激酶, 与中心体的微管成核活性、有丝分裂纺锤体形成以及细胞分裂有关. NEK7被证实是NSD2的直接下游靶基因, 敲减NSD2可诱导下游NEK7表达水平的下降及细胞周期阻滞[25]. NSD2在DNA复制中也发挥作用: 在细胞周期进展过程中S期需要NSD2的参与, 且沉默NSD2会导致S期的细胞会显著积累[26]. NSD2可以直接结合到细胞周期蛋白D1(cyclin-D1, CCND1)的启动子区域, 且敲减NSD2可使β连环蛋白(β-catenin)/Tcf-4复合物的靶基因CCND1表达降低, 从而影响细胞周期[1].
NSD2具有募集DNA损伤反应性p53结合蛋白1(TP53BP1)的能力. Pei等[27]报道说, TP53BP1可识别在DNA损伤区域由NSD2诱导生成的H4K20me2并被其招募, 从而参与DNA损伤修复. Hajdu等[28]实验结果显示, 在shRNA介导的HCT116结肠癌细胞中, 沉默NSD2会导致细胞对DNA损伤剂如羟基脲、喜树碱和丝裂霉素C以及电离辐射的敏感性增加. 有证据表明在NSD2缺失的情况下, DNA损伤反应系统保持完整, 但DNA损伤修复信号传导通路被延长. Shah等[18]最近报道, NSD2调节DNA修复基因的表达, 并且可能在MM化学耐药中起关键作用-表达高水平NSD2的癌细胞能更快地修复DNA损伤, 这使得他们在经历DNA损伤化疗后仍然能存活和增殖.
NSD2在多种肿瘤中高表达会使相关信号通路过度激活. NSD2可激活WNT信号通路: WNT信号通路与胚胎发育及肿瘤形成有关. 免疫共沉淀和质谱分析显示NSD2与β-catenin相互作用可激活WNT信号通路[1]. 且敲减NSD2可使β-catenin/Tcf-4复合物的靶基因CCND1表达降低, 提示β-catenin与NSD2之间可能的协同作用. NSD2可与核转录因子-κB(nuclear factor kappa B, NF-κB)相互作用: Yang等[29]发现NSD2诱导NF-κB下游基因启动子区域H3K36发生me2和me3, 从而促使该基因转录激活. 这提示NSD2在NF-κB通路中行使转录共激活因子的功能, 可通过与NF-κB相互作用激活白介素-6(interleukin-6, IL-6)、白介素-8(interleukin-8, IL-8)、血管内皮生长因(vascular endothelial growth factor, VEGFA)、 cyclin D、抗凋亡基因(Bcl-2)、凋亡抑制蛋白-5(BIRC5)、c-MYC及CCND1等靶基因[29](图2).
NSD2基因的易位、上调和激活能驱动肿瘤发生发展, Wang等[30]通过点突变使NUP98-NSD1的甲基转移酶失活而成功抑制了Hox-A基因的激活和骨髓祖细胞的永生化, 以及在相关肿瘤模型中敲减NSD2可以下调肿瘤细胞的临床表型等都证明了特异性赖氨酸组蛋白甲基转移酶(histone methyl transferases, HMT)抑制剂用于治疗NSD2相关肿瘤的应用前景. 鉴于活性赖氨酸HMT最有可能驱动致癌基因重新编码[31], 因此靶向SET结构域将是开发NSD2家族抑制剂有吸引力的策略. NSD2的PHD锌指2、3和4以及PWWP结构域2有利于t(4;14)MM中H3K36me2的上调[17], 而且在NSD2过表达的MM细胞中观察到第4个PHD手指的缺失消除了H3K27me3的减少, 从而间接影响了EZH2-microRNA-NSD2轴, 这提示靶向NSD2中其他结构域也有很好的应用前景. 考虑到EZH2可以上调NSD2的表达, 而过表达NSD2的MM细胞又能抑制EZH2, 这提示EZH2和NSD2双重靶向可能将在NSD2过表达的癌症中提供最佳的抗肿瘤作用. NSD2可与组蛋白去乙酰化酶(HDAC)形成复合物发挥生物功能[32], 研究针对两者复合物形成的HDAC抑制剂具有应用前景. 在t(4;14)MM细胞中miR-126的表达可抑制肿瘤细胞增殖, 通过调控体内miR-126的表达来治疗肿瘤也值得深入研究[33]. 尽管有证据表明, 遗传和表观遗传学改变在癌症的逐步发生和进展中相互配合, 但只有表观遗传学异常才能被逆转. 因此, 抑制NSD2相关表观遗传调节因子或许将在下一代癌症治疗中发挥核心作用.
随着大规模基因组学和转录组学分析的出现, NSD2的多种遗传学改变逐步被揭示. NSD2的许多功能性分析证实了其在相关肿瘤发生发展中的关键作用, 并且完善了他与其他信号分子的共同作用方式, 这使得其在肿瘤发病机制中的重要性也越来越被科学家关注. 目前国内外关于NSD2的基础研究正在开展, 其在肿瘤中的作用机制将会得到更明确的阐述. 该综述突显了NSD2甲基转移酶用于药物开发的潜力, 从而为药物的研发以及临床的治疗提供线索. 进一步研究NSD2的调控机制, 将为相关癌症的治疗提供新的有意义的靶点. 目前甲基转移酶抑制剂及HDAC抑制剂已经得到了很好的发展, 我们期待不久之后NSD2相关药物能在相关肿瘤治疗的方面得到广泛的应用.
手稿来源: 自由投稿
学科分类: 胃肠病学和肝病学
手稿来源地: 湖北省
同行评议报告分类
A级 (优秀): A
B级 (非常好): B, B
C级 (良好): 0
D级 (一般): D
E级 (差): 0
编辑:马亚娟 电编:杜冉冉
| 1. | Toyokawa G, Cho HS, Masuda K, Yamane Y, Yoshimatsu M, Hayami S, Takawa M, Iwai Y, Daigo Y, Tsuchiya E. Histone lysine methyltransferase Wolf-Hirschhorn syndrome candidate 1 is involved in human carcinogenesis through regulation of the Wnt pathway. Neoplasia. 2011;13:887-898. [PubMed] [DOI] |
| 2. | Xiao M, Yang S, Chen J, Ning X, Guo L, Huang K, Sui L. Overexpression of MMSET in endometrial cancer: a clinicopathologic study. J Surg Oncol. 2013;107:428-432. [PubMed] [DOI] |
| 3. | Baker LA, Allis CD, Wang GG. PHD fingers in human diseases: disorders arising from misinterpreting epigenetic marks. Mutat Res. 2008;647:3-12. [PubMed] [DOI] |
| 4. | Ge YZ, Pu MT, Gowher H, Wu HP, Ding JP, Jeltsch A, Xu GL. Chromatin targeting of de novo DNA methyltransferases by the PWWP domain. J Biol Chem. 2004;279:25447-25454. [PubMed] [DOI] |
| 5. | Shi X, Hong T, Walter KL, Ewalt M, Michishita E, Hung T, Carney D, Peña P, Lan F, Kaadige MR. ING2 PHD domain links histone H3 lysine 4 methylation to active gene repression. Nature. 2006;442:96-99. [PubMed] [DOI] |
| 6. | Huang Z, Wu H, Chuai S, Xu F, Yan F, Englund N, Wang Z, Zhang H, Fang M, Wang Y. NSD2 is recruited through its PHD domain to oncogenic gene loci to drive multiple myeloma. Cancer Res. 2013;73:6277-6288. [PubMed] [DOI] |
| 7. | Bennett RL, Swaroop A, Troche C, Licht JD. The Role of Nuclear Receptor-Binding SET Domain Family Histone Lysine Methyltransferases in Cancer. Cold Spring Harb Perspect Med. 2017;7. [PubMed] [DOI] |
| 8. | Morishita M, Mevius D, di Luccio E. In vitro histone lysine methylation by NSD1, NSD2/MMSET/WHSC1 and NSD3/WHSC1L. BMC Struct Biol. 2014;14:25. [PubMed] [DOI] |
| 9. | Poulin MB, Schneck JL, Matico RE, McDevitt PJ, Huddleston MJ, Hou W, Johnson NW, Thrall SH, Meek TD, Schramm VL. Transition state for the NSD2-catalyzed methylation of histone H3 lysine 36. Proc Natl Acad Sci USA. 2016;113:1197-1201. [PubMed] [DOI] |
| 10. | Poulin MB, Schneck JL, Matico RE, Hou W, McDevitt PJ, Holbert M, Schramm VL. Nucleosome Binding Alters the Substrate Bonding Environment of Histone H3 Lysine 36 Methyltransferase NSD2. J Am Chem Soc. 2016;138:6699-6702. [PubMed] [DOI] |
| 11. | Keats JJ, Reiman T, Maxwell CA, Taylor BJ, Larratt LM, Mant MJ, Belch AR, Pilarski LM. In multiple myeloma, t(4;14)(p16;q32) is an adverse prognostic factor irrespective of FGFR3 expression. Blood. 2003;101:1520-1529. [PubMed] [DOI] |
| 12. | Santra M, Zhan F, Tian E, Barlogie B, Shaughnessy J Jr. A subset of multiple myeloma harboring the t(4;14)(p16;q32) translocation lacks FGFR3 expression but maintains an IGH/MMSET fusion transcript. Blood. 2003;101:2374-2376. [PubMed] [DOI] |
| 13. | Chesi M, Nardini E, Lim RS, Smith KD, Kuehl WM, Bergsagel PL. The t(4;14) translocation in myeloma dysregulates both FGFR3 and a novel gene, MMSET, resulting in IgH/MMSET hybrid transcripts. Blood. 1998;92:3025-3034. [PubMed] |
| 14. | Min DJ, Ezponda T, Kim MK, Will CM, Martinez-Garcia E, Popovic R, Basrur V, Elenitoba-Johnson KS, Licht JD. MMSET stimulates myeloma cell growth through microRNA-mediated modulation of c-MYC. Leukemia. 2013;27:686-694. [PubMed] [DOI] |
| 15. | Asangani IA, Ateeq B, Cao Q, Dodson L, Pandhi M, Kunju LP, Mehra R, Lonigro RJ, Siddiqui J, Palanisamy N. Characterization of the EZH2-MMSET histone methyltransferase regulatory axis in cancer. Mol Cell. 2013;49:80-93. [PubMed] [DOI] |
| 16. | Hernando H, Gelato KA, Lesche R, Beckmann G, Koehr S, Otto S, Steigemann P, Stresemann C. EZH2 Inhibition Blocks Multiple Myeloma Cell Growth through Upregulation of Epithelial Tumor Suppressor Genes. Mol Cancer Ther. 2016;15:287-298. [PubMed] [DOI] |
| 17. | Popovic R, Martinez-Garcia E, Giannopoulou EG, Zhang Q, Zhang Q, Ezponda T, Shah MY, Zheng Y, Will CM, Small EC. Histone methyltransferase MMSET/NSD2 alters EZH2 binding and reprograms the myeloma epigenome through global and focal changes in H3K36 and H3K27 methylation. PLoS Genet. 2014;10:e1004566. [PubMed] [DOI] |
| 18. | Shah MY, Martinez-Garcia E, Phillip JM, Chambliss AB, Popovic R, Ezponda T, Small EC, Will C, Phillip MP, Neri P. MMSET/WHSC1 enhances DNA damage repair leading to an increase in resistance to chemotherapeutic agents. Oncogene. 2016;35:5905-5915. [PubMed] [DOI] |
| 19. | Hudlebusch HR, Santoni-Rugiu E, Simon R, Ralfkiær E, Rossing HH, Johansen JV, Jørgensen M, Sauter G, Helin K. The histone methyltransferase and putative oncoprotein MMSET is overexpressed in a large variety of human tumors. Clin Cancer Res. 2011;17:2919-2933. [PubMed] [DOI] |
| 20. | Li J, Li T, Lu Y, Shen G, Guo H, Wu J, Lei C, Du F, Zhou F, Zhao X. MiR-2392 suppresses metastasis and epithelial-mesenchymal transition by targeting MAML3 and WHSC1 in gastric cancer. FASEB J. 2017;31:3774-3786. [PubMed] [DOI] |
| 21. | Hudlebusch HR, Skotte J, Santoni-Rugiu E, Zimling ZG, Lees MJ, Simon R, Sauter G, Rota R, De Ioris MA, Quarto M. MMSET is highly expressed and associated with aggressiveness in neuroblastoma. Cancer Res. 2011;71:4226-4235. [PubMed] [DOI] |
| 22. | Ezponda T, Popovic R, Shah MY, Martinez-Garcia E, Zheng Y, Min DJ, Will C, Neri A, Kelleher NL, Yu J. The histone methyltransferase MMSET/WHSC1 activates TWIST1 to promote an epithelial-mesenchymal transition and invasive properties of prostate cancer. Oncogene. 2013;32:2882-2890. [PubMed] [DOI] |
| 23. | Li N, Xue W, Yuan H, Dong B, Ding Y, Liu Y, Jiang M, Kan S, Sun T, Ren J. AKT-mediated stabilization of histone methyltransferase WHSC1 promotes prostate cancer metastasis. J Clin Invest. 2017;127:1284-1302. [PubMed] [DOI] |
| 24. | Lu MH, Fan MF, Yu XD. NSD2 promotes osteosarcoma cell proliferation and metastasis by inhibiting E-cadherin expression. Eur Rev Med Pharmacol Sci. 2017;21:928-936. [PubMed] |
| 25. | Saloura V, Cho HS, Kiyotani K, Alachkar H, Zuo Z, Nakakido M, Tsunoda T, Seiwert T, Lingen M, Licht J. WHSC1 promotes oncogenesis through regulation of NIMA-related kinase-7 in squamous cell carcinoma of the head and neck. Mol Cancer Res. 2015;13:293-304. [PubMed] [DOI] |
| 26. | Evans DL, Zhang H, Ham H, Pei H, Lee S, Kim J, Billadeau DD, Lou Z. MMSET is dynamically regulated during cell-cycle progression and promotes normal DNA replication. Cell Cycle. 2016;15:95-105. [PubMed] [DOI] |
| 27. | Pei H, Zhang L, Luo K, Qin Y, Chesi M, Fei F, Bergsagel PL, Wang L, You Z, Lou Z. MMSET regulates histone H4K20 methylation and 53BP1 accumulation at DNA damage sites. Nature. 2011;470:124-128. [PubMed] [DOI] |
| 28. | Hajdu I, Ciccia A, Lewis SM, Elledge SJ. Wolf-Hirschhorn syndrome candidate 1 is involved in the cellular response to DNA damage. Proc Natl Acad Sci USA. 2011;108:13130-13134. [PubMed] [DOI] |
| 29. | Yang P, Guo L, Duan ZJ, Tepper CG, Xue L, Chen X, Kung HJ, Gao AC, Zou JX, Chen HW. Histone methyltransferase NSD2/MMSET mediates constitutive NF-κB signaling for cancer cell proliferation, survival, and tumor growth via a feed-forward loop. Mol Cell Biol. 2012;32:3121-3131. [PubMed] [DOI] |
| 30. | Wang GG, Cai L, Pasillas MP, Kamps MP. NUP98-NSD1 links H3K36 methylation to Hox-A gene activation and leukaemogenesis. Nat Cell Biol. 2007;9:804-812. [PubMed] [DOI] |
| 31. | Martinez-Garcia E, Popovic R, Min DJ, Sweet SM, Thomas PM, Zamdborg L, Heffner A, Will C, Lamy L, Staudt LM. The MMSET histone methyl transferase switches global histone methylation and alters gene expression in t(4;14) multiple myeloma cells. Blood. 2011;117:211-220. [PubMed] [DOI] |
| 32. | Furukawa Y, Kikuchi J. Epigenetic mechanisms of cell adhesion-mediated drug resistance in multiple myeloma. Int J Hematol. 2016;104:281-292. [PubMed] [DOI] |
| 33. | Xie Z, Chng WJ. MMSET: role and therapeutic opportunities in multiple myeloma. Biomed Res Int. 2014;2014:636514. [PubMed] [DOI] |