修回日期: 2017-09-04
接受日期: 2017-09-25
在线出版日期: 2017-11-28
探讨环状RNAs(circular RNAs, circRNAs)在恶性肿瘤组织中的潜在诊断价值, 本研究就circRNAs作为恶性肿瘤生物标志物的相关研究进行系统综述.
通过计算机检索中国知网、万方数据库、PubMed和Web of Science 4个数据库, 查找国内外circRNAs在恶性肿瘤组织中表达水平的相关研究, 检索时限从建库至2017-05-05, 由两名研究者独立进行文献筛选、资料提取, 并使用NOS量表独立进行文献质量评价.
最终共纳入文献32篇, 文献质量评价结果显示所有文章NOS评分均为7分. 研究较多的为circRNAs在肝癌、结直肠癌及胃癌的癌组织中的差异性表达. 其中有研究明确指出circRNAs可作为肿瘤标志物, 并且具有较高的灵敏度和特异度.
circRNAs在肿瘤组织中存在表达差异, 可以作为恶性肿瘤的诊断标志物, 具有潜在的诊断价值.
核心提要: 环状RNAs(circular RNAs, circRNAs)是一类较为特殊的内源性非编码RNA, 与肿瘤的发生发展关系密切. 本研究就circRNAs作为恶性肿瘤生物标志物的相关研究进行系统综述, 探讨环状RNAs在恶性肿瘤组织中的差异表达及其潜在的诊断价值.
引文著录: 沈艺, 郭旭东, 丁元杰, 魏文强, 刘芬. 环状RNAs作为肿瘤标志物的系统综述. 世界华人消化杂志 2017; 25(33): 2992-2999
Revised: September 4, 2017
Accepted: September 25, 2017
Published online: November 28, 2017
To assess the diagnostic value of circRNAs in detection of tumors through a systematic review.
Articles were searched in CNKI, WanFang database, PubMed and Web of Science databases up to May 5, 2017 with the Title/Abstract words ("circular RNA" or "circRNA" and "cancer" or "tumor" or "carcinoma"). Two investigators extracted the data and assessed the quality of included articles using the Newcastle-Ottawa Scale (NOS) independently.
Thirty-two studies were finally included, which covered hepatocellular carcinoma, gastric cancer, colorectal cancer, and other cancers. The NOS scores of all included studies were 7. Among them, few studies clearly indicated that circRNAs can serve as potential biomarkers and provided the ROC curve, AUC (area under the curve), sensitivity, and specificity.
CircRNAs have significant potential to serve as biomarkers for the diagnosis of tumors. Further studies with larger number of patients will be needed to validate these findings.
- Citation: Shen Y, Guo XD, Ding YJ, Wei WQ, Liu F. Systematic review of circular RNAs as tumor biomarkers for tumor detection. Shijie Huaren Xiaohua Zazhi 2017; 25(33): 2992-2999
- URL: https://www.wjgnet.com/1009-3079/full/v25/i33/2992.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v25.i33.2992
恶性肿瘤是威胁人类生命和社会经济发展的重大疾病之一, 根据GLOBOCAN估计2012年全球约有1410万新发肿瘤病例, 820万人死于恶性肿瘤[1]. 恶性肿瘤的高发病率、高死亡率及其所造成的社会负担成为人们所关注的焦点. 环状RNA(circular RNAs, circRNAs)是一类较为特殊的内源性非编码RNA(non-coding RNAs, ncRNAs), 他不具有5'帽结构和3'多聚腺苷酸尾, 从而比传统的线性RNA更加稳定[2]. 早在20世纪70年代, circRNAs就已经被发现存在于某些高等植物体内, 但是由于当时技术手段所限, circRNAs的研究进展十分缓慢, 人们对他的了解仅仅停留在RNA异常剪接的副产物阶段. 近年来随着分子纯化及高通量测序技术的发展, 人们对于circRNAs有了更深的认识, 截止2015-10, deepBase v2.0数据库[3]已经收集了近14万种circRNAs. 作为ncRNAs的家族成员之一, circRNAs在各种慢性病尤其是恶性肿瘤的发生发展过程中的作用也备受关注. 已有研究[4-13]证实circRNAs能够调控原癌基因或者抑癌基因的表达, 有可能成为恶性肿瘤的诊断标志物. 本研究将对现已公开发表的有关circRNAs在恶性肿瘤中的异常表达情况及其潜在的诊断价值的研究进行系统综述.
本研究主要查找国内外公开发表的有关恶性肿瘤中circRNAs表达水平的文献, 文献筛选时严格按照纳入及排除标准. 纳入标准: (1)文献中所研究的疾病为人类恶性肿瘤; (2)生物样本取自恶性肿瘤患者(癌及癌旁组织)或者健康对照者(正常组织). 排除标准: (1)综述、会议记录等非原始研究类的报道; (2)仅通过circRNAs芯片或高通量测序得到表达量异常的circRNAs而未扩大样本量进行验证.
1.2.1 资料来源: 利用计算机检索中国知网、万方数据库、PubMed和Web of Science共4个数据库, 收集国内外公开发表的所有关于恶性肿瘤中的circRNAs表达情况的研究.
1.2.2 检索策略: 检索词的选择: 英文检索词为"circular RNA or circRNA" and "cancer or tumor or carcinoma", 中文检索词为"环状RNA或circRNA"和"癌症或肿瘤", 检索时限从建库至2017-05-05, 不限制语言及研究类型, 此外手动检索文献中的参考文献及新近发表的综述性文章.
两名研究人员分别根据检索策略在4个数据库中进行独立检索. 随后按照纳入及排除标准对文献进行筛选, 筛选过程中如出现意见不一致的情况, 由第3名研究人员加入讨论, 并以最后讨论的结果为准. 随后对所有符合纳入标准的研究进行数据提取, 主要包括: (1)第一作者; (2)发表年限; (3)研究类型; (4)存在差异表达的circRNAs; (5)癌症类型; (6)circRNA在肿瘤组织中的表达情况; (7)样本来源及数量; (8)相关分子或通路; (9)主要结论.
两名研究者采用Newcastle-Ottawa Scale(NOS)文献质量评价量表中病例对照研究的评价标准, 分别对所纳入的研究进行质量评价. 该评价标准包括"选择"、"可比性"和"暴露"三大部分, 总共8个条目, "选择"和"暴露"中每个条目最多可得1分, 最低不得分, "可比性"的条目最多可得2分, 最低不得分, 总分为9分. 若产生分歧则讨论解决, 必要时根据第三方意见进行商议, 最后达成一致性意见. NOS评分越高代表该研究的质量越好.
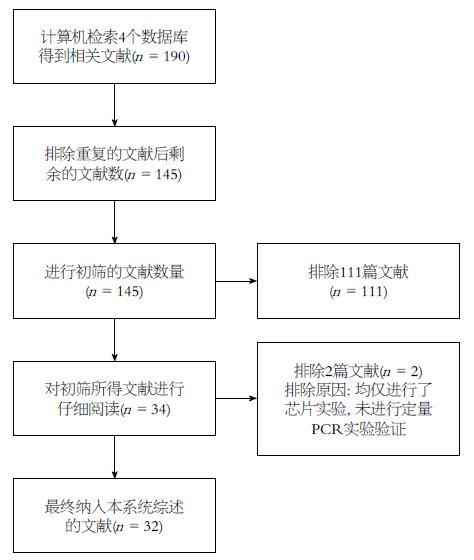
按照制定的检索策略进行检索, 共得到190篇与circRNAs相关的文献, 去掉45篇重复文献后, 通过阅读文献标题及摘要排除111篇文献. 余下34篇文献经全文仔细阅读排除2篇, 最终有32篇原始研究符合纳入标准, 文献筛选流程如图1. 文献质量评价结果显示, 所有纳入文献的NOS得分均为7分, 文献质量较高.
所纳入的32篇研究中, 共报道了42种在肿瘤组织中表达异常的circRNAs, 涉及18种不同类型的恶性肿瘤, 以消化系恶性肿瘤为主(共计19项研究), 其中关于肝癌(hepatocellular carcinoma, HCC)的研究最多(7篇), 而后依次为胃癌(gastric cancer, GC)(5篇), 结直肠癌(colorectal cancer, CRC)(4篇)和食管癌(esophageal squamous cell carcinoma, ESCC)(2篇). 纳入的研究中所用样本均来自于医院的恶性肿瘤患者的冰冻或者石蜡包埋的组织切片, 均进行了circRNAs芯片初筛并经实时荧光定量PCR验证, 部分研究还通过体外实验对circRNAs的分子通路及作用机制进行探索或者通过生物信息学分析对circRNAs可能的靶基因进行预测. 在研究所涉及的42种circRNAs中, 大多数circRNAs都是首次发现在癌组织中存在高表达或者低表达, 而cir-ITCH、ciRS-7(Cdr1as)和Hsa_circ_001988这3种circRNAs在不同类型的癌组织中发现均存在差异性表达. 研究中涉及的circRNAs在肿瘤组织中的表达情况及相关的分子和通路如表1[14-36].
| circRNAs | 癌症类型(n) | circRNAs的表达(高或低) | 相关分子、通路及功能 |
| cir-ITCH | 食管癌1(684)[14] | 癌旁组织高表达 | miR-7, miR-17, miR-214, Wnt/-catenin |
| 结直肠癌1(45)[15] | 高表达 | miR-7, miR-214, miR-20a, Wnt/-catenin | |
| 肺癌1(78)[16] | 低表达 | oncogenicmiR-7, miR-214, Wnt/-catenin | |
| ciRS-7(Cdr1as) | 宫颈癌(47)[17] | 高表达 | miR-7, FAK |
| 正常(35) | |||
| 肝癌1(35)[18] | 高表达 | ciR-7-miR-7-CCNE1/ PIK3CD | |
| 肝癌12(108)[19] | - | miR-7 | |
| PIK3CD/p70S6K/mTOR pathway | |||
| 恶性胶质瘤1(20)[20] | 低表达 | p53 | |
| Hsa_circ_0001649 | 肝癌1(89)[7] | 低表达 | Hsa-miR-1283, Hsa-miR-4310, |
| Hsa-miR-182-3p, Hsa-miR-888-3p, Hsa-miR-4502, Hsa-miR-6811-5p, | |||
| Hsa-miR-6511b-5p Hsa-miR-1972 | |||
| Hsa_circ_0005075 | 肝癌1(30)[8] | 高表达 | Hsa-miR-23b-5p, Hsa-miR-93-3p, |
| Hsa-miR-581, Hsa-miR-23a-5p, | |||
| 细胞黏附因子 | |||
| circZKSCAN1 | 肝癌1(102)[13] | 低表达 | 肝癌细胞的增殖浸润和迁移相关 |
| Hsa_circ_0004018 | 肝癌1(102)[11] | 低表达 | Hsa-miR-30e-5p, Hsa-miR-626 |
| Hsa_circ_0005986 | 肝癌1(81)[21] | 低表达 | miR-129-5p, NotcH1mRNA |
| Hsa_circ_002059 | 胃癌1(101)[6] | 低表达 | - |
| Hsa_circ_0000096 | 胃癌1(101)[5] | 低表达 | miR-224, miR-200a |
| circPVT1 | 胃癌1(187)[22] | 高表达 | miR-125家族 |
| Hsa_circ_0000190 | 胃癌1(104)[4] | 低表达 | - |
| Hsa_circ_0001895 | 胃癌及癌旁1(96)[12] | 低表达 | 细胞分化 |
| 癌前病变(30) | |||
| 正常胃黏膜(35) | |||
| circ_001569 | 结直肠癌1(30)[23] | 高表达 | miR-143, miR-145-E2F5, BAG4, FMNL2 |
| Hsa_circ_0000069 | 结直肠癌1(30)[24] | 高表达 | 促进CRC细胞的增殖、浸润和转移 |
| Hsa_circ_001988 | 结直肠癌1(31)[9] | 低表达 | - |
| 肺癌1(20)[25] | 低表达 | Hsa-miR-382-5p, Hsa-miR-583, CCDC6 | |
| Hsa_circ_0008732 | 基底细胞癌1(6)[26] | 高表达 | OncomiR-1, miR-19-92 family |
| Hsa_circ_0075829 | 高表达 | ||
| Hsa_circ_0005912 | 高表达 | ||
| circ-Amotl1 | 乳腺癌1(16)[27] | 高表达 | circ-Amotl1m RNA, c-myc |
| Hsa_circ_0041103 | 膀胱癌1(40)[28] | 高表达 | circTCF25-miR-103a-3p/miR-107-CDK6 |
| Hsa_circ_0072088 | 高表达 | ||
| Hsa_circ_0005273 | 高表达 | ||
| Hsa_circ_0061265 | 高表达 | ||
| Hsa_circ_0007158 | 低表达 | ||
| Hsa_circ_0082582 | 低表达 | ||
| Hsa_circ_0067934 | 食管癌1(51)[29] | 高表达 | 促进ESCC 细胞的增殖、浸润和转移 |
| Hsa_circ_0001946 | 胰腺导管癌1(20)[30] | 高表达 | Hsa_circ_0005785-miR181a/miR181b-VEGF |
| Hsa_circ_0005397 | 高表达 | ||
| Hsa_circ_0005785 | 低表达 | ||
| Hsa_circ_0006913 | 低表达 | ||
| Hsa_circ_0000257 | 低表达 | ||
| Hsa_circ_0041150 | 低表达 | ||
| Hsa_circ_0008719 | 低表达 | ||
| Hsa_circ_103736 | 皮肤鳞癌1(6)[31] | 高表达 | Hsa-miR-876-5p, Hsa-miR-192-3p, |
| Hsa-mi-34b-3pR-34b-3p, Hsa-miR-34c-3p, | |||
| Hsa-miR-181b-3p | |||
| Hsa_circ_103737 | 高表达 | Hsa-miR-877-3p, Hsa-miR-876-5p, | |
| Hsa-miR-181b-2-3p, Hsa-miR-181b-3p, | |||
| Hsa-miR-627-3p | |||
| Hsa_circ_101555 | 高表达 | Hsa-miR-644a, Hsa-miR-485-5p, | |
| Hsa-miR-889-5p, Hsa-miR-329-5p, | |||
| Hsa-miR-148a-5p | |||
| Hsa_circ_100855 | 喉鳞癌1(52)[32] | 高表达 | - |
| Hsa_circ_104912 | 低表达 | - | |
| circRNA_100290 | 口腔鳞癌1(5)[33] | 高表达 | miR-29家族 |
| Has_circ_0016347 | 骨肉瘤1(6)[34] | 高表达 | miR-214 |
| Hsa_circ_0004277 | 急性髓系白血病患者骨髓(107)[35] | 低表达 | Hsa-miR-138-5p, Hsa-miR-30c-1-3p Hsa-miR-892b, Hsa-miR-571 |
| 健康对照骨髓(8) | Hsa-miR-328-3p | ||
| circHIAT1 | 肾透明细胞癌1(40)[36] | 高表达 | miR-195-5p/29a-3p/29c-3p |
circRNAs的差异表达在恶性肿瘤中几乎普遍存在, 不同类型的恶性肿瘤具有不同的circRNAs表达谱. ciRS-7(Cdr1as)、Hsa_circ_0001649、Hsa_circ_0005075、circZKSCAN1、Hsa_circ_0004018及Hsa_circ_0005986等circRNAs在肝癌组织中均存在差异性表达. 根据受试者工作特征(receiver operating characteristic, ROC)曲线, Hsa_circ_0005075作为肝癌诊断标志物的灵敏度为83.3%, 特异度为90.0%, ROC曲线下面积(area under curve, AUC)为0.94, 具有较高的诊断价值[8]. Hsa_circ_002059、Hsa_circ_0000096及Hsa_circ_0000190在胃癌组织中均处于低表达的状态: Hsa_circ_0000190作为胃癌的诊断标志物时灵敏度与特异度分别为72.1%和68.3%, AUC = 0.75. Hsa_circ_002059与Hsa_circ_0000096联合诊断胃癌的AUC为0.91, 高于Hsa_circ_002059与Hsa_circ_0000096单独的诊断效能[5,6]. 此外, 同种circRNA在不同类型肿瘤组织中的表达情况也并不完全一致, cir-ITCH在食管癌和肺癌组织中低表达, 但在结直肠癌组织中高表达[14-16]; ciRS-7(Cdr1as)在宫颈癌和肝癌组织中均为高表达状态[17,18], 在恶性胶质瘤组织中却为低表达[20], hsa_circ_001988在肺癌和结直肠癌组织中均为低表达[9,25], 这提示我们同一种circRNA在不同恶性肿瘤的发生发展过程中可能发挥着不同的生物功能.
circRNAs具有特殊的闭合环状结构, 不易受RNA外切酶的影响, 因此具有更好的稳定性, 同时circRNAs具有组织及发育阶段特异性, 在疾病的发生发展过程中发挥着很重要的调节作用. circRNAs芯片以及qRT-PCR等技术已经广泛应用于circRNAs与恶性肿瘤的相关研究. 到目前为止, 已经发现许多circRNAs与恶性肿瘤关系密切. 不同类型的癌组织具有不同的circRNAs表达谱, 同一circRNA在不同类型的癌组织中的表达情况也不尽相同, 目前研究较多的是ciRS-7(CDR1as), 据报道ciRS-7在肝癌、宫颈癌和恶性胶质瘤的癌组织中都存在差异性表达. cir-ITCH与食管癌、结直肠癌和肺癌也有着密切关系. 少部分研究不仅检测了circRNAs在肿瘤组织中的表达水平, 还绘制出ROC曲线, 计算circRNAs作为肿瘤标志物的灵敏度、特异度和AUC, 并且有研究表明circRNAs的联合诊断胃癌时的AUC值达到0.9以上, 诊断效能明显优于单一circRNA分子[5,6]. Hsa_circ_002059不仅在胃癌组织中表达下调, 其低表达状态还与胃癌的TNM分期及转移有着密切关联[6]. 提示circRNAs对于恶性肿瘤的调节作用具有特异性, 很可能作为恶性肿瘤的生物标志物, 发挥潜在的诊断价值. 然而本研究并未对纳入研究的样本量进行限制, 个别研究的样本量太少, 结果的可靠性低, 仍需进一步扩大样本量研究.
近来有研究[37]指出, circRNAs作为一种内源性非编码RNA分子, 在外泌体中含量丰富且稳定, 可能作为检测恶性肿瘤的潜在标志物. 缺氧状态下, 机体所产生的外泌体中的RNA和蛋白质会发生变化, 影响恶性肿瘤的发生发展过程[38]. 体外研究[39]结果表明, 结肠癌细胞所产生的外泌体中, circRNAs的表达下调, 且外泌体中的circRNAs的变化水平较细胞内更为显著, 提示外泌体中circRNAs有可能作为检测肿瘤发生发展的重要标志物. Chen等[4]发现circRNAs除了在癌组织中表达异常, 在肿瘤患者的血浆中也存在差异性表达, 这也为circRNAs作为肿瘤早期筛查或诊断的生物标志物提供了可能.目前普遍认为circRNAs能够作为微小RNA(microRNA, miRNA)的分子海绵, 竞争性结合miRNA, 降低miRNA活性. Hansen等[40]在人及鼠脑中发现了一种circRNA, 并将其命名为ciRS-7(circular RNA sponge for miR-7), 该circRNA包含了超过70个miRNA选择性结合位点, 能够强烈抑制miR-7的活性, 进而激活miR-7的下游基因, 并通过实验证实在小鼠脑中cirRS-7与miR-7具有重叠共表达性. 同时还发现了性别决定区域(sex-determining region Y)circRNA可作为miR-138的分子海绵. 郝臻凤[17]通过实验发现在宫颈癌Hela细胞中ciRS-7的过表达造成miR-7的表达量降低, 这提示ciRS-7能够作为miR-7的分子海绵竞争性抑制miR-7的活性. 此外Liu等[41]发现在软骨退化过程中, 软骨细胞外基质相关circRNA(circRNA-CER)通过作为miR-136的分子海绵调节MMP13基因的表达. Peng等[42]发现在先天性巨结肠病中cir-ZNF609通过作为miR-150-5P的分子海绵调节AKT3基因的表达. Chen等[33]发现在口腔鳞癌中circRNA_100290可作为miR-29家族的分子海绵而发挥作用. 以上提示circRNAs作为miRNA分子海绵可能是普遍现象. 然而有些研究所报道的circRNAs的靶基因通过靶基因预测软件获得, 极少进行了实验验证, 加之不同靶基因预测软件的算法不尽相同, 降低了结果的可靠性. 尽管研究存在不足但已有许多研究证实circRNAs确实有潜力作为肿瘤的诊断或预后标志物[4-9,11-13,19,22].
circRNAs在肿瘤组织中存在特异性表达, 可作为肿瘤潜在标志物, 且circRNAs作为miRNA的分子海绵竞争性结合miRNA, 降低miRNA活性的机制已被多项研究反复验证, 其中涉及的相关分子通路, 可以为研究相关治疗靶点提供新的思路. 虽然相较于传统的线性RNA, circRNAs的研究仍处于起步状态, 还有很多的问题尚待解决, 然而随着研究的深入将会有更多的circRNAs被发现, 其功能及机制也会更加明确, 将会为复杂疾病的精确诊断、靶向治疗提供重要的分子基础.
环状RNA(circular RNAs, circRNAs)是一类较为特殊的内源性非编码RNA(non-coding RNAs, ncRNAs), 比传统的线性RNA更加稳定. circRNAs在恶性肿瘤的发生发展过程中发挥着作用, 近来研究发现circRNAs在恶性肿瘤中的表达水平发生特异性改变, 对于恶性肿瘤的诊断具有重要意义.
近年来随着分子纯化及高通量测序技术的发展, 人们对于circRNAs有了更深的认识, circRNAs在恶性肿瘤的发生发展过程中的作用备受关注. 已有研究表明circRNAs能够调控原癌基因或者抑癌基因的表达, 有可能成为恶性肿瘤的诊断标志物.
现有研究指出, circRNAs在肿瘤患者的癌组织与癌旁组织中的表达量存在显著差异, cir-ITCH、ciRS-7(Cdr1as)等在不同类型的肿瘤组织中均存在差异性表达, circRNAs能够作用于相关的分子通路, 影响恶性肿瘤的发生与发展.
本研究就近年来circRNAs作为恶性肿瘤标志物的相关研究作出系统综述, 对所有研究进行文献筛选及质量评价, 对符合纳入标准的研究进行总结, 概括circRNAs作为恶性肿瘤生物标志物的研究进展.
circRNAs逐渐渗透到恶性肿瘤的研究中, circRNAs在肿瘤组织中的差异性表达, 对于临床上恶性肿瘤的诊断具有一定的参考价值, circRNAs调控的相关分子通路, 可以为恶性肿瘤的靶向治疗提供新的思路.
微小RNA(miRNA): 一类来自于内源性染色体上的长度为21-23个核苷酸的非编码单链小分子核酸, 它能够作用于生物体内的相关信号通路, 参与细胞的增殖、分化、衰老和凋亡过程, 调控细胞周期, 与恶性肿瘤的发生发展密切相关;
外泌体(exosome): 由多种活细胞分泌的30-120 nm的囊泡小体, 内含有蛋白质和RNA等多种组分, 能够参与细胞间的物质交换和信息交流, 与机体内的多种生理及病理过程密切相关.
本文对circRNAs在不同恶性肿瘤中的表达情况及其与相关分子和信号通路的关联进行了文献综述和梳理, 提出circRNAs可作为恶性肿瘤的生物标志物, 在临床肿瘤诊断和预后判断中具有潜在的应用价值, 对相关临床和基础研究均具有一定的指导意义.
冯继红, 副教授, 遵义医学院附属医院肿瘤医院腹部肿瘤科; 陆斌, 副教授, 第二军医大学肿瘤学专业; 田华, 教授, 上海交通大学医学院附属仁济医院上海市肿瘤研究所
手稿来源: 自由投稿
学科分类: 胃肠病学和肝病学
手稿来源地: 北京市
同行评议报告分类
A级 (优秀): 0
B级 (非常好): B, B
C级 (良好): 0
D级 (一般): D
E级 (差): 0
编辑:马亚娟 电编:李瑞芳
| 1. | Torre LA, Bray F, Siegel RL, Ferlay J, Lortet-Tieulent J, Jemal A. Global cancer statistics, 2012. CA Cancer J Clin. 2015;65:87-108. [PubMed] [DOI] |
| 3. | Zheng LL, Li JH, Wu J, Sun WJ, Liu S, Wang ZL, Zhou H, Yang JH, Qu LH. deepBase v2.0: identification, expression, evolution and function of small RNAs, LncRNAs and circular RNAs from deep-sequencing data. Nucleic Acids Res. 2016;44:D196-D202. [PubMed] [DOI] |
| 4. | Chen S, Li T, Zhao Q, Xiao B, Guo J. Using circular RNA hsa_circ_0000190 as a new biomarker in the diagnosis of gastric cancer. Clin Chim Acta. 2017;466:167-171. [PubMed] [DOI] |
| 5. | Li P, Chen H, Chen S, Mo X, Li T, Xiao B, Yu R, Guo J. Circular RNA 0000096 affects cell growth and migration in gastric cancer. Br J Cancer. 2017;116:626-633. [PubMed] [DOI] |
| 6. | Li P, Chen S, Chen H, Mo X, Li T, Shao Y, Xiao B, Guo J. Using circular RNA as a novel type of biomarker in the screening of gastric cancer. Clin Chim Acta. 2015;444:132-136. [PubMed] [DOI] |
| 7. | Qin M, Liu G, Huo X, Tao X, Sun X, Ge Z, Yang J, Fan J, Liu L, Qin W. Hsa_circ_0001649: A circular RNA and potential novel biomarker for hepatocellular carcinoma. Cancer Biomark. 2016;16:161-169. [PubMed] [DOI] |
| 8. | Shang X, Li G, Liu H, Li T, Liu J, Zhao Q, Wang C. Comprehensive Circular RNA Profiling Reveals That hsa_circ_0005075, a New Circular RNA Biomarker, Is Involved in Hepatocellular Crcinoma Development. Medicine (Baltimore). 2016;95:e3811. [PubMed] [DOI] |
| 9. | Wang X, Zhang Y, Huang L, Zhang J, Pan F, Li B, Yan Y, Jia B, Liu H, Li S. Decreased expression of hsa_circ_001988 in colorectal cancer and its clinical significances. Int J Clin Exp Pathol. 2015;8:16020-16025. [PubMed] |
| 10. | Chen LL. The biogenesis and emerging roles of circular RNAs. Nat Rev Mol Cell Biol. 2016;17:205-211. [PubMed] [DOI] |
| 11. | Fu L, Yao T, Chen Q, Mo X, Hu Y, Guo J. Screening differential circular RNA expression profiles reveals hsa_circ_0004018 is associated with hepatocellular carcinoma. Oncotarget. 2017;8:58405-58416. [PubMed] [DOI] |
| 12. | Shao Y, Chen L, Lu R, Zhang X, Xiao B, Ye G, Guo J. Decreased expression of hsa_circ_0001895 in human gastric cancer and its clinical significances. Tumour Biol. 2017;39:1010428317699125. [PubMed] [DOI] |
| 13. | Yao Z, Luo J, Hu K, Lin J, Huang H, Wang Q, Zhang P, Xiong Z, He C, Huang Z. ZKSCAN1 gene and its related circular RNA (circZKSCAN1) both inhibit hepatocellular carcinoma cell growth, migration, and invasion but through different signaling pathways. Mol Oncol. 2017;11:422-437. [PubMed] [DOI] |
| 14. | Li F, Zhang L, Li W, Deng J, Zheng J, An M, Lu J, Zhou Y. Circular RNA ITCH has inhibitory effect on ESCC by suppressing the Wnt/β-catenin pathway. Oncotarget. 2015;6:6001-6013. [PubMed] [DOI] |
| 15. | Huang G, Zhu H, Shi Y, Wu W, Cai H, Chen X. cir-ITCH plays an inhibitory role in colorectal cancer by regulating the Wnt/β-catenin pathway. PLoS One. 2015;10:e0131225. [PubMed] [DOI] |
| 16. | Wan L, Zhang L, Fan K, Cheng ZX, Sun QC, Wang JJ. Circular RNA-ITCH Suppresses Lung Cancer Proliferation via Inhibiting the Wnt/β-Catenin Pathway. Biomed Res Int. 2016;2016:1579490. [PubMed] [DOI] |
| 17. | 郝 臻凤. ciRS-7/miR-7/FAK调节轴对宫颈癌侵袭迁移的影响及紫草素干预机制的研究. 扬州: 扬州大学 2015; . |
| 18. | Yu L, Gong X, Sun L, Zhou Q, Lu B, Zhu L. The Circular RNA Cdr1as Act as an Oncogene in Hepatocellular Carcinoma through Targeting miR-7 Expression. PLoS One. 2016;11:e0158347. [PubMed] [DOI] |
| 19. | Xu L, Zhang M, Zheng X, Yi P, Lan C, Xu M. The circular RNA ciRS-7 (Cdr1as) acts as a risk factor of hepatic microvascular invasion in hepatocellular carcinoma. J Cancer Res Clin Oncol. 2017;143:17-27. [PubMed] [DOI] |
| 20. | 娄 佳成. 环状RNA CDR1as调控胶质瘤恶性生长的分子机制. 大连: 大连医科大学 2015; . |
| 21. | Fu L, Chen Q, Yao T, Li T, Ying S, Hu Y, Guo J. Hsa_circ_0005986 inhibits carcinogenesis by acting as a miR-129-5p sponge and is used as a novel biomarker for hepatocellular carcinoma. Oncotarget. 2017;8:43878-43888. [PubMed] [DOI] |
| 22. | Chen J, Li Y, Zheng Q, Bao C, He J, Chen B, Lyu D, Zheng B, Xu Y, Long Z. Circular RNA profile identifies circPVT1 as a proliferative factor and prognostic marker in gastric cancer. Cancer Lett. 2017;388:208-219. [PubMed] [DOI] |
| 23. | Xie H, Ren X, Xin S, Lan X, Lu G, Lin Y, Yang S, Zeng Z, Liao W, Ding YQ. Emerging roles of circRNA_001569 targeting miR-145 in the proliferation and invasion of colorectal cancer. Oncotarget. 2016;7:26680-26691. [PubMed] [DOI] |
| 24. | Guo JN, Li J, Zhu CL, Feng WT, Shao JX, Wan L, Huang MD, He JD. Comprehensive profile of differentially expressed circular RNAs reveals that hsa_circ_0000069 is upregulated and promotes cell proliferation, migration, and invasion in colorectal cancer. Onco Targets Ther. 2016;9:7451-7458. [PubMed] [DOI] |
| 26. | Sand M, Bechara FG, Sand D, Gambichler T, Hahn SA, Bromba M, Stockfleth E, Hessam S. Circular RNA expression in basal cell carcinoma. Epigenomics. 2016;8:619-632. [PubMed] [DOI] |
| 27. | 杨 琪. 环状RNA AMOTL-1上调增强人乳腺癌细胞体内外恶性行为及其机制的研究. 长春: 吉林大学 2016; . |
| 28. | Zhong Z, Lv M, Chen J. Screening differential circular RNA expression profiles reveals the regulatory role of circTCF25-miR-103a-3p/miR-107-CDK6 pathway in bladder carcinoma. Sci Rep. 2016;6:30919. [PubMed] [DOI] |
| 29. | Xia W, Qiu M, Chen R, Wang S, Leng X, Wang J, Xu Y, Hu J, Dong G, Xu PL. Circular RNA has_circ_0067934 is upregulated in esophageal squamous cell carcinoma and promoted proliferation. Sci Rep. 2016;6:35576. [PubMed] [DOI] |
| 30. | Li H, Hao X, Wang H, Liu Z, He Y, Pu M, Zhang H, Yu H, Duan J, Qu S. Circular RNA Expression Profile of Pancreatic Ductal Adenocarcinoma Revealed by Microarray. Cell Physiol Biochem. 2016;40:1334-1344. [PubMed] [DOI] |
| 31. | Sand M, Bechara FG, Gambichler T, Sand D, Bromba M, Hahn SA, Stockfleth E, Hessam S. Circular RNA expression in cutaneous squamous cell carcinoma. J Dermatol Sci. 2016;83:210-218. [PubMed] [DOI] |
| 32. | Xuan L, Qu L, Zhou H, Wang P, Yu H, Wu T, Wang X, Li Q, Tian L, Liu M. Circular RNA: a novel biomarker for progressive laryngeal cancer. Am J Transl Res. 2016;8:932-939. [PubMed] |
| 33. | Chen L, Zhang S, Wu J, Cui J, Zhong L, Zeng L, Ge S. circRNA_100290 plays a role in oral cancer by functioning as a sponge of the miR-29 family. Oncogene. 2017;36:4551-4561. [PubMed] [DOI] |
| 34. | Jin H, Jin X, Zhang H, Wang W. Circular RNA hsa-circ-0016347 promotes proliferation, invasion and metastasis of osteosarcoma cells. Oncotarget. 2017;8:25571-25581. [PubMed] [DOI] |
| 35. | Li W, Zhong C, Jiao J, Li P, Cui B, Ji C, Ma D. Characterization of hsa_circ_0004277 as a New Biomarker for Acute Myeloid Leukemia via Circular RNA Profile and Bioinformatics Analysis. Int J Mol Sci. 2017;18:pii E597. [PubMed] [DOI] |
| 36. | Wang K, Sun Y, Tao W, Fei X, Chang C. Androgen receptor (AR) promotes clear cell renal cell carcinoma (ccRCC) migration and invasion via altering the circHIAT1/miR-195-5p/29a-3p/29c-3p/CDC42 signals. Cancer Lett. 2017;394:1-12. [PubMed] [DOI] |
| 37. | Li Y, Zheng Q, Bao C, Li S, Guo W, Zhao J, Chen D, Gu J, He X, Huang S. Circular RNA is enriched and stable in exosomes: a promising biomarker for cancer diagnosis. Cell Res. 2015;25:981-984. [PubMed] [DOI] |
| 38. | Qiu GZ, Jin MZ, Dai JX, Sun W, Feng JH, Jin WL. Reprogramming of the Tumor in the Hypoxic Niche: The Emerging Concept and Associated Therapeutic Strategies. J Fish Biology. 2017;38:669-686. [DOI] |
| 39. | Dou Y, Cha DJ, Franklin JL, Higginbotham JN, Jeppesen DK, Weaver AM, Prasad N, Levy S, Coffey RJ, Patton JG. Circular RNAs are down-regulated in KRAS mutant colon cancer cells and can be transferred to exosomes. Sci Rep. 2016;6:37982. [PubMed] [DOI] |
| 40. | Hansen TB, Jensen TI, Clausen BH, Bramsen JB, Finsen B, Damgaard CK, Kjems J. Natural RNA circles function as efficient microRNA sponges. Nature. 2013;495:384-388. [PubMed] [DOI] |
| 41. | Liu Q, Zhang X, Hu X, Dai L, Fu X, Zhang J, Ao Y. Circular RNA Related to the Chondrocyte ECM Regulates MMP13 Expression by Functioning as a MiR-136 'Sponge' in Human Cartilage Degradation. Sci Rep. 2016;6:22572. [PubMed] [DOI] |
| 42. | Peng L, Chen G, Zhu Z, Shen Z, Du C, Zang R, Su Y, Xie H, Li H, Xu X. Circular RNA ZNF609 functions as a competitive endogenous RNA to regulate AKT3 expression by sponging miR-150-5p in Hirschsprung's disease. Oncotarget. 2017;8:808-818. [PubMed] [DOI] |