修回日期: 2013-07-05
接受日期: 2013-07-15
在线出版日期: 2013-08-18
目的: 筛选上调尾型同源盒基因2(caudal type homeobox gene 2, CDX2)基因表达时人的胃癌细胞SGC-7901中差异表达的miRNA并预测其靶基因, 并观察其对胃癌细胞生物学行为的影响.
方法: 提取瞬时转染48 h后空载体组(转染PEGFP-N1组)和转染组(转染PEGFP-N1-CDX2组)细胞的总RNA, 应用miRNA芯片检测差异表达的miRNA, 并通过Miranda、TargetScan、Mirtarget2软件预测其靶基因, 通过Gene Ontology(GO)和KEGG pathway分析了解靶基因功能. CCK8法检测细胞增殖能力, 划痕试验、Transwell小室、细胞黏附实验检测各组细胞体外迁移黏附能力.
结果: PEGFP-N1-CDX2组较空载体组有59种差异表达的miRNA, 其中25种miRNA发生2倍以上表达上调, 34种miRNA发生2倍以上表达下调. 通过Gene Ontology(GO)和KEGG pathway分析得到部分miRNA靶基因功能, 这些靶基因参与了肿瘤的发生、发展、转移及预后. 与对照组相比, PEGFP-N1-CDX2组的胃癌细胞生长、迁移和黏附能力均明显受到抑制(P<0.05).
结论: 上调CDX2基因表达可明显抑制人胃癌细胞SGC-7901的生长, 抑制其迁移黏附能力, CDX2基因的抗肿瘤作用可能与miRNA有关.
核心提示: 近年来发现miRNA与肿瘤的发病密切相关, 参与多种肿瘤细胞增殖、凋亡、侵袭或血管形成等过程. 上调尾型同源盒基因2(caudal type homeobox gene 2, CDX2)基因表达可明显抑制人胃癌细胞SGC-7901的生长, 抑制其迁移黏附能力, CDX2基因的抗肿瘤作用可能与miRNA有关.
引文著录: 张健锋, 董丽娟, 蒋伟, 张弘, 丁伟峰, 周国雄, 毛振彪. 上调CDX2基因表达对人胃癌SGC-7901细胞miRNA表达谱及生物学功能的影响. 世界华人消化杂志 2013; 21(23): 2241-2249
Revised: July 5, 2013
Accepted: July 15, 2013
Published online: August 18, 2013
AIM: To screen differentially expressed miRNAs in human gastric carcinoma cells with up-regulated caudal type homeobox gene 2 (CDX2) expression and to analyze their effect on biological behavior of cells.
METHODS: A recombinant eukaryotic expression plasmid carrying the CDX2 gene was constructed. SGC-7901 cells were divided into three groups: non-transfected cells, cells transfected with PEGFP-N1 or PEGFP-N1-CDX2. The expression of GFP was observed by fluorescent microscopy. Expression of CDX2 mRNA and EGFP-CDX2 fusion protein was detected by qRT-PCR and Western blot, respectively. Cell proliferation was measured using CCK8 assay. In vitro cell migration and adhesion were measured by cell scratch assay, Transwell assay and cell adhesion assay. Differentially expressed miRNAs were detected using a miRNA chip, and their target genes were forecasted using Miranda, TargetScan and Mirtarget2 software.
RESULTS: The eukaryotic expression vector PEGFP-N1-CDX2 was successfully constructed. The expression levels of CDX2 mRNA and protein were higher in SGC-7901 transfected with PEGFP-N1-CDX2 than in control cells. In SGC-7901 cells transfected with PEGFP-N1-CDX2, cell proliferation, invasion and adhesion were significantly inhibited (all P < 0.05) compared with non-transfected cell or cells transfected with PEGFP-N1. Of 59 identified differentially expressed miRNAs between cells transfected PEGFP-N1-CDX2 and those transfected with PEGFP-N1, 25 had > 2-fold up-regulation and 34 had > 2-fold down-regulation in cells transfected with PEGFP-N1-CDX2. Many target genes of these differentially expressed miRNAs were predicted using miRNA target predication tools.
CONCLUSION: Up-regulation of CDX2 expression inhibits the growth, migration and adhesion of SGC-7901 cells. The antitumor effect of CDX2 may be associated with miRNA expression.
- Citation: Zhang JF, Dong LJ, Jiang W, Zhang H, Ding WF, Zhou GX, Mao ZB. Effect of up-regulation of CDX2 expression on miRNA expression profiles and biological behavior in gastric cancer SGC-7901 cells. Shijie Huaren Xiaohua Zazhi 2013; 21(23): 2241-2249
- URL: https://www.wjgnet.com/1009-3079/full/v21/i23/2241.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i23.2241
胃癌是常见的消化系恶性肿瘤之一, 在我国其发病率占消化系恶性肿瘤的首位, 严重危害着人类的生命健康. 胃癌的发生发展是一个多阶段、多步骤、涉及多基因的病理过程, 其确切的发病机制至今尚不清楚[1,2].
尾型同源盒基因2(caudal type homeobox gene 2, CDX2)是一种特异性核转录因子, 在正常胃黏膜上皮中CDX2阴性表达, 在慢性萎缩性胃炎伴肠化的胃黏膜上皮中, 随着杯状细胞等一系列肠道特异性细胞的出现, CDX2的表达明显增强, 提示CDX2的表达是胃黏膜癌变过程中的早期事件[3,4]. 研究发现CDX2能够抑制肠化生组织向胃癌的转变, 因此推测他是抑制胃癌侵袭和转移的重要因素[5-7]. 近年来发现miRNA与肿瘤的发病密切相关, 参与多种肿瘤细胞增殖、凋亡、侵袭或血管形成等过程[8,9]. 本项目通过分子克隆、实时荧光定量PCR、Western blot、CCK8法、细胞划痕试验、Transwell小室、细胞黏附实验等现代分子生物学技术、在体外环境的试验中研究上调CDX2基因表达对胃癌细胞生物学行为的影响, 并通过基因芯片扫描技术筛选上调CDX2基因表达时差异表达的miRNA, 预测其靶基因, 分析CDX2基因在胃癌发生发展中的可能作用及机制.
人胃腺癌细胞株AGS、MKN-45和SGC-7901均购自中国科学院细胞库. 3种人胃癌细胞株均在含10%胎牛血清的RPMI 1640培养液、37 ℃、5%CO2培养箱内培养. PEGFP-N1、大肠杆菌感受态细胞由南通大学外科实验室提供. TRIzol、PrimeScript RT reagent Kit、SYBR Premix Ex TaqTM(Perfect Real Time)(日本TaKaRa公司), 引物合成(上海生工生物工程有限公司), CDX2抗体(Santa Cruz公司), 辣根过氧化物酶标记山羊抗小鼠IgG(H+L)(碧云天生物技术研究所), T4 DNA连接酶、限制性核酸内切酶XhoⅠ、HindⅢ(Fermentas公司), 纯化回收试剂盒(Omega公司), 质粒抽提试剂盒(Axygen公司), TurboFectTMin vitro Transfection Reagent(Fermentas公司), CCK8(日本同仁), Matrigel胶(BD公司), 结晶紫染色液(碧云天生物技术研究所), 多聚甲醛(Sigma公司), Transwell小室(Costar公司), 多功能酶标仪(Thermo公司), miRCURYTM Hy3TM/Hy5TM Power标记试剂盒(丹麦Exiqon公司), Axon GenePix4000B芯片扫描仪(美国Molecular Devices公司).
1.2.1 CDX2低表达: 胃癌细胞株筛选: 取人胃癌细胞株AGS、SGC-7901、MKN-45各组对数生长期的细胞, TRIzol试剂抽提细胞总RNA. 按说明书分别进行逆转录合成cDNA和实时荧光定量PCR. (1)逆转录反应: 20 µL反应体系含5×PrimeScript Buffer 4 µL、1.5 µL PrimeScript RT Enzyme Mix 1 µL、1 µL Oligo dT Primer(50 µmol/L)、1 µL Random 6 mers(100 µmol/L)、2.7 µL RNA模板、10.3 µL RNase Free ddH2O; 逆转录反应37 ℃ 15 min, 逆转录酶失活85 ℃ 5 s; (2)实时荧光定量PCR: CDX2引物由Primer 5.0软件设计CDX2 mRNA上游引物序列: 5'-CGCCGCAGAACTTCGTCAG-3', 下游引物序列: 5'-CGTAGCCATTCCAGTCCTCCC-3', β-actin上游引物序列: 5'-TGACGTGGACATCCGCAAAG-3', 下游引物序列: 5'-CTGGAAGGTGGACAGCGAGG-3'. 20 µL反应体系含10 µL SYBRPremix Ex TaqTM(2×)、0.4 µL PCR Forward Primer(10 µmol/L)、0.4 µL PCR Reverse Primer(10 µmol/L)、0.4 µL ROX Reference Dye(50×)、2.0 µL DNA模板、6.8 µL dH2O. 反应条件: 95 ℃ 30 s预变性, 95 ℃ 5 s, 60 ℃ 34 s, 共40个循环. 实验结果重复3次. 扩增完毕后分析溶解曲线, 用2-∆∆Ct法分析数据结果.
1.2.2 PEGFP-N1-CDX2真核表达质粒的构建: 提取人胃癌细胞株AGS总RNA, 按逆转录试剂盒操作步骤得到CDX2 cDNA, 从Genbank中查找CDX2开放阅读框(942 bp), 用Primer Primier 5.0软件设计带有XhoⅠ和HindⅢ酶切位点的引物. 上游引物: 5'-TTTTCTCGAGATGTACGTGAGCTACCTCCTG-3', 下游引物: 5'-TTTTAAGCTTCTGGGTGACGGTGGGGTTTAG-3'. 以CDX2 cDNA为模板进行PCR反应扩增目的基因, 反应产物为5'端带有XhoⅠ位点, 3'端带有HindⅢ位点的目的基因片段. PCR扩增反应条件: 94 ℃预变性2 min, 94 ℃变性1 min, 55 ℃退火1 min, 72 ℃延伸2 min, 共25个循环, 最后72 ℃延伸5 min. 取PCR反应产物在1%琼脂糖凝胶中电泳分析, 得到一条特异性区带, 切取目的条带, 将含目的片段的琼脂糖凝胶经DNA纯化回收试剂盒回收. 用XhoⅠ和HindⅢ分别双酶切回收的CDX2片段和PEGFP-N1空载体, 酶切反应体系如下: CDX2(或PEGFP-N1)40 µL, 10×buffer 10 µL, XhoⅠ 1 µL, HindⅢ 1 µL, 37 ℃酶切消化12-16 h, 后行琼脂糖电泳后用纯化回收试剂盒回收. 接下来采用T4 DNA连接酶将经酶切的目的片段及经相同酶切的PEGFP-N1空载体连接, 将连接产物转化大肠杆菌, 在含有卡那霉素的LB培养板中培养16 h. 挑取菌落进行质粒扩增, 提取质粒. 用XhoⅠ和HindⅢ双酶切提取的重组质粒, 反应体系如下: PEGFP-N1-CDX2 40 µL, 10×buffer 10 µL, XhoⅠ 1 µL, HindⅢ 1 µL, 37 ℃酶切消化12-16 h. 取酶切产物5 µL进行琼脂糖凝胶电泳鉴定, 将双酶切鉴定阳性的重组载体送Invitrogen公司进行测序.
1.2.3 重组质粒在SGC-7901细胞中的表达与检测: 实验分为3组: 未转染组, 转染PEGFP-N1组, 转染PEGFP-N1-CDX2组. 取对数生长期细胞重悬, 以2.5×105/孔接种于六孔板上, 待细胞融合达70%-90%时用阳离子聚合物TurboFectTMin vitroTransfection Reagent介导转染, 4 h后换液继续培养. 转染24 h后倒置荧光显微镜下观察增强型绿色荧光蛋白(enhanced green fluorescent protein, EGFP)的表达情况. 分别取转染48 h后3组细胞应用实时荧光定量PCR检测mRNA表达, 操作同1.2.1. Western blot检测融合蛋白表达: 用细胞裂解液分别提取转染48 h后3组细胞的蛋白质, 紫外分光光度计测定蛋白质浓度, 以1:4比例与5×样品缓冲液混匀, 煮沸5 min. 将样品(60 µg/lane)在10%SDS-PAGE胶上电泳(80 V, 40 min, 后100 V, 90 min). 后通过湿转(恒流300 mA, 2 h)转印到PVDF膜上, 在含5%脱脂奶粉的TBS封闭液中封闭2 h, 按预染Marker标记的分子量剪裁转印膜, 加入CDX2抗体(稀释度均为1:100), 4 ℃过夜, 加入辣根过氧化物酶标记山羊抗小鼠IgG(H+L)(稀释度均为1:5000)室温孵育2 h, 显色、图像采集及分析处理, 以β-actin为内参照. 实验重复3次.
1.2.4 CCK8法观察细胞增殖: 于96孔板中按 3×103/孔接种转染PEGFP-N1、转染PEGFP-N1-CDX2和未转染的SGC-7901细胞, 每组设6个复孔, 分为0、24、48、72 h 4个时间段观测, 每孔加入10 µL CCK8液, 37 ℃、5%CO2培养箱培养3 h, 酶标仪测定各孔A450值. 计算细胞生长抑制率(IR) = (1-实验组A值/对照组A值)×100%.
1.2.5 划痕试验观察细胞迁移能力: 取对数生长期细胞重悬, 以2.5×105/孔接种于六孔板上, 待细胞融合达70%-90%时用阳离子聚合物TurboFectTM in vitro Transfection Reagent介导转染48 h后, 将各组细胞PBS洗2遍, 用无菌200 µL枪尖在单细胞层上划痕, PBS将脱壁的细胞洗掉, 用含1%胎牛血清的RPMI 1640培养液, 放入培养箱继续培养, 分别于划痕后0、24 h在倒置显微镜下每孔随机取4个视野拍照, 观察划痕的修复情况, 划痕修复率 = [(0 h划痕宽度-24 h划痕宽度)/0 h划痕宽度]×100%.
1.2.6 Transwell观察细胞迁移能力: 收集瞬时转染48 h后各组细胞重悬, 1×105/孔接种于Transwell小室的上室, 上室为无血清RPMI 1640培养液, 下室为含20%胎牛血清的RPMI 1640培养液600 µL, 37 ℃、5%CO2培养箱中培养24 h, 用棉签轻轻拭去上室滤膜内侧面贴壁细胞, PBS洗两遍, 4%的多聚甲醛固定10 min, 吸去固定液, 将膜风干, 每孔加入600 µL 0.1%结晶紫染液, 室温置放30 min, 除去染色液, PBS洗2遍, 将上室取出, 显微镜下计数膜背面迁移的细胞数, 计数每张膜的中央部分和周围部分随机3个视野(共15个视野), 计算平均值.
1.2.7 CCK8法检测细胞黏附能力: 取96孔培养板, 覆以Matrigel胶8 µg/孔, 4 ℃过夜, 使用前37 ℃孵育1 h, 无血清RPMI 1640洗2次, 加入瞬时转染48 h后各组细胞, 5×103/孔, 37 ℃孵育, 分别于1、2、3、4 h弃培养液, PBS洗2遍, 每孔各加入100 µL无血清RPMI 1640及10 µL CCK8液, 37 ℃、5%CO2培养箱培养3 h, 酶标仪测定各孔A450值, 每组细胞每个时间段各设6个复孔.
1.2.8 miRNA表达谱的检测: 收集瞬时转染48 h后空载体组(转染PEGFP-N1组)及转染组(转染PEGFP-N1-CDX2组)细胞, 细胞数在1×106-1×107, PBS洗2遍, 用TRIzol和miRNeasy mini kit 提取总RNA, 操作按说明书进行. 应用miRCURYTM Hy3TM/Hy5TM标记试剂盒标记miRNA. 将25 µL Hy3TM标记样品和25 µL杂交缓冲混合物首先95 ℃变性2 min, 冰上孵育2 min, 然后应用12排杂交系统在56 ℃芯片杂交16-20 h, 杂交之后, 取得芯片, 洗涤液洗片, 400 r/min, 5 min离心干燥. 使用Axon GenePix4000B芯片扫描仪扫描芯片的荧光强度. 比较标准化后的数据值, 上调或下调2倍以上即认为存在显著上调或下调趋势.
1.2.9 实时定量PCR验证miRNA: 随机选取hsa-miR-374a和hsa-miR-374b, 实时定量PCR验证miRNA表达谱芯片结果, 引物序列如下: U6: F: 5'-GCTTCGGCAGCACATATACTAAAAT-3', R: 5'-CGCTTCACGAATTTGCGTGTCAT-3'; hsa-miR-374a: GSP: 5'-GGGCTTATAATACAACCTGA-3', R: 5'-TGCGTGTCGTGGAGTC-3'; hsa-miR-374b: GSP: 5'-GGGGATATAATACAACCTGC-3'; R: 5'-CAGTGCGTGTCGTGGAGT-3'. 其中, GSP是对应miRNA的特异引物, R是与RT引物相匹配的引物. 反应条件如下: 95 ℃, 5 min; 40个PCR循环(95 ℃, 10 s; 60 ℃, 20 s; 72 ℃, 20 s; 78 ℃, 20 s). 各样品的目的miRNA和内参(U6)分别进行实时定量PCR反应. 数据采用2-△△Ct法进行分析.
1.2.10 靶基因预测及功能分析: 应用Miranda、TargetScan、Mirtarget2软件分别对其进行目标miRNA的预测, 取同时满足3个软件预测到的基因作为靶基因. 对各个miRNA的靶基因分别进行Gene Ontology(GO)和KEGG pathway分析了解其功能.
统计学处理 所有数据均用统计软件SPSS13.0处理, 计量资料以mean±SD表示, 组间比较采用单因素方差分析, 组间两两比较采用t检验. 芯片图像扫描后导入GenePix Pro 6.0软件, 重复的miRNA和miRNA的强度≥50的所有样品使用中位数进行数据标准化处理. 标准化后差异表达的miRNA鉴定通过的倍数变化进行分析和使用MEV软件进行层次聚类分析. P<0.05为差异有统计学意义.
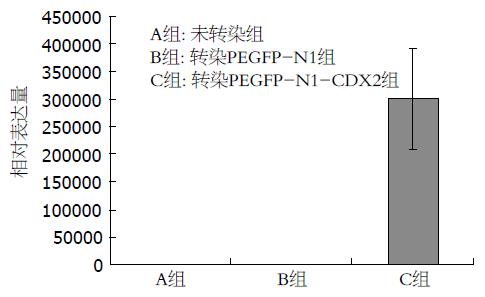
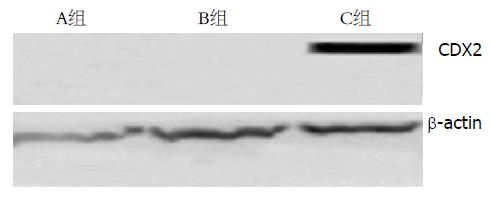
实时荧光定量PCR和Western blot检测结果提示, 转染48 h后转染PEGFP-N1-CDX2组细胞CDX2 mRNA和蛋白含量均高于对照组, 差异均具有统计学意义(P<0.05)(图1, 2).
2.2.1 细胞增殖实验: 瞬时转染48、72 h后, 转染PEGFP-N1-CDX2组的细胞与未转染组、转染PEGFP-N1组相比出现明显生长抑制(P<0.05, 表1).
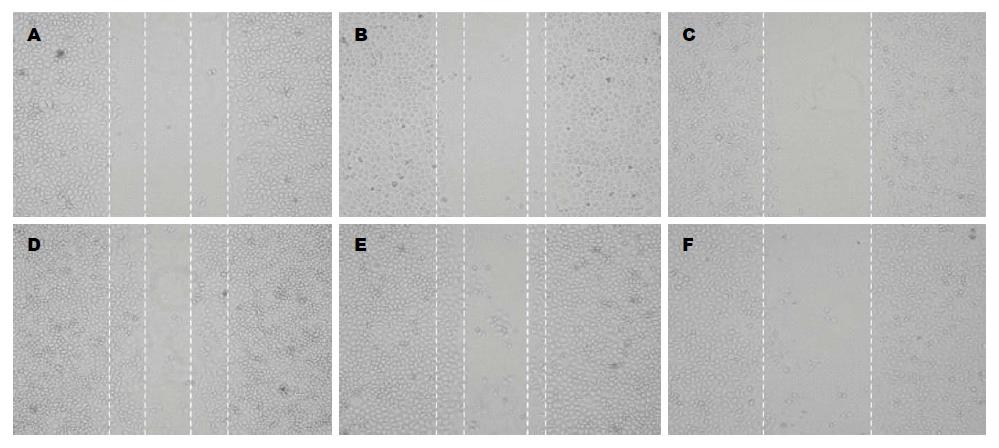
2.2.2 细胞迁移能力: 瞬时转染48 h后的各组细胞, 比较划痕后24 h划痕宽度, 结果提示, 转染PEGFP-N1-CDX2组(划痕修复率为3.64%±2.3%)的细胞与未转染组(划痕修复率为57.60%±8.8%)、转染PEGFP-N1组(划痕修复率为51.52%±3.09%)相比迁移能力受到抑制(P<0.05, 图3).
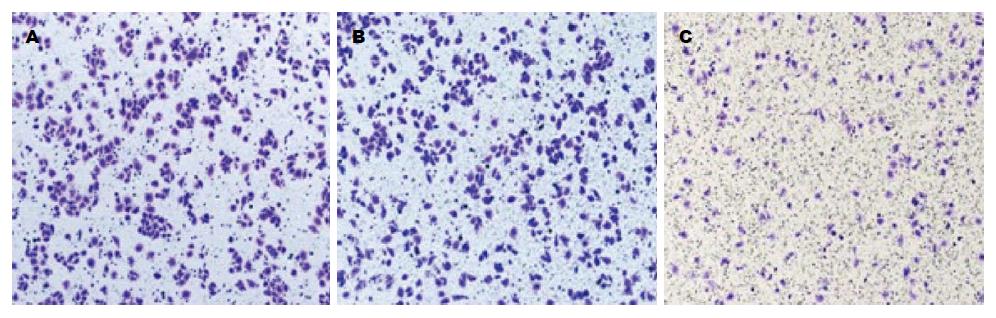
2.2.3 细胞侵袭能力: 瞬时转染48 h后的各组细胞, 通过计数穿过Transwell小孔细胞数目比较侵袭运动能力, 转染PEGFP-N1-CDX2组(22.33±4.77)的细胞与未转染组(47.93±6.29)、转染PEGFP-N1组(45.33±4.59)相比侵袭能力受到抑制(P<0.05)(图4).
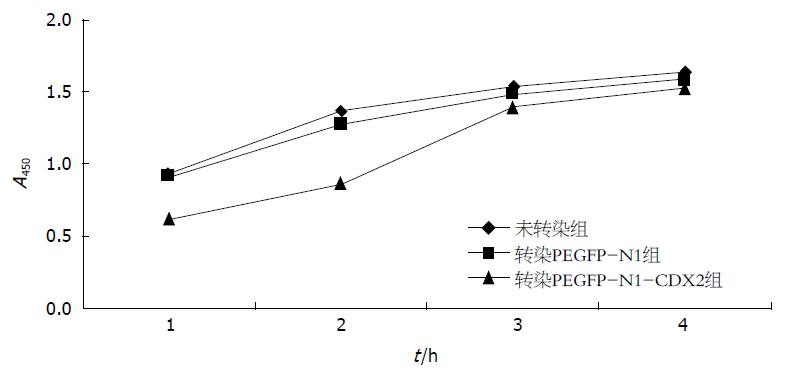
2.2.4 细胞黏附能力: 瞬时转染48 h后的各组细胞, 根据CCK8法检测的A值可见在覆盖Matrigel胶的96孔板培养1、2 h时, 转染PEGFP-N1-CDX2组(0.621±0.151和0.874±0.127)的细胞黏附能力低于未转染组(0.932±0.121和1.369±0.120)和转染PEGFP-N1组(0.915±0.119和1.277±0.172)(P<0.05, 图5).
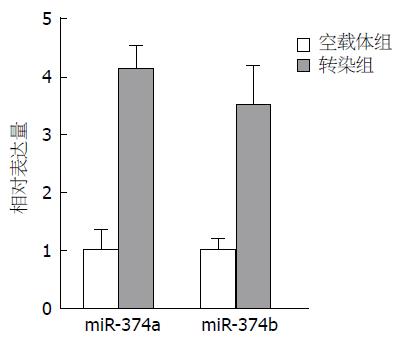
与空载体组细胞比较, 转染组细胞有59种miRNA的表达出现显著改变, 其中25种表达上调2倍以上, 包括miR-150, miR-3170, miR-374a, miR-4292, miR-BHRF1-1, miR-625, miR-4285, miR-138-1, miR-19a, miR-32, miR-498, miR-877, miRPlus-A1086, miR-361-3p, miR-K12-5, miRPlus-I874, miR-29c, miR-1915, miR-525-5p, miR-20a, miR-30bm, miR-374b, miR-106a, miR-17, miR-3126-3p; 34种表达下调2倍以上, 包括let-7b, miR-3178, miR-423-5p, miR-125b, miR-21, miR-361-5p, miR-320c, miR-181a, miR-130a, miR-320b, let-7g, miR-196a, miR-92a, miR-BART19-3p, miR-4301, miR-125a-5p, miR-513a-5p, miRPlus-A1087, miR-33b, miR-1973, miR-205, miR-299-3p, miRPlus-A1072, miR-186, miR-25, miR-106b, miR-K12-6-3p, miR-320a, let-7i, miRPlus-G1246-3p, miR-BHRF1-2, miR-100, miRPlus-A1015, miR-378. 随机选取hsa-miR-374a和hsa-miR-374b进行实时定量PCR验证, 以U6为内参, 结果与芯片检测结果一致(图6).
大多数miRNA基因均具有多个可能的靶位点, 其功能涉及细胞增殖、细胞周期调控、肿瘤浸润转移、肿瘤免疫、细胞信号转导、癌基因、抑癌基因等生物特性密切相关的基因, 参与肿瘤发生、发展、转移的各个方面(表2).
| 名称 | CDX2/空载 | 靶基因 | 功能 |
| hsa-miR-32 | 14.42 | Bim, BTG2等 | 与人类髓系白血病和前列腺癌发生和预后有关 |
| hsa-miR-374a | 9.95 | PBEF, CDH17, K-ras等 | 与胃癌的转移及预后有关 |
| hsa-miR-374b | 2.32 | CDH17等 | 与胃癌的转移及预后有关 |
| hsa-miR-150 | 2.62 | c-Myb, NOTCH3, EGR2, P2X7等 | 通过靶向c-Myb抑制肝癌干细胞; 靶向NOTCH3抑制T细胞的分化和生存; 靶向抑癌基因EGR2促进胃癌细胞增生和生长; 靶向影响凋亡调控细胞生长的P2X7等 |
| hsa-miR-106a | 3.24 | IL-10等 | 星形细胞瘤预后指标; 调节IL-10表达 |
| hsa-miR-320c | 0.44 | MUC2等 | 与肿瘤侵袭转移有关 |
| hsa-miR-320b | 0.30 | MUC2, TFF3等 | 与肿瘤侵袭转移有关 |
| hsa-miR-196a | 0.44 | CDH17等 | 与胃癌的发生、发展、转移及预后有关 |
| hsa-miR-92a | 0.43 | GATA-2等 | GATA-2可能是白血病细胞的增殖过程中所需要的转录因子之一 |
| hsa-miR-320a | 0.42 | BMP2, Osteocalcin, Runx2, MUC2, TFF3等 | 与胃癌的发生、发展、转移及预后有关 |
| hsa-let-7i | 0.46 | TRIM41, SOX13, SLC25A4, SEMA4F, RPUSD2, BTBD3, PLEKHG6, CCND2等 | 与肠癌转移及预后有关 |
近年来, 大量研究已证实胃癌的发生、发展和预后是多基因改变的结果, 研究胃癌相关基因及其蛋白分子不仅对于理解胃癌的发病机制十分重要, 而且对于发现在诊断和评价预后中有临床意义的分子标志物也同样重要.
CDX2基因及相关蛋白最早由Mlodzik于果蝇中分离成功, 与parahox家族呈高度同源性[10,11]. 研究表明, 在人体正常上皮细胞中, CDX2可表达于内胚层来源的肠道上皮及胰腺导管和腺泡上皮, 但在食管和正常胃黏膜上皮中不表达[12]. Mallo等[11]研究发现CDX2 mRNA在85%的结直肠癌中表现出下调, 从而推测CDX2确为一种肿瘤抑制基因. 尽管确切机制尚不清楚, 但CDX2与其他肿瘤抑制因子如APC和E-Cadherin及细胞凋亡蛋白Bcl-2的相互作用进一步支持了这一观点[12-16]. Song等[17]认为CDX2基因突变将使得肠化生中的CDX2表达消失从而促使了肠型胃癌的发生. 由此可见, CDX2基因在胃癌的发生发展中起着重要的作用.
基因转染是指采用一定的方法和途径将DNA、RNA等外源性分子导入特定的细胞, 并表达目的基因, 产生特定功能的蛋白质的过程[18]. 一些传统的转染技术, 如DEAE右旋糖苷法, 磷酸钙法、电穿孔法、脂质体法各有利弊, 阳离子聚合物转染试剂可以用于多种细胞系的转染, 对细胞毒性小, 转染效率高, 操作简单, 转染时不需要除血清及抗生素, 重复性好, 转染安全可靠, 日益受到人们的重视, 本实验我们选用Fermentas公司研发的阳离子聚合物TurboFectTM in vitro Transfection Reagent转染试剂来介导基因转染. 真核表达载体PEGFP-N1是较常用的真核细胞表达外源蛋白的质粒, 他不仅具有多克隆位点, 便于目的基因插入, 而且能在增殖的细胞中稳定表达[19-23]. 本研究通过实时荧光定量PCR法筛选出低表达CDX2的胃癌细胞SGC-7901, 将成功构建的PEGFP-N1-CDX2真核表达载体成功转染胃癌细胞SGC-7901, 显著上调了CDX2 mRNA和蛋白的表达. 实验在此基础上设未转染组、转染PEGFP-N1组、转染PEGFP-N1-CDX2组. 分别观察研究转染前后肿瘤细胞增殖能力、细胞迁移、黏附能力, 以探讨CDX2重组载体对胃癌细胞SGC-7901生物学行为的影响, 从而了解CDX2基因在胃癌中发生发展中的作用.
无限增殖是肿瘤细胞的基本特征, 也是肿瘤转移、侵袭的基础和前提, 随着细胞的增殖导致肿瘤组织内部压力增高, 这种扩张性的压力有利于癌细胞向压力低的方向侵袭和转移[24,25]. 通过实验我们发现: 在转染48、72 h后, 转染PEGFP-N1-CDX2组细胞增殖抑制率明显高于未转染组、转染PEGFP-N1组(P<0.05), 未转染组和转染PEGFP-N1组细胞增殖抑制率相比无显著性差异(P>0.05). 推测CDX2具有抑制胃癌细胞SGC-7901增殖的作用. 运动性是评价肿瘤细胞体外转移潜能之一[26,27]. 本研究应用体外划痕试验测定3组细胞迁移能力, 结果显示转染PEGFP-N1-CDX2组细胞迁移能力与未转染组及转染PEGFP-N1组细胞相比受到明显抑制(P<0.05). Transwell小室培养是目前常用的体外研究肿瘤细胞迁移行为的经典而快速的方法[28], 通过计数穿过Transwell小孔细胞数目比较迁移运动能力, 结果显示转染PEGFP-N1-CDX2组细胞穿过小孔数目低于未转染组及转染PEGFP-N1组(P<0.05). 肿瘤细胞黏附其他肿瘤细胞、宿主细胞或细胞外基质成分的能力, 在原发肿瘤的侵袭、穿入和穿出血管以及在靶器官的侵袭中均有重要意义. 将Matrigel铺在96孔板上, 通过CCK8法检测细胞与细胞外基质的黏附能力. 实验结果证实: 将瞬时转染48 h后的各组细胞重悬于覆盖Matrigel胶的96孔板培养1、2、3、4 h, 在培养1、2 h时, CCK8检测显示转染PEGFP-N1-CDX2组的细胞黏附能力低于未转染组、转染PEGFP-N1(P<0.05), 推测CDX2抑制胃癌细胞SGC-7901与细胞外基质的黏附, 而在培养3、4 h时3组细胞黏附能力无明显区别(P>0.05), 可能是因为此时3组细胞基本全部黏附在基底膜上的原因.
到目前为止, 已报道有几千种miRNA存在于动物、植物、真菌等多细胞真核生物中, 进化上高度保守. 已有许多研究表明不同的miRNA在不同肿瘤中增高或者降低, 同一种肿瘤的不同发展阶段, miRNA的表达水平也不相同, 证明miRNA不仅可以作为肿瘤抑制因子, 也可以作为肿瘤发生因子[29,30]. miRNA的表达失控是各种疾病尤其是肿瘤的重要特征. 本实验我们通过miRNA芯片技术筛选出上调CDX2基因表达时胃癌细胞SGC-7901差异表达的miRNA, 结果发现转染组(转染PEGFP-N1-CDX2组)有59种miRNA的表达出现显著改变, 其中25种表达上调2倍以上, 34种表达下调2倍以上(P<0.05). miRNA芯片尽管融合了各种改良技术, 仍难以避免假阳性, 因此检测结果仍需要通过实时定量PCR进行验证[31]. 我们从筛选出的差异表达的miRNA中随机选取hsa-miR-374a和hsa-miR-374b, 以U6为内参, 进行实时定量PCR验证, 结果显示与芯片结果变化趋势一致, 从而认为本次芯片实验结果是可信的. miRNA检测技术发现差异表达的miRNA分子后, 可以通过靶基因预测的生物信息学方法对其靶基因进行预测, 进行电脑上的功能分析. 我们将得到的差异表达的miRNA应用Miranda、TargetScan、Mirtarget2软件分别预测其靶基因, 取同时满足3个软件预测到的基因作为靶基因. 对各个miRNA的靶基因分别进行Gene Ontology(GO)和KEGG pathway分析了解其功能. 大多数miRNA均具有多个可能的靶位点, 其功能涉及细胞增殖、细胞周期调控、肿瘤浸润转移、肿瘤免疫、细胞信号转导、癌基因、抑癌基因等生物特性密切相关的基因, 为我们进一步研究其与胃癌的发生、发展的关系奠定基础.
胃癌是常见的消化系恶性肿瘤之一, 严重危害着人类的生命健康. 胃癌的发生发展是一个多阶段、多步骤、涉及多基因的病理过程, 其确切的发病机制至今尚不清楚.
斯坎德尔•白克力, 教授, 新疆医科大学基础医学院生物化学教研室
研究发现尾型同源盒基因2(caudal type homeobox gene 2, CDX2)能够抑制肠化生组织向胃癌的转变, 推测他是抑制胃癌侵袭和转移的重要因素. 近年来发现miRNA与肿瘤的发病密切相关, 参与多种肿瘤细胞增殖、凋亡、侵袭或血管形成等过程.
miRNA不仅可以作为肿瘤抑制因子, 也可以作为肿瘤发生因子, miRNA的表达失控是各种疾病尤其是肿瘤的重要特征.
CDX2是一种特异性核转录因子, 文献报道CDX2的异位表达是胃黏膜癌变过程中的早期事件,但上调CDX2基因表达对胃癌生物学行为的影响及其与miRNA的关系目前尚不清楚.
上调CDX2基因表达后人胃癌细胞SGC-7901有59种差异表达的miRNA, 其中25种miRNA上调和34种miRNA下调, 这些靶基因参与了肿瘤的发生、发展、转移及预后, 为我们进一步研究其与胃癌的发生、发展的关系奠定基础.
本文研究内容充实, 应用方法较多, 得出的结果在胃癌研究中具有重要意义.
编辑: 田滢 电编: 鲁亚静
| 1. | Hartgrink HH, Jansen EP, van Grieken NC, van de Velde CJ. Gastric cancer. Lancet. 2009;374:477-490. [PubMed] [DOI] |
| 2. | Yang L. Incidence and mortality of gastric cancer in China. World J Gastroenterol. 2006;12:17-20. [PubMed] |
| 3. | 毛 振彪, 许 钟, 张 健锋, 朱 慧君, 章 建国, 潘 正平. 鸟苷酸环化酶C和尾型同源盒转录因子2在胃癌及癌前病变组织中的表达及意义. 中华消化杂志. 2008;28; 673-677. |
| 4. | Saad RS, Ghorab Z, Khalifa MA, Xu M. CDX2 as a marker for intestinal differentiation: Its utility and limitations. World J Gastrointest Surg. 2011;3:159-166. [PubMed] [DOI] |
| 7. | Mutoh H, Sakurai S, Satoh K, Tamada K, Kita H, Osawa H, Tomiyama T, Sato Y, Yamamoto H, Isoda N. Development of gastric carcinoma from intestinal metaplasia in Cdx2-transgenic mice. Cancer Res. 2004;64:7740-7747. [PubMed] [DOI] |
| 8. | He L, Thomson JM, Hemann MT, Hernando-Monge E, Mu D, Goodson S, Powers S, Cordon-Cardo C, Lowe SW, Hannon GJ. A microRNA polycistron as a potential human oncogene. Nature. 2005;435:828-833. [PubMed] [DOI] |
| 9. | Zhang B, Pan X, Cobb GP, Anderson TA. microRNAs as oncogenes and tumor suppressors. Dev Biol. 2007;302:1-12. [PubMed] [DOI] |
| 10. | Beck F, Chawengsaksophak K, Luckett J, Giblett S, Tucci J, Brown J, Poulsom R, Jeffery R, Wright NA. A study of regional gut endoderm potency by analysis of Cdx2 null mutant chimaeric mice. Dev Biol. 2003;255:399-406. [PubMed] [DOI] |
| 11. | Mallo GV, Rechreche H, Frigerio JM, Rocha D, Zweibaum A, Lacasa M, Jordan BR, Dusetti NJ, Dagorn JC, Iovanna JL. Molecular cloning, sequencing and expression of the mRNA encoding human Cdx1 and Cdx2 homeobox. Down-regulation of Cdx1 and Cdx2 mRNA expression during colorectal carcinogenesis. Int J Cancer. 1997;74:35-44. [PubMed] [DOI] |
| 12. | Werling RW, Yaziji H, Bacchi CE, Gown AM. CDX2, a highly sensitive and specific marker of adenocarcinomas of intestinal origin: an immunohistochemical survey of 476 primary and metastatic carcinomas. Am J Surg Pathol. 2003;27:303-310. [PubMed] |
| 13. | Mallo GV, Soubeyran P, Lissitzky JC, André F, Farnarier C, Marvaldi J, Dagorn JC, Iovanna JL. Expression of the Cdx1 and Cdx2 homeotic genes leads to reduced malignancy in colon cancer-derived cells. J Biol Chem. 1998;273:14030-14036. [PubMed] [DOI] |
| 14. | Freund JN, Domon-Dell C, Kedinger M, Duluc I. The Cdx-1 and Cdx-2 homeobox genes in the intestine. Biochem Cell Biol. 1998;76:957-969. [PubMed] |
| 15. | da Costa LT, He TC, Yu J, Sparks AB, Morin PJ, Polyak K, Laken S, Vogelstein B, Kinzler KW. CDX2 is mutated in a colorectal cancer with normal APC/beta-catenin signaling. Oncogene. 1999;18:5010-5014. [PubMed] |
| 16. | Hinoi T, Lucas PC, Kuick R, Hanash S, Cho KR, Fearon ER. CDX2 regulates liver intestine-cadherin expression in normal and malignant colon epithelium and intestinal metaplasia. Gastroenterology. 2002;123:1565-1577. [PubMed] |
| 17. | Song JH, Kim CJ, Cho YG, Chae JS, Cao Z, Nam SW, Lee JY, Park WS. Genetic alterations of the Cdx2 gene in gastric cancer. APMIS. 2008;116:74-80. [PubMed] [DOI] |
| 18. | Yoshida D, Watanabe K, Noha M, Takahashi H, Teramoto A, Sugisaki Y. Anti-invasive effect of an anti-matrix metalloproteinase agent in a murine brain slice model using the serial monitoring of green fluorescent protein-labeled glioma cells. Neurosurgery. 2003;52:187-196; discussion 196-197. [PubMed] |
| 19. | Cormack BP, Valdivia RH, Falkow S. FACS-optimized mutants of the green fluorescent protein (GFP). Gene. 1996;173:33-38. [PubMed] [DOI] |
| 20. | Tao W, Evans BG, Yao J, Cooper S, Cornetta K, Ballas CB, Hangoc G, Broxmeyer HE. Enhanced green fluorescent protein is a nearly ideal long-term expression tracer for hematopoietic stem cells, whereas DsRed-express fluorescent protein is not. Stem Cells. 2007;25:670-678. [PubMed] [DOI] |
| 21. | Ueta Y, Fujihara H, Serino R, Dayanithi G, Ozawa H, Matsuda K, Kawata M, Yamada J, Ueno S, Fukuda A. Transgenic expression of enhanced green fluorescent protein enables direct visualization for physiological studies of vasopressin neurons and isolated nerve terminals of the rat. Endocrinology. 2005;146:406-413. [PubMed] [DOI] |
| 22. | Vitriol EA, Uetrecht AC, Shen F, Jacobson K, Bear JE. Enhanced EGFP-chromophore-assisted laser inactivation using deficient cells rescued with functional EGFP-fusion proteins. Proc Natl Acad Sci USA. 2007;104:6702-6707. [PubMed] [DOI] |
| 23. | Tamberg N, Lulla V, Fragkoudis R, Lulla A, Fazakerley JK, Merits A. Insertion of EGFP into the replicase gene of Semliki Forest virus results in a novel, genetically stable marker virus. J Gen Virol. 2007;88:1225-1230. [PubMed] [DOI] |
| 25. | Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell. 2011;144:646-674. [PubMed] [DOI] |
| 27. | Eccles SA, Welch DR. Metastasis: recent discoveries and novel treatment strategies. Lancet. 2007;369:1742-1757. [PubMed] [DOI] |
| 28. | Ponath PD, Wang J, Heath H. Transwell chemotaxis. Methods Mol Biol. 2000;138:113-120. [PubMed] [DOI] |
| 29. | Esquela-Kerscher A, Slack FJ. Oncomirs - microRNAs with a role in cancer. Nat Rev Cancer. 2006;6:259-269. [PubMed] [DOI] |
| 30. | Hwang HW, Mendell JT. MicroRNAs in cell proliferation, cell death, and tumorigenesis. Br J Cancer. 2006;94:776-780. [PubMed] [DOI] |